Darstellung der intrazellulären Lokalisierung einer Protein-Protein-Interaktion mit NanoBiT Technologie
Eine Protein-Protein-Interaktion (PPI) ist der direkte physikalische Kontakt zwischen zwei Proteinen. Angesichts der Tatsache, dass Proteine die meisten zellulären Prozesse ausführen und selten allein funktionieren, ist die Untersuchung von Protein-Interaktionen zwischen zwei Paaren oder größeren Gruppen in allen Forschungsbereichen wichtig, insbesondere in der Arzneimittelentwicklung und der Krankheitsforschung. Auch wenn es verschiedene Methoden zur Überwachung von Protein-Protein-Interaktionen ohne die Verwendung von Zellen gibt, spiegeln diese Methoden nicht genau die intrazelluläre Umgebung wider. Zudem erfordern einige PPI-Überwachungsmethoden unter Verwendung von Zellen eine Lyse und spezielle Geräte für die quantitative Erkennung.
Um diese Herausforderungen zu bewältigen, entwickelte Promega die NanoLuc Binary Technology (NanoBiT). NanoBiT ist ein Reportersystem für die strukturelle Komplementierung, das für die intrazelluläre Erkennung von PPIs verwendet werden kann. Es wurde bereits erfolgreich in verschiedenen Forschungsbereichen eingesetzt, z. B. zur Überprüfung von Arzneimitteln, zur Analyse der Signaltransduktion und zur Analyse der Mechanismen von Virusinfektionen.
Das System besteht aus einer Large BiT (LgBiT; 17,6 kDa) und einer Small BiT (SmBiT; 11 Aminosäuren) Untereinheit, die mit den gewünschten Proteinen fusioniert werden. Die Untereinheiten werden in der Zelle exprimiert. Somit ergibt nur die gezielte PPI Untereinheiten, die ein funktionelles Enzym bilden, welches ein helles Lumineszenzsignal erzeugt (Abbildung 1).
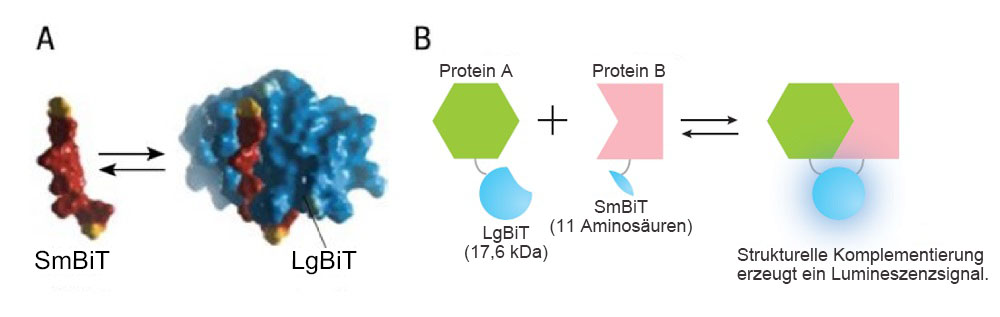
Abbildung 1: Überblick über das NanoBiT System zur Überwachung der Protein-Protein-Interaktion.
Bildquelle: Promega.
Bildgebung der intrazellulären Lokalisierung mit NanoBiT
Um eine zytosolische und nukleäre Expression zu erhalten, haben wir zwei Arten von Vektorpaaren in HeLa-Zellen transfiziert. Das erste Paar war ein nicht zielgerichteter FKBP-SmBiT- und ein FRB-LgBiT-Kontrollvektor. Das zweite Paar war ein NLS1-FKBP-SmBiT- und ein FRB-LgBiT -Kontrollvektor mit Zellkern-Targeting (Abbildung 2). Bekanntlich binden FKBP und FRB unter Rapamycin-Behandlung.
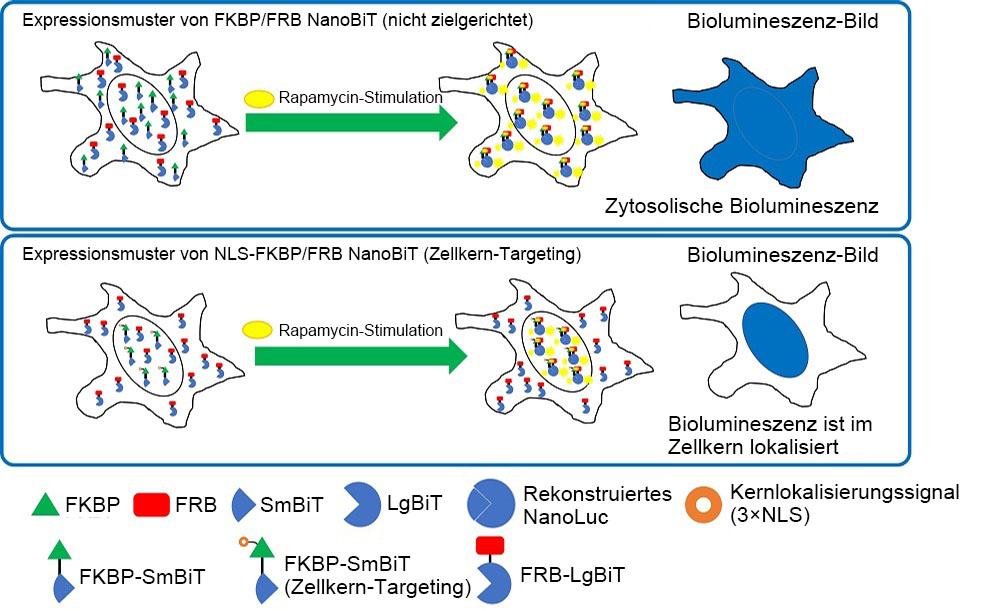
Abbildung 2: Intrazelluläre Lokalisierung von FKBP/FRB NanoBiT und NLS-FKBP/FRB NanoBiT.
Die Biolumineszenz aufgrund der Expression von NanoBiT in HeLa-Zellen wurde dann mit dem IXplore Live for Luminescence Mikroskopsystem abgebildet2. Um die Lokalisierung im Zellkern zu bestätigen, wurden HeLa-Zellen mit Hoechst33342 gegengefärbt und mit demselben Mikroskop unter Fluoreszenz abgebildet (Abbildung 3).
Es war zu beobachten, dass das Biolumineszenzsignal in den HeLa-Zellen, die das nicht zielgerichtete FKBP/FRB NanoBiT erhielten, im gesamten Zytosol verteilt war. Die Biolumineszenz des NLS-FKBP/FRB NanoBiT wurde jedoch aufgrund der Überlagerung mit der Hoechst33342-Fluoreszenzgegenfärbung im Zellkern lokalisiert und bestätigt.
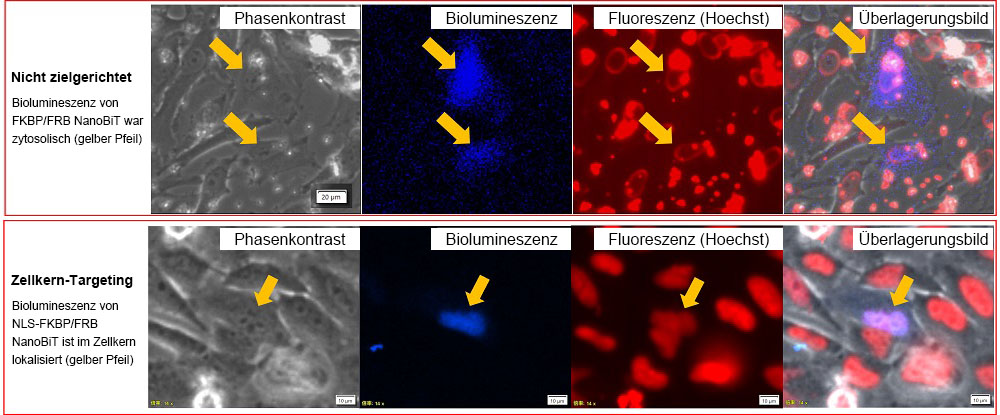
Abbildung 3: Intrazelluläre Lokalisierung von FKBP/FRB NanoBiT und NLS-FKBP/FRB NanoBiT.
Versuchsbedingungen:
- Mikroskop: IXplore Live for Luminescence Mikroskopsystem 2
- Kamera: imageEM EM-CCD-Kamera (Hamamatsu Photonics)
- Objektiv: LUCPLFLN60XPH
- Linse: 0,35X
- EM-Verstärkung: 1200
- Konzentration von Furimazin: Verdünnung von 1/200
- Endkonzentration von Rapamycin: 30 nM
- Belichtungszeit: Phasenkontrast 50 ms, Biolumineszenz 3 Min., Fluoreszenz 100 ms
Abbildung 4 zeigt die Veränderung der Biolumineszenz von NLS-FKBP/FRB NanoBiT vor und nach der Rapamycin-Stimulation. Die Intensität der Biolumineszenz im Zellkern nahm nach der Stimulation mit Rapamycin zu. Diese Ergebnisse zeigen, dass FKBP und FRB unter Rapamycin-Behandlung im Zellkern binden und die Interaktion die Biolumineszenz von NanoLuc induzierte. Die mikroskopische Bildgebung ermöglichte es, die durch Rapamycin-Stimulation induzierte Interaktion von FKBP und FRB als Veränderungen der zytosolischen und nukleären Biolumineszenzintensität in der Lebendzelle zu beobachten.
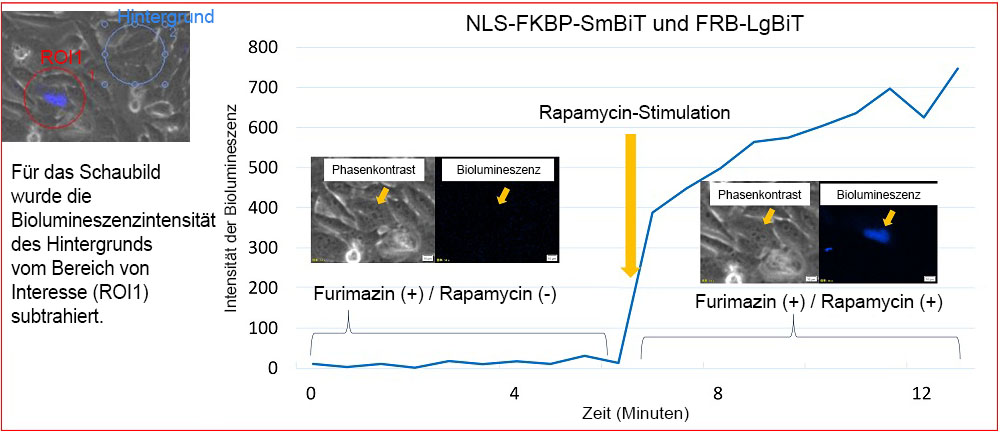
Abbildung 4: Änderung der Biolumineszenzintensität von NLS-FKBP/FRB durch Rapamycin-Stimulation.
Versuchsbedingungen:
- Vektoren: NLS-FKBP-SmBiT/FRB-LgBit Vektoren (je 50 ng)
- Mikroskop: IXplore Live for Luminescence Mikroskopsystem2
- Kamera: imageEM EM-CCD-Kamera (Hamamatsu Photonics)
- EM-Verstärkung: 1200
- Objektiv: UPLFLN40XPH
- Linse: 0,35X
- Belichtungszeit: Phasenkontrast 50 ms, Biolumineszenz 30 s, Fluoreszenz 100 ms
- Zeitraffer-Intervall: 40 Sekunden
PPI-Erkennung mit Luminometer und Lumineszenzmikroskopie
NanoBiT undHiBiT3 Technologien ermöglichen es, Hochdurchsatz-PPIs mit einem Luminometer zu erkennen, während die mikroskopische Bildgebung dieser Technologien die intrazelluläre Lokalisierung dieser PPIs darstellt.
Die Kombination eines Luminometers mit einem speziellen Mikroskop für Lumineszenz zur PPI-Erkennung bietet mehrere Vorteile. Bei der Beobachtung vorhandener PPIs oder Veränderungen ihrer intrazellulären Lokalisation in einem Bereich kann die Genauigkeit der Lokalisation mittels mikroskopischer Bildgebung bestätigt werden, bevor eine Hochdurchsatzmessung mit einem Luminometer durchgeführt wird. Durch dieses System wird eine zusätzliche Fluoreszenzbildgebung zur Bestätigung der intrazellulären Lokalisierung überflüssig. Wir können dieselbe konstruierte Zelllinie mit Expression von NanoBiT oder HiBiT verwenden, um die intrazelluläre Lokalisierung und Hochdurchsatzerkennungen mit einem Luminometer zu überprüfen.
Autor
Taro Hayashi
Wissenschaftler, Biological Engineering, Advanced Optics Biological Engineering, Research and Development, Evident
1. Zellkernlokalisierungssignal (wir haben das 3-fache des nukleären Repeat-Lokalisierungssignals verwendet).
2. Biolumineszenz-Bildgebungssystem basierend auf dem IXplore Live Mikroskop. Dieses System wurde unter Verwendung von Ergebnissen von Prof. Taro Nagai der Universität Osaka, Japan, Tokai Hit., Co, Ltd. in Zusammenarbeit mit Evident entwickelt. Diese Forschungsarbeiten entstanden im Rahmen des Programms der Japan Science and Technology Agency zur Entwicklung fortschrittlicher Mess- und Analysesysteme.
3.HiBiT-Technologie zur Erkennung von Targetproteinen mittels Biolumineszenz unter Verwendung eines 11-Aminosäure-Peptid-Tags (HiBiT) und eines NanoLuc-Luciferase-Fragments mit etwa 18 kDa (LgBiT).
Verwendete Produkte
wurde erfolgreich zu Ihren Lesezeichen hinzugefügt
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
Not Available in Your Country
Sorry, this page is not
available in your country.