Analyse-Arbeitsablauf für 3D-Assays auf lebende/tote Zellen
Zusammenfassung
Die NoviSight 3D-Zellanalyse-Software liefert statistische Daten zu Sphäroiden und anderen 3D-Objekten durch Messung von Parametern wie Volumen, Sphärizität und Zellzahl. In diesem Anwendungsbeispiel stellen wir den NoviSight 3D-Zellanalyse-Arbeitsablauf für einen Assay auf lebende/tote Sphäroide vor.
Einleitung
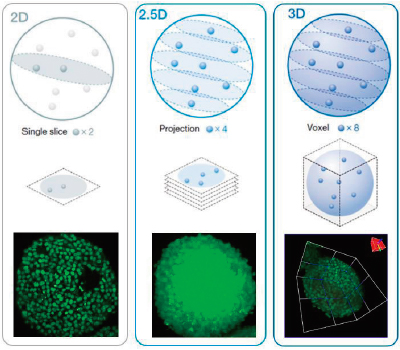
Die 3D-High-Content-Analyse (HCA) unter Verwendung von Sphäroiden oder Organoiden kann schwierig sein. Im Idealfall können 3D-Proben dreidimensional analysiert werden, um Informationen über ihre Morphologie und ihren inneren Zustand zu erhalten. Dank echter 3D-Analyse mit der NoviSight Software kann dieser Prozess durch die Bestimmung des Volumens, der Zellzahl und des Zustands von Zellen in 3D-Proben anhand sequenzieller Z-Stapel-Fluoreszenzbilder vereinfacht werden. Bei der herkömmlichen Analyse dagegen wird ein einzelnes Schnittbild (Abbildung 1, links, 2D-Analyse) oder ein Projektionsbild (Abbildung 1, Mitte, 2,5D-Analyse) verwendet. Bei beiden Techniken gehen viele Daten der 3D-Probe verloren, beispielsweise räumliche, morphologische und Volumeninformationen (Abbildung 1). In diesem Anwendungsbeispiel demonstrieren wir die quantitative Analyse von 3D-Proben mit der NoviSight Software unter Verwendung eines Assays auf lebende/tote Sphäroide. |
Abbildung 1 (von links nach rechts): 2D-, 2,5D- und 3D-Analyse, schematisch dargestellt. |
Methoden
Vorbereitung der Proben
HT-29-Zellen wurden in einer Dichte von 500 Zellen/Well in einer PrimeSurface 96U Platte (Sumitomo Bakelite) acht Tage lang kultiviert, um Sphäroide zu erhalten. Anschließend wurden verschiedene Konzentrationen von Staurosporin (STS) mit NucView 550 (Biotium) zugesetzt. NucView 550 ist eine fluoreszierende Caspase-3-Sonde, die tote Zellkerne anfärbt und rotes Fluoreszenzlicht emittiert. Nach Behandlung mit STS und NucView 550 wurden die HT-29-Sphäroide mit 4 % Paraformaldehyd fixiert und mit 0,5 % Triton-X-100/PBS(-) permeabilisiert. Die Sphäroid-Zellkerne wurden über Nacht bei 4 °C mit Hoechst 33342 angefärbt. Nach der Färbung wurden die Sphäroide mit SCALEVIEW-S4 geklärt.
Bildaufnahme und Analyse
Die Zellen wurden mit dem konfokalen FV3000 Mikroskop und einem semiapochromatischen Objektiv in drei Dimensionen aufgenommen. Die Aufnahmebedingungen sind nachstehend angegeben. Die Z-Schrittgröße betrug 2 μm. Für Hoechst 33342 (blaue Fluoreszenz) wurde ein 405-nm-Laser und für NucView 550 wurde ein 561-nm-Laser (magentafarbene Fluoreszenz) verwendet. Abbildung 2 zeigt mit dem Mikroskop von nicht behandelten und STS-behandelten Sphäroiden aufgenommene volumetrische Bilder. Der Kern eines nicht behandelten, 8 Tage lang kultivierten Sphäroids war tot, wahrscheinlich wegen Nährstoffmangels oder Hypoxie. Staurosporin induzierte generell den Tod von Zellen in den HT-29-Sphäroiden.
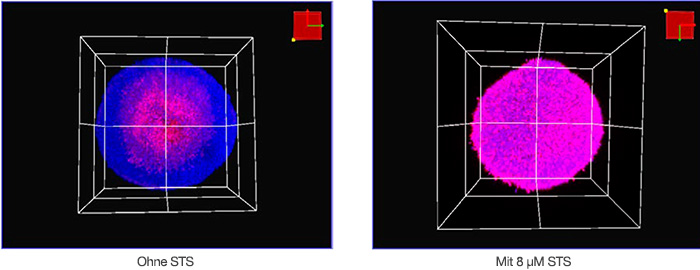
Abbildung 2: Volumetrische Ansicht von HT-29-Sphäroiden mit (rechts) und ohne (links) STS.
Vorteile
- Einfache Durchführung eines 3D-Viabilitätstests auf lebende/tote Sphäroide
- Intuitive Benutzeroberfläche erleichtert die Transformation von Z-Stapel-Bildern in reproduzierbare quantitative Daten
Strategie für die 3D-Analyse auf lebende/tote Sphäroide
Die in Tabelle 1 aufgeführten Schritte sind für die quantitative Analyse lebender/toter Sphäroide mit der NoviSight Software erforderlich. Damit die NoviSight Software die Zellkerne in den Z-Ebenen-Bildern dreidimensional erkennen kann, muss die Schrittgröße des Z-Abschnitts auf 3 μm eingestellt werden – die maximale Schrittgröße für die Erkennung einzelner Zellkerne.
| Schritt | Details |
| Vorbereitung der Probe |
Zellkultur und Fluoreszenzfärbung
Option: Fixierung und Klärung Wenn die Zellzahl analysiert werden soll, ist es empfehlenswert, die Zellkerne anzufärben ↓ |
| Bildgebung |
Aufnahme mehrerer Z-Stapel-Bilder
z. B. Konfokalmikroskopie, Multiphotonen-Mikroskopie ↓ |
| Analyse |
1. Erkennen von Objekten von Interesse, z. B Zellkern, Volumen, Organell
2. Einstellung der Analyse-Parameter 3. Grafische Analyse durch Gating 4. Statistische Auswertung |
Tabelle 1: Arbeitsablauf der 3D-Analyse von der Probenvorbereitung zur Analyse.
Analyseschritte mit der NoviSight Software
Hauptobjekterkennung
Mit dem Assay auf lebende/tote Zellen wurde die Anzahl der lebenden und toten Zellen bestimmt. Die NoviSight Software erkennt die Zellzahl anhand der Färbung der Zellkerne. Zur Erkennung der Zellkerne wurde ein spezielles NoviSight Modul zur Detektion von Zellkernen verwendet: NuclearL. Wenn die richtige Zellgröße, die Hintergrundintensität und das Z/XY-Verhältnis (Durchmesser des Zellkerns entlang der Z-Achse) eingegeben werden, erkennt die NoviSight Software automatisch die Zellkerne des Sphäroids im 3D-Raum (Abbildung 3A).
Einstellung der Analyseparameter
Unter den mit Hoechst 33342 angefärbten Zellkernen waren diejenigen mit hoher NucView 550 Signalintensität tote Zellen. Folglich kann die NoviSight Software lebende/tote Zellen anhand des Ch2(NucView 550)-Intensitätsniveaus der erkannten Zellkern-Objekte klassifizieren (Abbildung 3B). Das bedeutet, dass die NoviSight Software lebende/tote Zellen in Sphäroiden allein mit der Zellkernerkennungs-Einstellung klassifizieren kann.
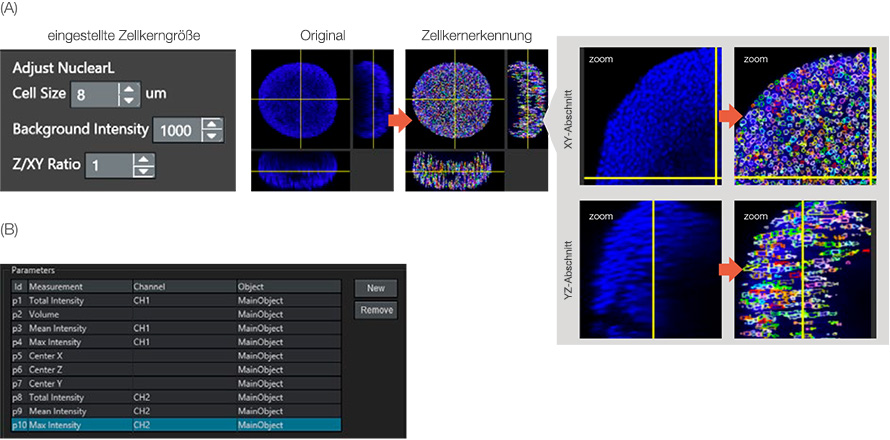
Abbildung 3: Erkennung übergeordneter Objekte. (A) Zellkerne wurden vom NuclearL-Modul erkannt. Jeder erkannte Zellkern wurde farbig eingekreist. (B) Analyseparameter wurden eingestellt. Beispielsweise ist Parameter 10 die Maximalintensität von CH2 (Signal „tote Zelle“) am Hauptobjekt (Zellkern).
Ist die Zellkernerkennung abgeschlossen, können die erkannten Zellkerne als Objektdaten in einem Streudiagramm oder einem Histogramm dargestellt werden. Die X- oder Y-Achse des Streudiagramms kann anhand der Analyseparameter angepasst werden. Wenn diese auf den zentralen X/Y-Wert gesetzt werden, wird der zentrale X/Y-Wert der Hauptobjekte im Diagramm eingezeichnet (Abbildung 4A). Wird ein Punkt im Diagramm angeklickt, kann die NoviSight Software dieselbe Position im Originalbild anzeigen (Abbildung 4B).
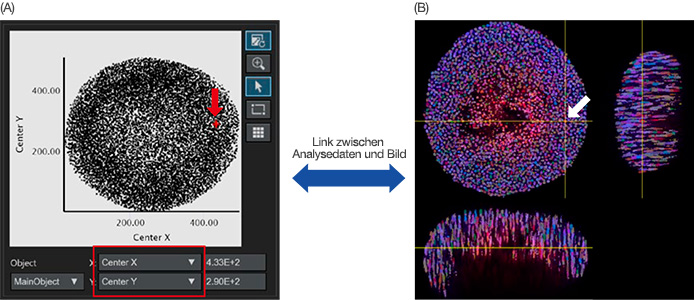
Abbildung 4: Bild zur quantitativen Analyse eines Sphäroids. (A) Wenn die Analyseparameter zentraler X- und Y-Wert (eingestellt in Abbildung 3B) auf der X- und Y-Achse eingestellt wurden, wird jeder erkannte Zellkern im Diagramm eingetragen. Die Analyseparameter auf der X/Y-Achse können bei Bedarf angepasst werden (rotes Rechteck). (A/B) die NoviSight Software kann die Analysedaten sichtbar mit den Originalbildern verknüpfen. Wenn der eingetragene erkannte Zellkern im Diagramm
angeklickt wird (A, roter Pfeil), wird dieselbe Position automatisch im Originalbild angezeigt (weißer Pfeil).
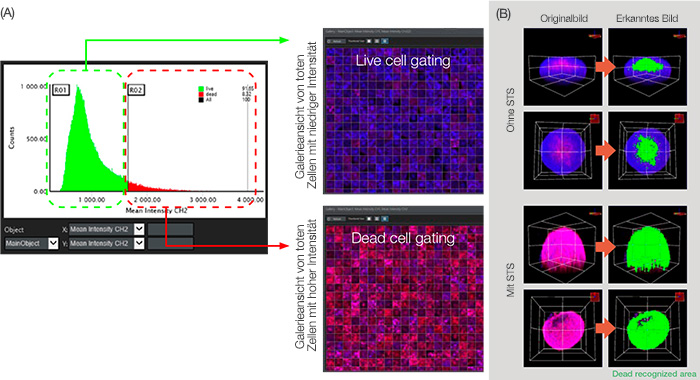
Abbildung 5: Grafische Analyse zur Zellklassifikation.
(A) Wenn der Analyseparameter mittlere Intensität CH2 (in Abbildung 3B) in der X- bzw. Y-Achse eingestellt wurde, zeigt die Grafik die Anzahl der Hauptobjekte (Zellkerne) in der Reihenfolge der mittleren Intensität von CH2 (Signal „tote Zelle“). Durch Gating der Intensität von CH2 können lebende und tote Zellen unterschieden werden. (B) Volumetrische Ansicht des als tot erkannten Bereichs (in grüner Farbe).
Statistische Auswertung
Die NoviSight Software kann mehrere Proben gleichzeitig analysieren. Die quantitativen Daten der Analyse können als Heatmap dargestellt (Abbildung 6A) oder für weitere Analysen als CSV-Datei exportiert werden. Die Daten können auch für jedes Gating ausgegeben werden. Hier zeigen die Daten, dass STS die Apoptose von HT-29-Zellen dosisabhängig induzierte, wenn die HT-29-Sphäroide mit verschiedenen STS-Konzentrationen behandelt wurden (Abbildung 6B).
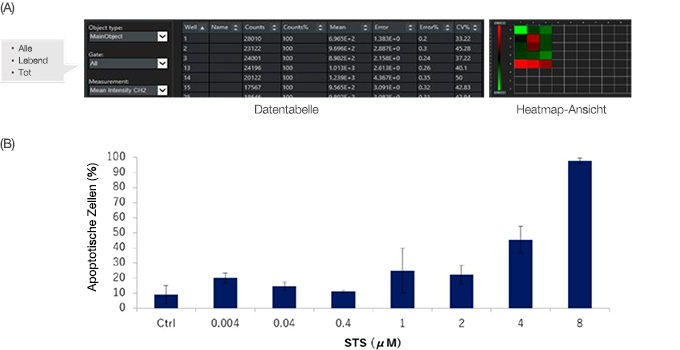
Abbildung 6: Quantifizierungsergebnisse der Analyse auf lebende/tote Zellen in STS-behandelten oder unbehandelten HT-29-Sphäroiden. (A) Die NoviSight Software gibt die Quantifizierungsdaten für die einzelnen Gates aus.
Die Daten können auch in einer Heatmap dargestellt werden. (B) STS erhöhte den HT-29-Zelltod dosisabhängig.
Verknüpfung von Daten und Bildern
Die Verknüpfung von Daten und Bildern ist ein wichtiger Vorteil der High-Content-Analyse (HCA). Anders als bei einem Plattenlesegerät kann bei der HCA auf das Originalbild referenziert werden, um die Morphologie, Position und Intensität der Zellen visuell zu bestätigen. Die NoviSight Software nutzt diesen Vorteil in vollem Umfang. So kann die Software das zugehörige Bild bei einem Klick auf einen bestimmten Datenpunkt oder ein Objekt anzeigen (Abbildung 7), beispielsweise:
- ein Punkt oder ein Streudiagramm
- ein Objektbild in der Galerieansicht
- ein Well in einer Heatmap
- eine Zahl in einer Tabelle
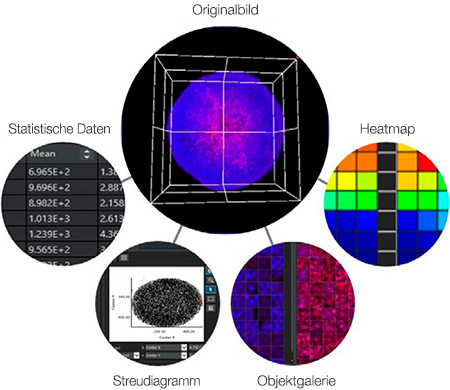
Abbildung 7: Die NoviSight Software zeigt denselben Punkt auf dem zugehörigen Bild an, wenn auf einen Datenpunkt oder ein Objekt geklickt wird.
Schlussfolgerung
Die quantitative 3D-Analyse erscheint möglicherweise kompliziert, doch die NoviSight Software kann den Arbeitsablauf von Anfang bis Ende beschleunigen. Beispielsweise ist die Zellkernerkennung der einzige Parameter, der für die Analyse auf lebende/tote Zellen in Sphäroiden erforderlich ist. Dieser gestraffte Arbeitsablauf kann auch auf relevante 3D-Modelle angewendet werden.
Autor
Mayu Ogawa (Olympus)
Verwendete Produkte
wurde erfolgreich zu Ihren Lesezeichen hinzugefügt
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
Not Available in Your Country
Sorry, this page is not
available in your country.