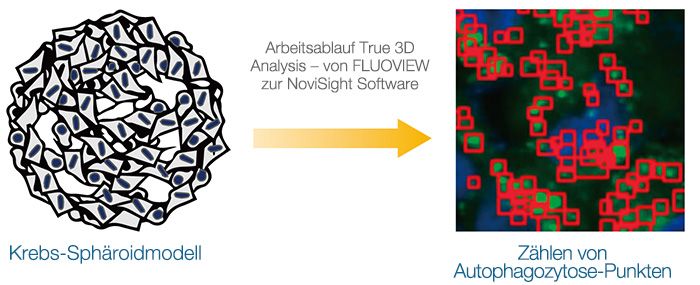
Echte 3D-Analyse eines intrazellulären Autophagozytose-Signalwegs in einem 3D-Sphäroidmodell
Zusammenfassung
Zur Vorhersage der Wirksamkeit von Arzneimitteln oder -toxizität werden 3D-Kulturmodelle eingesetzt. In diesem Anwendungsbeispiel konzentrieren wir uns auf einen intrazellulären Autophagozytose-Signalweg. Zunächst wurde die Eignung der EGFP-LC3-Sonde für die Überwachung des Autophagozytose-Status der Modelle bestätigt. Anschließend wurde der Arbeitsablauf True 3D Analysis unter Verwendung des konfokalen FLUOVIEW FV3000 Mikroskops und der NoviSight Software eingesetzt, um die Wirkung eines Arzneimittelkandidaten mit der Bezeichnung Chloroquin quantitativ zu beurteilen. Es zeigte sich, dass dieser analytische Arbeitsablauf eine Alternative zur Profilierung von Arzneimittelkandidaten durch In-vivo-Studien bietet.
Einleitung
Intrazelluläre molekulare Ereignisse stehen in einem engen Zusammenhang mit einer Vielzahl von Erkrankungen des Menschen. Autophagozytose ist ein intrazellulärer Prozess, bei dem zelluläre Organellen und Proteine abgebaut und verwertet werden. Eine Fehlregulierung der Autophagozytose spielt bei Erkrankungen des Menschen eine Rolle. Um diese Krankheitsprozesse besser zu verstehen und die Wirkung von Arzneimitteln auf sie zu prüfen, ist es wichtig, den Autophagozytose-Status menschlicher Zellen unter pathologischen und therapeutischen Bedingungen zu untersuchen.
Dreidimensionale Kulturmodelle wie Sphäroide oder Organoide bieten eine Mikroumgebung, die der In-vivo-Situation sehr ähnlich ist. Zur Beurteilung der Wirkung eines Arzneimittels auf die Zellen im Sphäroid ist eine quantitative Analyse intrazellulärer Mikroskopbilder von entscheidender Bedeutung. In diesem Anwendungsbeispiel wurden unter Verwendung des konfokalen FV3000 Mikroskops von Olympus und des Arbeitsablaufs True 3D Analysis ein dynamischer intramolekularer Autophagieprozess untersucht und die Wirkung eines Arzneimittelkandidaten beurteilt.
Vorteile
- Quantitative Analyse intramolekularer Ereignisse in 3D-Kulturmodellen
Ergebnisse und Diskussion
Das LC3-Protein ist ein nützlicher Marker bei Autophagieprozessen. So wurde zum Beispiel berichtet, dass während der Akkumulation von Autophagosomen die Anzahl von EGFP-LC3-Fluoreszenzpunkten zunahm. Die Bildung von Autophagosomen wird durch verschiedene Verbindungen gesteuert, die die Autophagozytose-Aktivität oder den Turnover von Autophagosomen regulieren. Einige dieser Verbindungen werden nun in klinischen Studien untersucht. Eine davon, Chloroquin (CQ), ursprünglich ein Malariamittel, wird aktuell in einer klinischen Studie zur Krebsbehandlung geprüft, da es die Fusion von Autophagosomen und Lysosomen inhibiert.
Validierung eines Autophagozytose-Markers mithilfe eines 2D-Kultursystems
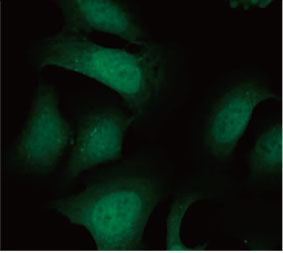
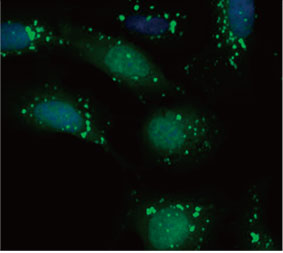
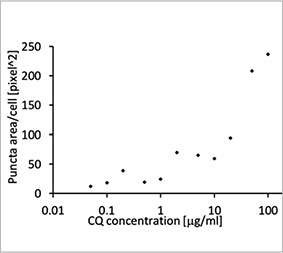
Die Eignung der EGFP-LC3-Sonde zur Ermittlung der inhibitorischen Wirkung von CQ auf die Autophagozytose wurde in einem 2D-Kultursystem validiert. Hierzu wurde eine U2OS-Osteosarkom-Zelllinie hergestellt, die EGFP-LC3 stabil exprimiert. Die Zellen wurden als Monolagen in den Wells von Mikroplatten gezogen. Nach Zugabe von CQ wurden die Zellen fixiert und mit Hoechst 33342 gefärbt. Die Fluoreszenzbilder zeigten, dass die Behandlung mit CQ zur Bildung von EGFP-LC3-Punkten führte (Abbildungen 1 A und B). Die Punktfläche pro Kammer wurde auf Basis der Position des Zellkerns der einzelnen Zellen bestimmt. Die Punktfläche nahm mit steigender CQ-Konzentration zu, was die Eignung der EGFP-LC3-Sonde für die Untersuchung einer Autophagozytose bestätigte.
(A)
| (B)
| (C)
|
Abbildung 1 A und 1 B: (A) EGFP-LC3-exprimierende U2OS-Zellen kultiviert unter normalen Bedingungen. (B) EGFP-LC3-exprimierende U2OS-Zellen nach einstündiger Behandlung mit CQ. Abbildung 1 C: Zunahme der Punktfläche pro Zelle, aufgetragen gegen die ansteigende CQ-Konzentration. | ||
Quantifizierung von LC3-Punkten im Krebs-Sphäroidmodell
Für Untersuchungen zum Autophagozytose-Status von 3D-Kulturmodellen wurde die aus einem Zervixkarzinom etablierte Zelllinie HeLa ausgewählt. HeLa-Zellen eignen sich für ein gerüstfreies 3D-Kultur-Protokoll und bilden Sphäroide einheitlicher Größe. Hierzu wurde eine HeLa-Zelllinie hergestellt, die EGFP-LC3 stabil exprimiert.
Die Zellen wurden in Mikroplatten mit Rundboden und geringer Adhäsion ausgesät. Nach fünf Tagen in Kultur wurden die Sphäroide sieben Stunden lang mit CQ behandelt. Anschließend wurden die Sphäroide fixiert, membranpermeabilisiert, einer Kernfärbung unterzogen und schließlich mit SCALEVIEW-S4 (FUJIFILM WAKO) geklärt.
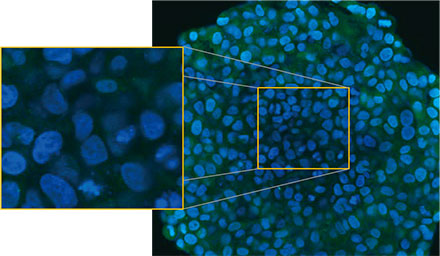
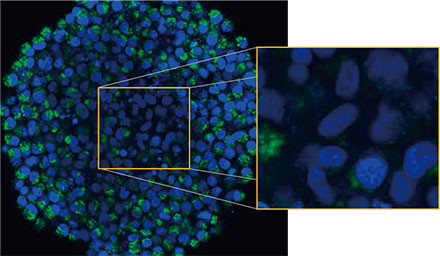
Die mikroskopische Untersuchung der Sphäroide wurde mit dem FLUOVIEW FV3000 Konfokalsystem durchgeführt. Aufgrund seiner hohen numerischen Apertur (NA) und seiner reduzierten sphärischen Aberration wurde ein Silikon-Immersionsobjektiv (UPLSAPO30XSIR) verwendet. Abbildung 2 (A)* zeigt eine einzelne Z-Schichtaufnahme des Äquatorbereichs eines nicht behandelten Sphäroids. Das EGFP-Signal ist in den Zellen gleichmäßig verteilt. Die CQ-Behandlung induzierte dagegen die Bildung von Punkten von EGFP-Signalen in den Zellen (Abbildung 2 (B)*). Die Bildung von Punkten wurde sowohl im peripheren als auch im zentralen Bereich des Sphäroids beobachtet. Die CQ-Behandlung verursachte keine sichtbaren Veränderungen der Sphäroidgröße, was darauf hinweist, dass die intrazelluläre Autophagozytose ein besserer Marker für die Beurteilung der Wirkung von CQ in 3D-Krebs-Sphäroiden sein könnte.
Zur quantitativen Analyse der intrazellulären Ereignisse in 3D-Kulturmodellen wurde der Arbeitsablauf True 3D Analysis von Olympus eingesetzt, der die Aufnahme von Bildern mit dem konfokalen FV3000 Mikroskop und die Analyse mit der NoviSight Software miteinander kombiniert. Dies bietet im Vergleich zu konventionellen analytischen Methoden viele Vorteile. Bei einer konventionellen Analyse würde der integrierte Merkmalswert der erkannten Objekte aus mehreren Z-Schichtaufnahmen verwendet oder das Projektionsbild mehrerer Z-Schichtaufnahmen analysiert. Diese Verfahren sind unzuverlässig, da sie dazu führen können, dass Objekte von Interesse mehrfach gezählt oder, wenn sie mit anderen Objekten überlappen oder von diesen verdeckt sind, übersehen werden. Der Arbeitsablauf mit der True 3D Analysis Software von Olympus schließt dagegen die Aufnahme sequenzieller Z-Schichtbilder mit von der FLUOVIEW Software optimierten Z-Intervallen ein. Die Software rekonstruiert das 3D-Volumen und erkennt die Objekte von Interesse als Cluster von Voxeln. Anschließend wird die NoviSight Software für die statistische Auswertung verwendet. Der Arbeitsablauf für FLUOVIEW mit NoviSight Software ist unkompliziert und bietet eine ideale Plattform für die Untersuchung von 3D-Kulturmodellen.
(A)
| (B)
|
Abbildung 2 A und 2 B: EGFP-LC3-exprimierende HeLa-Zell-Sphäroide (A) unter normalen Bedingungen und (B) nach Behandlung mit CQ. Die vergrößerten Bilder zeigen den in den ursprünglichen Sphäroidbildern durch ein gelbes Rechteck gekennzeichneten Bereich.* | |
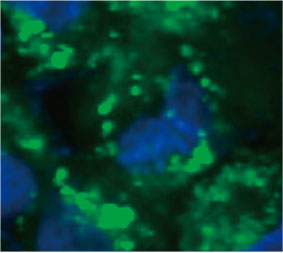
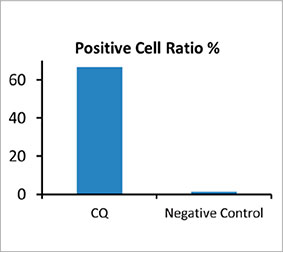
Zur Berechnung der Anzahl Autophagozytose-positiver Zellen in den Sphäroiden wurde von der Software in jeder Zelle der Zellkern identifiziert. Als Nächstes wurde der Bereich um die einzelnen Zellkerne jeweils als Maskenbereich definiert. Schließlich wurden die Punkte von EGFP-Signalen in den Maskenbereichen als Punktobjekte erkannt (Abbildung 2 (C)*). Zellen mit mehr als drei Punkten wurden als Autophagozytose-positive Zellen betrachtet. Das unbehandelte 3D-Sphäroid wies etwa 1,4 % Autophagozytose-positive Zellen auf, das mit CQ behandelte Sphäroid dagegen 66,6 % (Abbildung 2 (D)*). Die quantitative Analyse spricht auch dafür, dass die Bildung der Punkte in den Krebs-Sphäroiden durch CQ ausgelöst wurde.
(C)
|
| (D)
|
Abbildung 2 C links: Ursprüngliches Bild der Zellen im Sphäroid mit CQ. Rechts: Dasselbe Bild mit Objekterkennung von Punkten (rote Rechtecke).* | ||
Schlussfolgerung
Im Rahmen des Arbeitsablaufs für eine echte 3D-Analyse wurden das konfokale FLUOVIEW FV3000 Mikroskop von Olympus und die NoviSight Software verwendet, um die Wirkung von CQ auf die intrazellulären Autophagozytose in 3D-Kulturmodellen zu beurteilen. Diese Studie zeigt, dass dieser Arbeitsablauf genutzt werden kann, um ein Profil der dosisabhängigen Wirksamkeit von Arzneimitteln zu erstellen oder um mehrere Arzneimittelkandidaten objektiv zu screenen. Es stehen noch weitere intrazelluläre molekulare Marker zur Verfügung, die für die Wirksamkeit oder Toxizität von Medikamenten relevant sind, wie beispielsweise FISH-Signale an Chromosomen oder die Translokation fluoreszenzmarkierter Signalmoleküle. Dieser Arbeitsablauf kann dazu beitragen, ein breites Spektrum intrazellulärer Ereignisse in 3D-Kulturmodellen quantitativ zu untersuchen und bietet eine Alternative zur Profilierung von Arzneimittelkandidaten durch In-vivo-Studien.
Literaturnachweis
- Maria C et al., (2019) Targeting autophagy to overcome human disease. International Journal of Molecular Sciences. 20(3): E725. doi: 10.3390/ijms20030725
- Yan C et al., (2013) Therapeutic targeting of autophagy in disease: Biology and Pharmacology. Pharmacological Reviews. 65: 1162–1197. doi: 10.1124/pr.112.007120
- Daniel JK et al., (2008) Guidelines for the use and interpretation of assays for monitoring autophagy in higher eukaryotes. Autophagy. 4(2): 151–175. doi: 10.4161/auto.5338
Autor
Kazuhito Goda
*Obwohl sie zu einer der wichtigsten Zelllinien in der medizinischen Forschung wurde, müssen wir unbedingt anerkennen, dass Henrietta Lacks Beitrag zur Wissenschaft ohne ihre Zustimmung erfolgte. Diese Ungerechtigkeit führte nicht nur zu wichtigen Entdeckungen in der Immunologie, bei Infektionskrankheiten und Krebs, sondern warf auch wichtige Diskussionen über Datenschutz, Ethik und Einwilligung in der Medizin auf.
Um mehr über das Leben von Henrietta Lacks und ihren Beitrag zur modernen Medizin zu erfahren, klicken Sie hier.
http://henriettalacksfoundation.org/
Verwendete Produkte
wurde erfolgreich zu Ihren Lesezeichen hinzugefügt
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
Not Available in Your Country
Sorry, this page is not
available in your country.