Die TruAI Deep-Learning-Technologie ist ein bahnbrechendes Konzept für die Bildanalyse, da sie für Anwendungen einsetzbar ist, die bisher unmöglich waren oder eine große Herausforderung darstellten, z. B. die zuverlässige markerfreie Analyse oder das Tracking lebender Zellen bei sehr geringen Intensitäten über mehrere Tage.
Bei diesen Anwendungen erfolgt die Analyse durch semantische Segmentierung, das heißt, es wird geprüft, ob ein Pixel zum Hintergrund oder zum Vordergrund einer bestimmten Klasse gehört. Für die endgültige Objektsegmentierung (in der Regel Zellkerne oder ganze Zellen) werden nach der semantischen Segmentierung Nachbearbeitungsschritte wie die Wasserscheidentransformation angewandt.
Die TruAI Deep-Learning-Technologie wurde nun durch die Einführung der Instanzsegmentierung nochmals verbessert. Mit dieser Funktion werden die semantische Segmentierung und die anschließende Objektsegmentierung in einem Schritt zusammengefasst. Die Instanzsegmentierung verbessert die Arbeitsabläufe, da komplexe Anwendungen in einem Schritt durchgeführt werden können, beispielsweise die Segmentierung von Zellen in Zellkolonien, die in Nachbearbeitungsschritten aufgeteilt werden.
Bei Verwendung der TruAI Deep-Learning-Technologie können Anwender von Anfang an ihre Daten zum Training ihrer neuronalen Netze (DNN) verwenden und deren Leistung dadurch immer weiter verbessern. Forscher können DNN-Bibliotheken für verschiedene Anwendungen entwickeln und sie sogar mit Kollegen teilen.
Dies bedeutet allerdings, dass der Deep-Learning-Arbeitsablauf immer aus drei Schritten besteht:
- Beschriftung mit eigenen Daten
- Training
- Inferenz
Auch mit unserer benutzerfreundlichen Software kostet der Beschriftungsschritt nach wie vor die meiste Zeit – insbesondere bei anspruchsvollen Anwendungen, bei denen viele Objekte beschriftet werden müssen, um gute Ergebnisse zu erzielen.
Vereinfachte Segmentierung von Zellen und Zellkernen mit vortrainierten Deep-Learning-Modellen
Zur Erleichterung dieser Arbeit enthält die TruAI Deep-Learning-Technologie jetzt einige vortrainierte Modelle für Zellkerne und Zellen, die bei der Analyse von biologischen Bildern die am häufigsten zu segmentierenden Objekte sind. Auf diese Weise lässt sich der Zeitaufwand für das Beschriften und das Training einsparen (Abbildung 1).

Abbildung 1. Mit der TruAI Deep-Learning-Technologie werden bei Verwendung eigener Anwendungsdaten für den Trainings- und Inferenzschritt optimale Ergebnisse erzielt. Um diesen Prozess zu beschleunigen, stehen vortrainierte TruAI Modelle zur Verfügung, die die Analyse in einem Schritt erledigen.
Diese TruAI Modelle für Zellkerne und Zellen sind für die Segmentierung von Instanzen in Fluoreszenzbildern für verschiedene Szenarien vortrainiert, unter anderem für:
- Mikroskopiemodalitäten (konfokale Punkt-Scanning-, konfokale Spinning-Disk-, Weitfeld-Mikroskopie)
- Färbungen (DAPI, Phalloidin, MitoTracker, Lamin usw.)
- Zelllinien (HeLa, U2OS, SK-HEP-1, Pflanzenzellen, Gewebe usw.)
- Kontrast, Signal-Rausch-Verhältnis und Hintergrundstufen
- Pixelauflösung und Vergrößerungen
Es sei darauf hingewiesen, dass es sich bei den vortrainierten Modellen um allgemeine Modelle handelt, mit denen nicht in allen möglichen Situationen perfekte Segmentierungen erzielt werden. Zum besseren Verständnis der Möglichkeiten haben wir eine Liste von Zellsegmentierungsszenarien zusammengestellt.
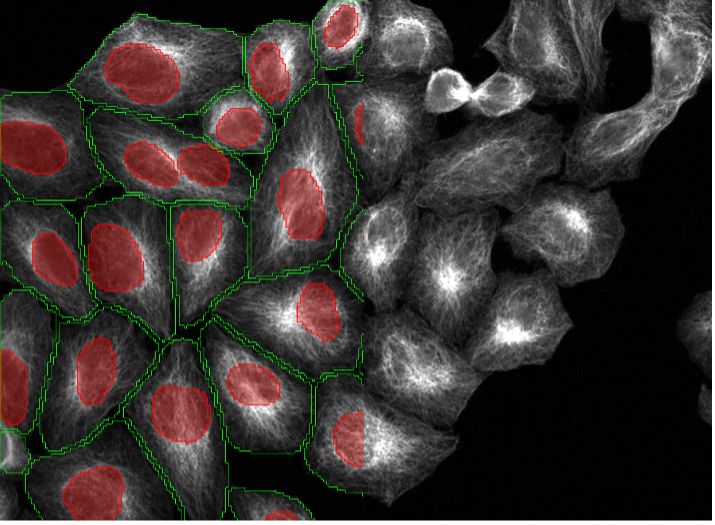
20 Beispiele für die Segmentierung von Zellkernen und Zellen mit vortrainierten Deep-Learning-Modellen
Die folgenden Bildbeispiele (Abbildung 2, a-t) zeigen die Leistung der vortrainierten TruAI Modelle für Zellkerne (rot) und Zellen (grün) in einer Vielzahl von Segmentierungsszenarien. Verwendet wurden unterschiedliche Zelllinien, Markerfärbungen, Vergrößerungen, Auflösungen, Kontraste, Signal-Rausch-Verhältnisse und inhomogene Hintergründe. Es ist zu beachten, dass beide Modelle auf dasselbe Bild angewendet werden können (Abbildung 2, p-t). Die Modelle können sogar auf unspezifische Zellkerne (Abbildung 2, q-t) oder Zellfärbungen (Abbildung 2p, s) angewendet werden.
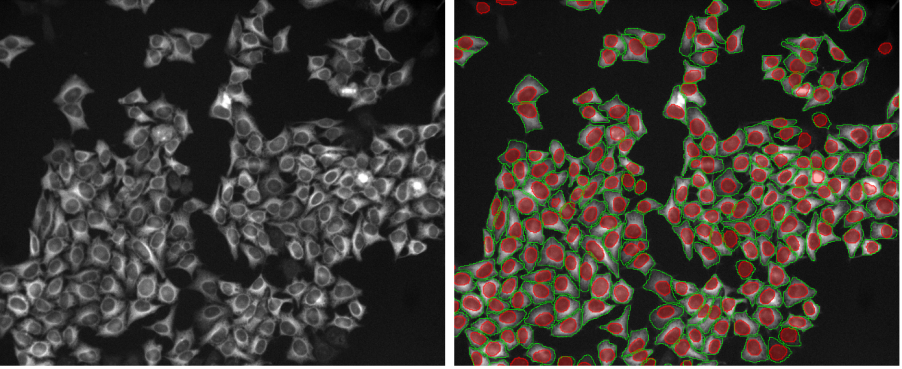
1. U2OS-Zellen, Zellkernfärbung, Weitfeld-Mikroskopie 20X, mittlere Konfluenz mit Zellen unterschiedlicher Größe und Morphologie
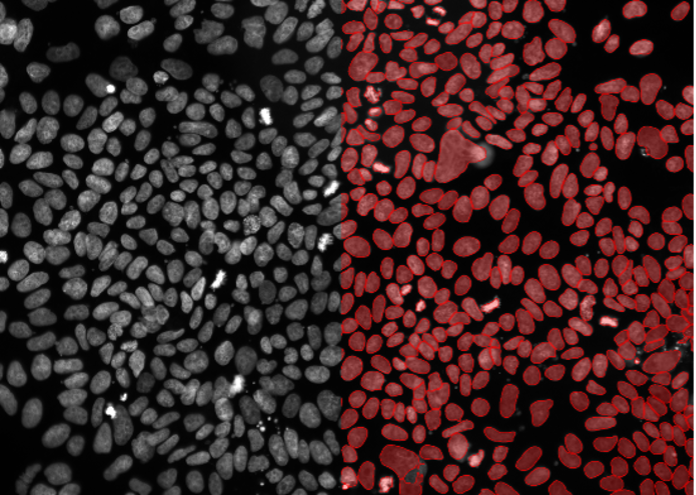
Abbildung 2a
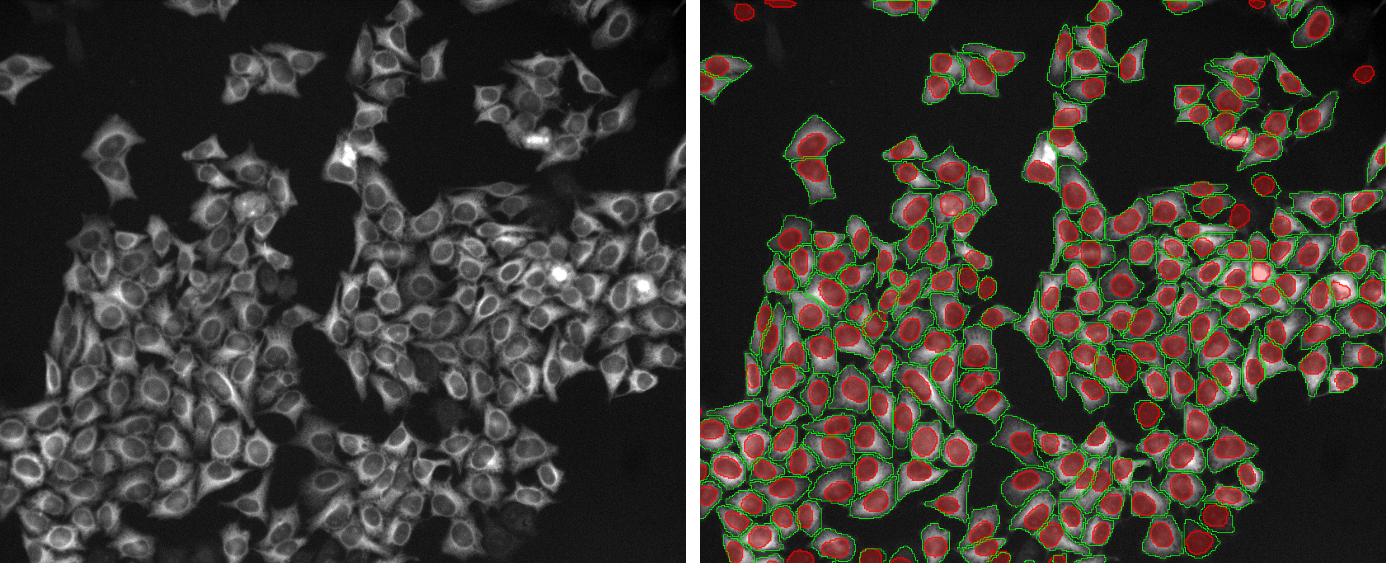
2. HeLa-Zellen, Zellkernfärbung, Weitfeld-Mikroskopie 10X, inhomogener Hintergrund
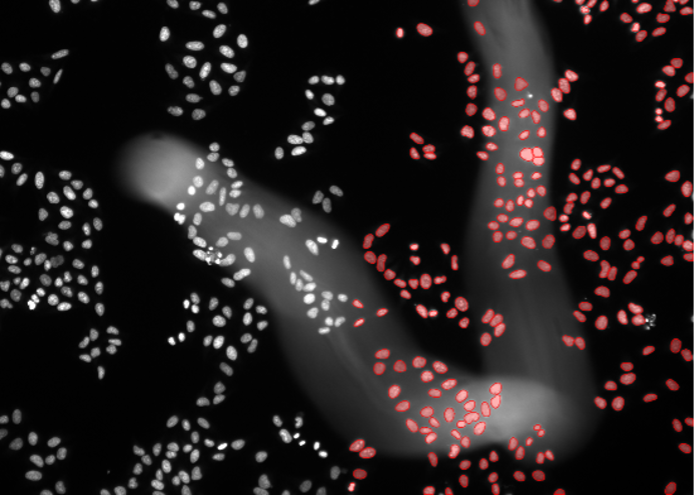
Abbildung 2b
3. Pluripotente Stammzellen, Zellkernmarkierung, Weitfeld-Mikroskopie 20X, sehr hohe Konfluenz

Abbildung 2c
4. Nierenorganoide, Zellkernfärbung, konfokale Spinning-Disk-Mikroskopie 30X, hohe Eindringtiefe
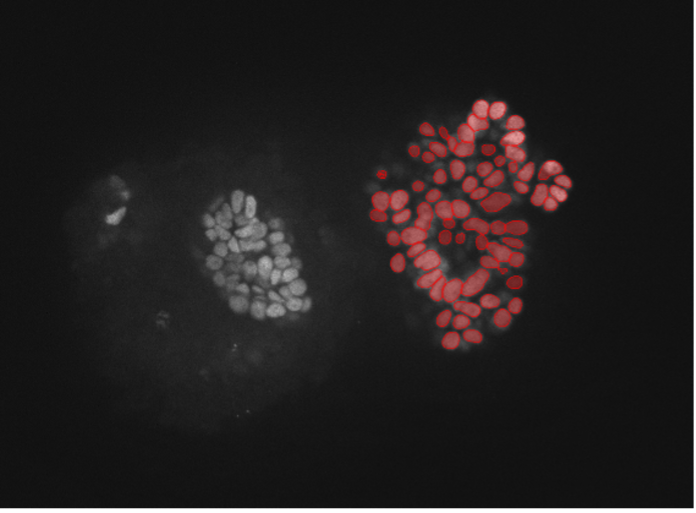
Abbildung 2d
5. HeLa-Zellen, Zellkernfärbung, konfokale Spinning-Disk-Mikroskopie 40X, hoher Kontrast und hohe Auflösung

Abbildung 2e
6. Humane A549Karzinomzellen, Zellkernmarkierung, konfokale Spinning-Disk-Mikroskopie 40X, niedriges Signal-Rausch-Verhältnis
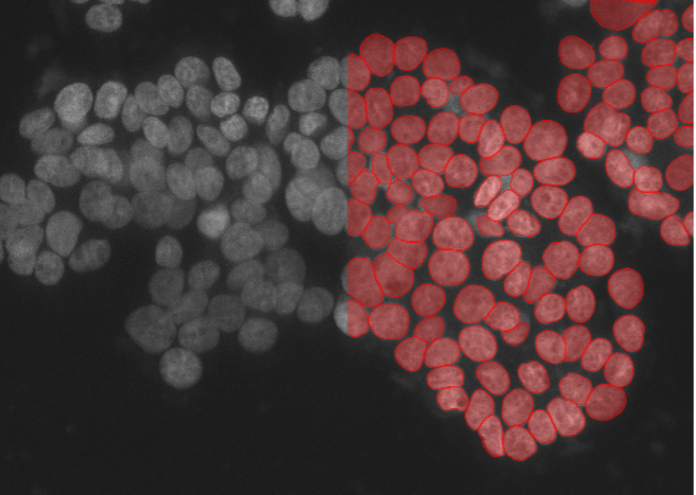
Abbildung 2f
7. Humane Gewebetumorzellen, Zellkernfärbung, Weitfeld-Mikroskopie 20X

Abbildung 2g
8. Nierengewebezellen der Maus, Zellkernfärbung, Weitfeld-Mikroskopie 20X, dickes Präparat

Abbildung 2h
9. Rat-1-Zellen, zytosolische Färbung, Weitfeld-Mikroskopie 10X, hohe Zellkonfluenz
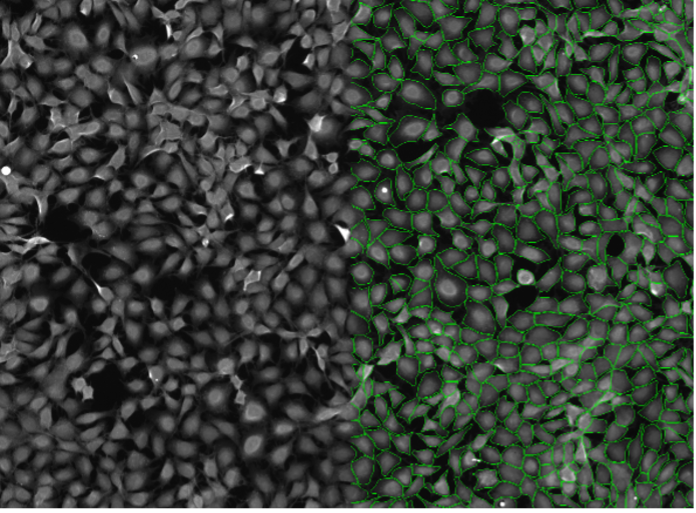
Abbildung 2i
10. SK-HEP-1-Zellen, Zellkontaktfärbung, Weitfeld-Mikroskopie 20X
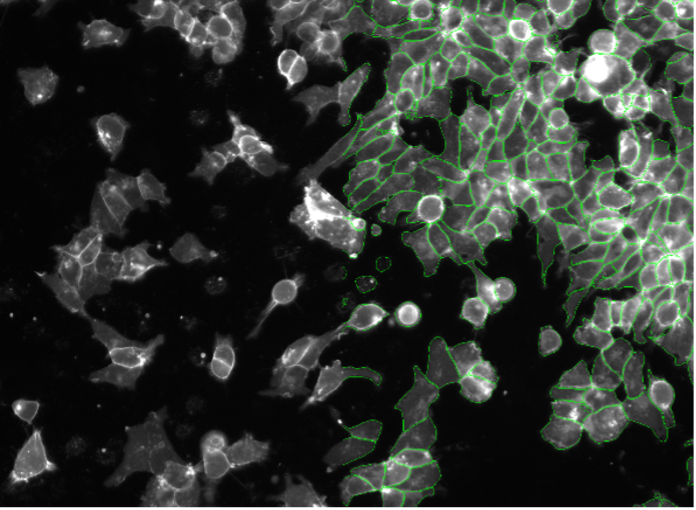
Abbildung 2j
11. Pflanzenzellen, Autofluoreszenz, Weitfeld-Mikroskopie 20X, niedriges Signal-Rausch-Verhältnis
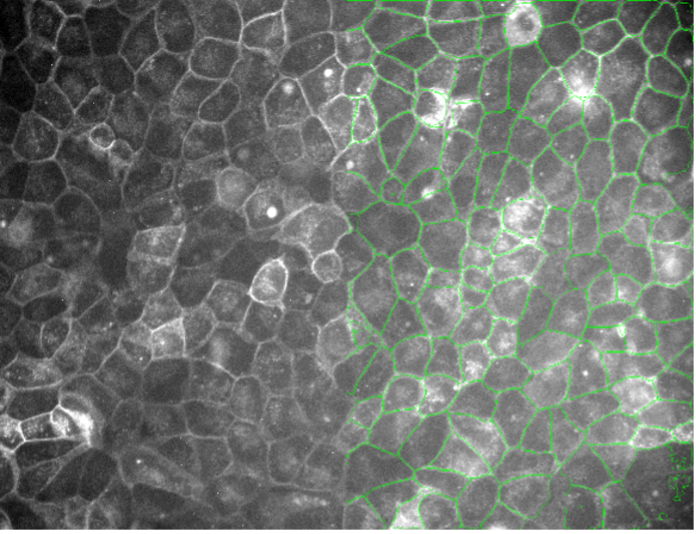
Abbildung 2k
12. HeLa-Zellen, Zellkernmembranfärbung, Weitfeld-Mikroskopie 20X
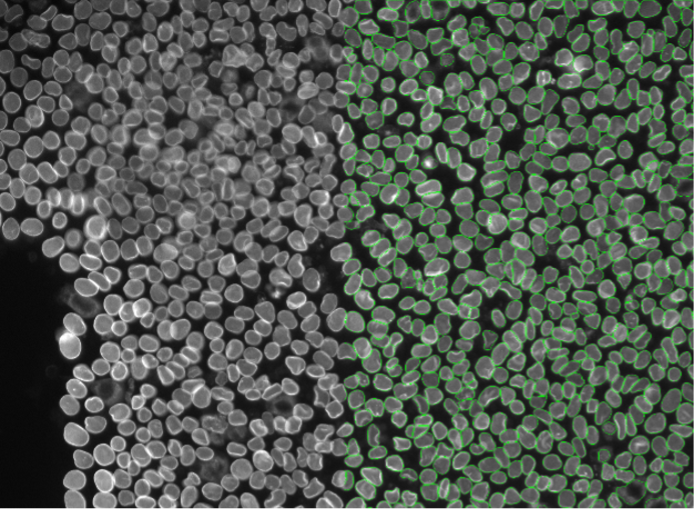
Abbildung 2l
13. huFIB-Zellen, Tubulin-Färbung, Weitfeld-Mikroskopie 20X
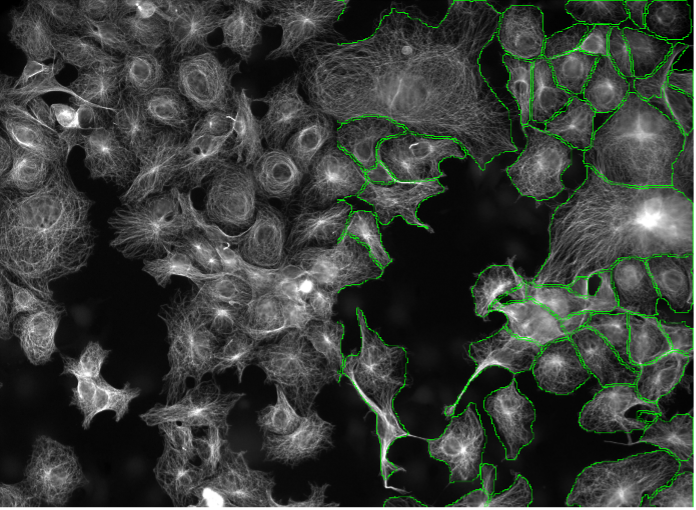
Abbildung 2m
14. Rat-1-Zellen, Aktin-Färbung, Weitfeld-Mikroskopie 20X
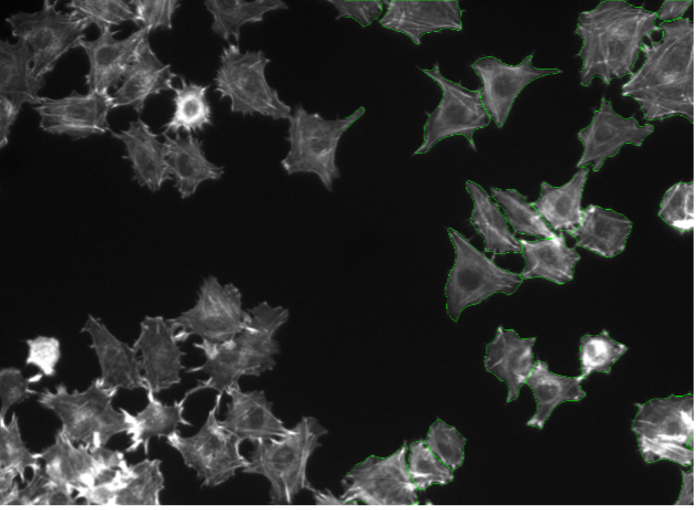
Abbildung 2n
15. BPAE-Zellen, Aktin-Färbung, konfokale Punkt-Scanning-Mikroskopie 40X
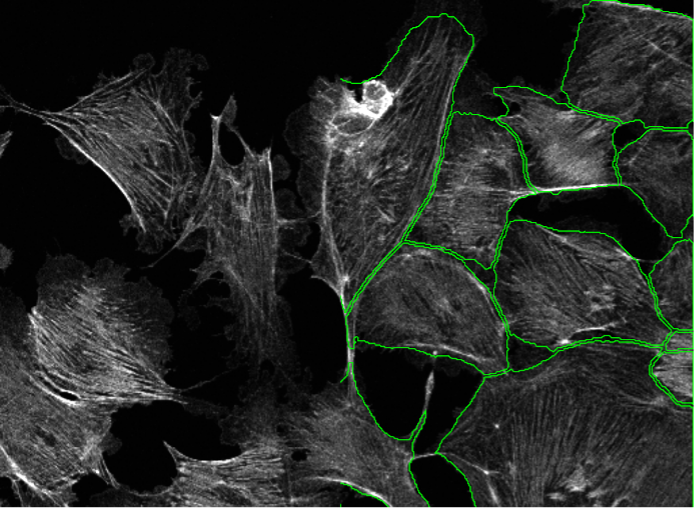
Abbildung 2o
16. Rat-1-Zellen, Zellkernfärbung schwach ins Zytoplasma ausblutend, Weitfeld-Mikroskopie 20X
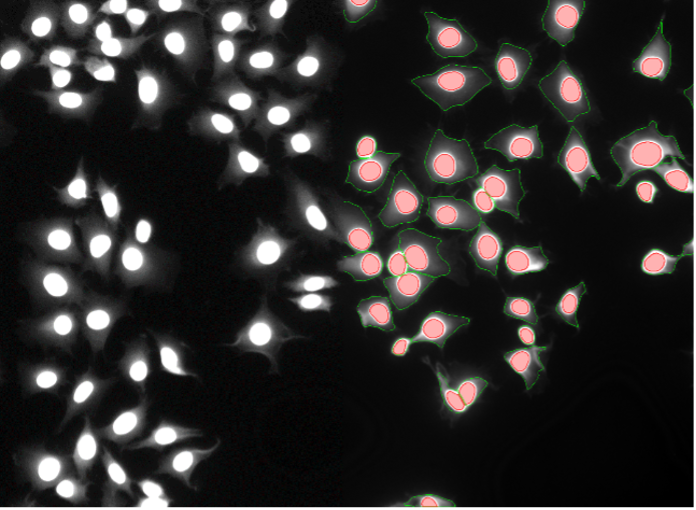
Abbildung 2p
17. HeLa-Zellen, Tubulin-Färbung, Weitfeld-Mikroskopie 4X, niedrige Auflösung und niedriges Signal-Rausch-Verhältnis

Abbildung 2q
18. Rat-1-Zellen, zytosolische Färbung, Weitfeld-Mikroskopie 20X
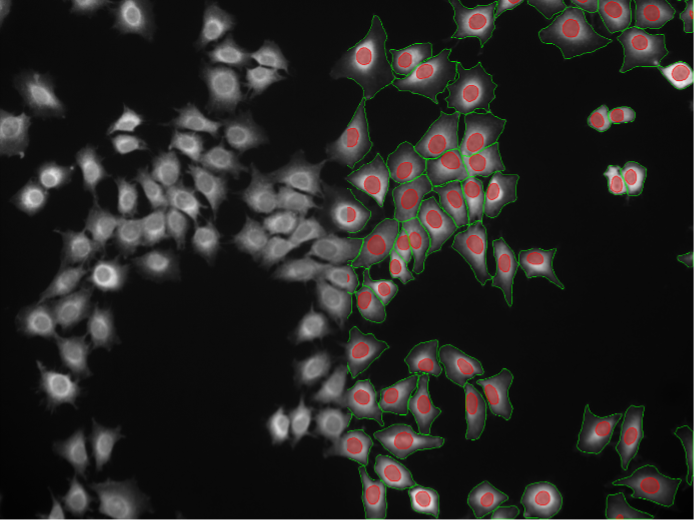
Abbildung 2r
19. COS-7-Zellen, Mitochondrienfärbung, konfokale Punkt-Scanning-Mikroskopie 40X
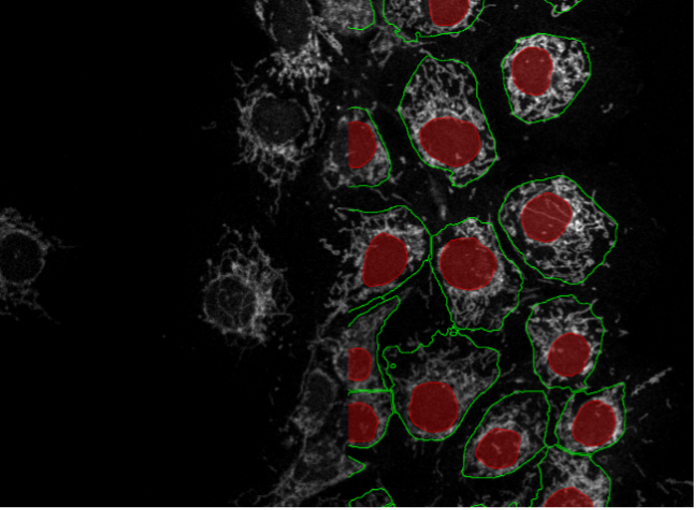
Abbildung 2s
20. BPAE-Zellen, Tubulin-Färbung, konfokale Punkt-Scanning-Mikroskopie 40X
Erweiterte Deep-Learning-Funktionen für die Segmentierung von Zellen und Zellkernen
Die Einführung der Instanzsegmentierung und der vortrainierten Modelle erweitert die Möglichkeiten der TruAI Technologie. Mit den vortrainierten Modellen und durch die mit einem Klick mögliche Segmentierung von Zellen und Zellkernen lässt sich Zeit sparen. Bei suboptimalen Ergebnissen können die TruAI Vorhersagen in neue Beschriftungen umgewandelt und anschließend Korrekturen vorgenommen werden. Diese korrigierten Beschriftungen dienen als Grundlage für das nächste Training, was Zeit und Aufwand spart.
Unsere Kunden sehen bereits die Vorteile der erweiterten Deep-Learning-Funktionen. Robert Strauss, Senior Scientist am Danish Cancer Society Research Center, merkt an: „Die vortrainierte Detektion von Zellkernen ist absolut verblüffend und ermöglicht die Analyse sehr heterogener Proben, ohne die Zellfraktionen zu beschädigen. Vor allem in Bereichen mit hoher Zelldichte ist die TruAI-basierte Separation deutlich schneller und leistungsfähiger als die Intensitäts- oder Kantendetektion.“
Ähnliche Artikel
Vereinfachung der Instanzsegmentierung von Zellen und Zellkernen durch Deep Learning
Webinar: Verbesserte Bildanalyse mit der TruAI Deep-Learning-Technologie