Höchste Auflösung (Super Resolution) für alle Arten des Lebendzell-Imaging
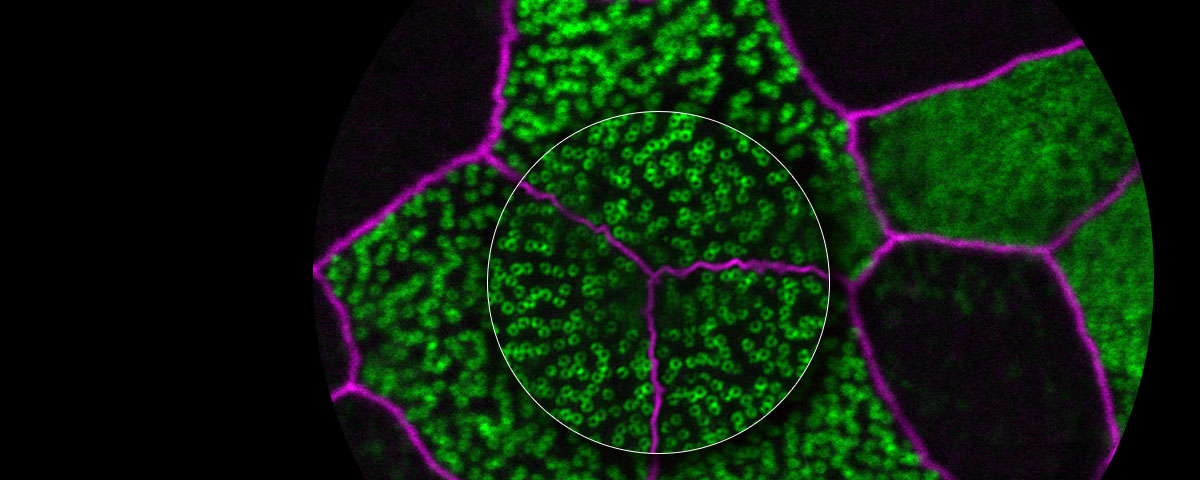
Mit Fluoreszenzmikroskopen lassen sich mithilfe fluoreszierender Sonden spezifische Proteine in vivo identifizieren. Die Auflösung vieler dieser Mikroskope ist wegen der Beugungsgrenze auf etwa 200 nm begrenzt, sodass feine Strukturen nicht dargestellt werden können. Dank der Olympus Super Resolution Technologie, auch als OSR bezeichnet, erhalten Sie scharfe Bilder mit einer Auflösung von bis zu 120 nm in horizontaler Richtung.
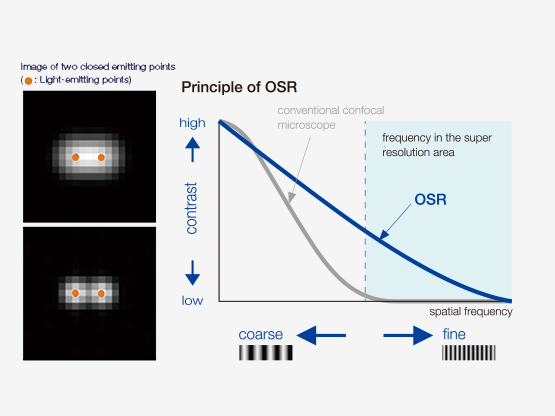
Wie funktioniert OSR?
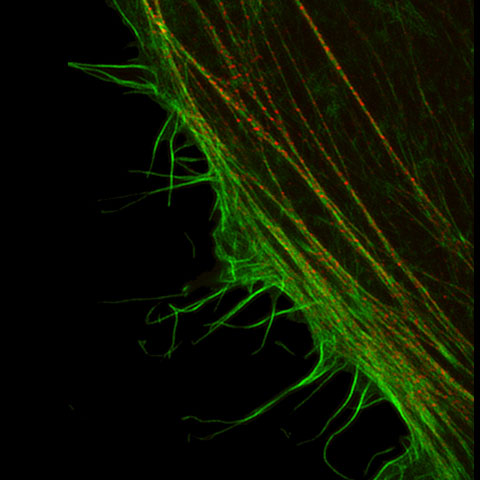
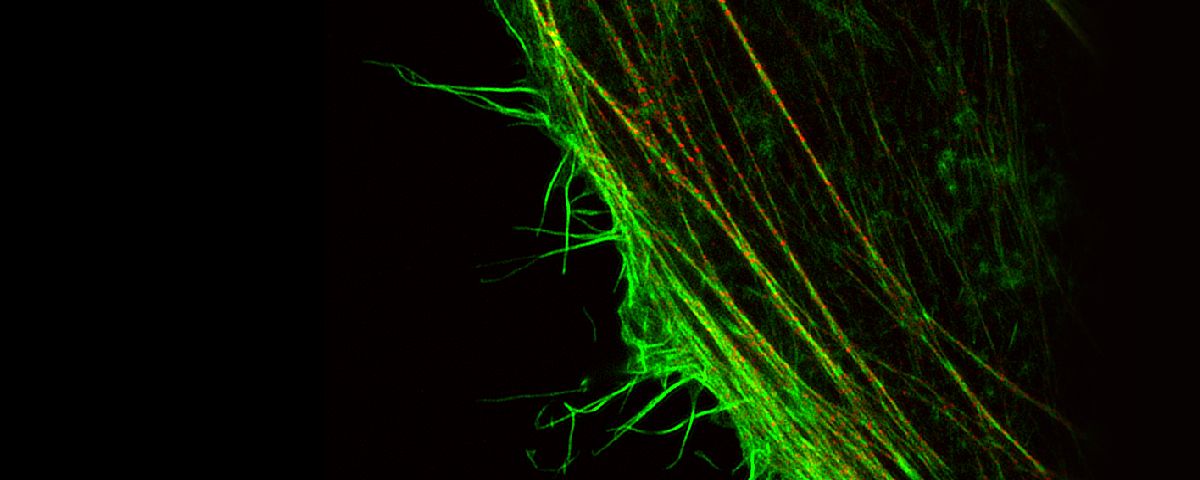
| Durch eine sorgfältig aufeinander abgestimmte Kombination aus verbesserter Detektion, speziellen Hardwareeinstellungen, optimierter konfokaler Aperturdurchmesser und modernster Signalverarbeitung werden Bilder mit höchster Auflösung erstellt. Die OSR-Technologie ermöglicht dabei eine laterale Auflösung (X-Y-Ebene) von bis zu 120 nm. Quelle: |
Olympus Super Resolution (OSR)
IXplore SpinSR10
Weitere Informationen über das SpinSR10 FLUOVIEW FV3000 mit FV-OSR
Weitere Informationen über das FV3000 Erfahren Sie mehr über das Software-Modul für Super Resolution |
Noch mehr sehen
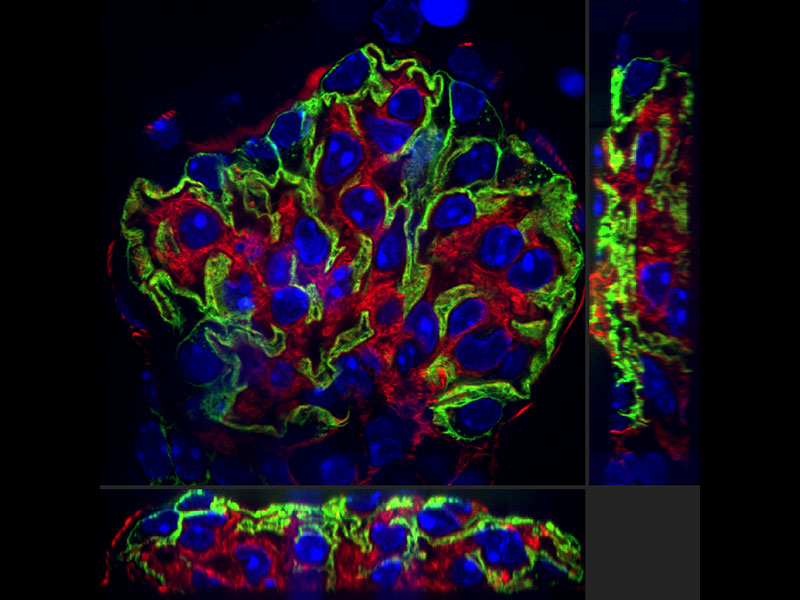
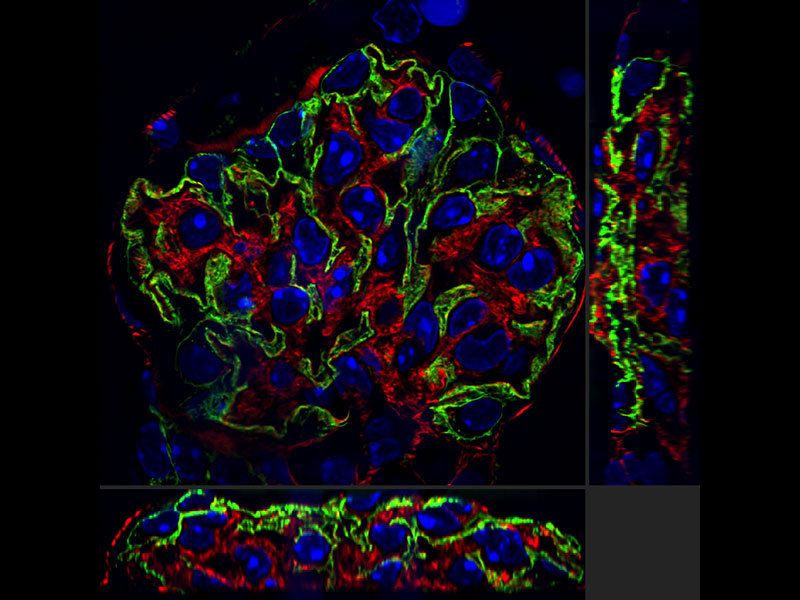
Unsere Dekonvolutionsalgorithmen machen hochaufgelöste Bilder noch klarer und schärfer. Durch iterative Dekonvolution mit 3D-Nebenbedingungen lassen sich Unschärfen in der Z-Achse entfernen und klarere dreidimensionale Bilder aufnehmen.
|
Erfahrungsberichte
Yasushi Okada, M.D. und Ph.D., Riken Quantitative Biology Center | Die Größe der meisten intrazellulären Organellen und supramolekularen Komplexe liegt im Bereich um 100 nm. Mit normalen optischen Mikroskopen ist es einfach nicht möglich, die Struktur dieser Komplexe darzustellen. Derzeit werden verschiedene Methoden für die hochauflösende Mikroskopie entwickelt, die sich, weil sie besondere Farbstoffe, spezielle Beobachtungsbedingungen oder spezielle optische Systeme erfordern, in der Biologie nur schwierig anwenden lassen. Ein Upgrade eines Spinning-Disc-Konfokalmikroskops von Olympus auf ein Super Resolution Mikroskop ist jedoch problemlos möglich. Darüber hinaus trägt die Kombination mit Silikon-Immersionsobjektiven dazu bei, die sphärische Aberration zu verringern, und ermöglicht die Erfassung von Live-Bildern aus tieferen Ebenen von Zellen mit höchster Auflösung. Ich rechne fest damit, dass diese Technologie bei den meisten Anwendungen in der Biologie zum Einsatz kommen wird. |
Sachiko Tsukita, Graduate School of Frontier Biosciences und Graduate School of Medicine, Osaka University, Japan | Wir untersuchen den Mechanismus der koordinierten Anordnung und Ausrichtung der Basalkörperchen, die in zilienbesetzten Zellen die Zilien bilden. Das regelmäßige Muster der Basalkörperchen ist entscheidend für die metachronen Wellen der Zilien. Um die GFP-Centrin-markierten Basalkörperchen während der Differenzierung zilienbesetzter Zellen in einem Primärkultursystem der Trachea scharf darstellen zu können, ist eine Auflösung von 200 nm oder weniger in allen drei Raumachsen (X, Y und Z) erforderlich. Mit einem Lebendzell-Imaging-System auf der Grundlage des SpinSR10 ist es möglich, die Bewegungen der Basalkörperchen zu analysieren. Mithilfe dieser Methode entdeckten wir einen neuartigen Mechanismus, durch den sich die Basalkörperchen im Rahmen der Selbstorganisation des apikalen Zytoskeletts in zilienbesetzten Zellen der Trachea ausrichten. |
Yuji Ikegaya, Ph.D., Laboratory of Chemical Pharmacology, Graduate School of Pharmaceutical Sciences, The University of Tokyo, Japan | Als ich die mit höchster Auflösung aufgenommenen Fluoreszenzbilder des SpinSR10-Mikroskops zum ersten Mal sah, war ich von ihrer Schönheit überwältigt. Ich untersuche die Plastizität von Mikrostrukturen im Gehirn, insbesondere der Postsynapse (Rückenmark), der Nervenfasern und der Fortsätze von Gliazellen. Anhand der Bilder, die ich mit dem SpinSR10 erhalten hatte, hatte ich endlich das Gefühl, „sehen“ zu können. Durch die Verwendung einer anderen Methode als der Elektronenmikroskopie zur detaillierten Aufnahme von Mikrostrukturen verbesserte sich die Zuverlässigkeit der Daten, und die Aussagekraft des Experiments war dank eines vereinfachten Datenerfassungsverfahrens deutlich stärker. Darüber hinaus gelang durch die Realisierung längerer Zeitrafferaufnahmen die Einführung neuer Forschungsmethoden. |
Literaturnachweise
S. Hayashi and Y. Okada, “Ultrafast superresolution fluorescence imaging with spinning disk confocal microscope optics,” Mol. Biol. Cell 26(9), 1743–1751 (2015).
S. Hayashi, “Resolution doubling using confocal microscopy via analogy with structured illumination microscopy,” Jpn. J. Appl. Phys. 55(8), 082501 (2016).
A. Nagasawa-Masuda and K. Terai, “Yap/Taz transcriptional activity is essential for vascular regression via Ctgf expression and actin polymerization,” PLoS ONE 12(4), e0174633 (2017).
H. Nakajima, et al., “Flow-Dependent Endothelial YAP Regulation Contributes to Vessel Maintenance,” Dev. Cell 40(6), 523-536.e6 (2017).
K. Tateishi, et al., “Three-dimensional Organization of Layered Apical Cytoskeletal Networks Associated with Mouse Airway Tissue Development,” Sci. Rep. 7, 43783 (2017).
E. Herawati, et al., “Multiciliated cell basal bodies align in stereotypical patterns coordinated by the apical cytoskeleton,” J. Cell Biol. 214(5) 571-586 (2016).
M.-T. Ke, et al., “Super-Resolution Mapping of Neuronal Circuitry With an Index-Optimized Clearing Agent,” Cell Rep. 14(11) 2718–2732 (2016).
Sorry, this page is not
available in your country.
Sorry, this page is not
available in your country.
.jpg?rev=2087)