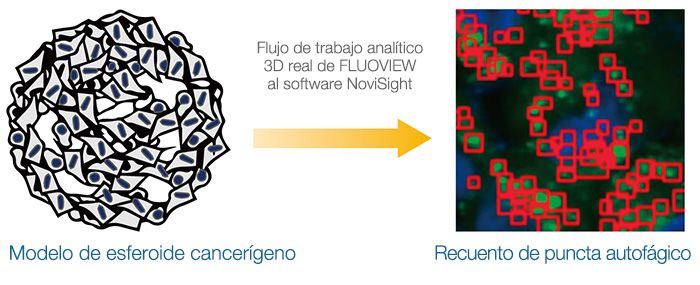
Análisis 3D real de la trayectoria autofágica intracelular en un modelo de esferoide 3D
Resumen
Se han usado modelos de cultivos 3D para predecir la eficacia y la toxicidad de los fármacos. Y, esta nota de aplicación se centra en la trayectoria autofágica intracelular. En primer lugar, se confirmó la utilidad de la sonda EGFP-LC3 para monitorear el estado autofágico de los modelos. Después, se usó el flujo de trabajo analítico 3D real con el microscopio confocal FLUOVIEW™ FV3000 y el software NoviSight™ para evaluar de forma cuantitativa el efecto de un candidato a fármaco, denominado cloroquina. Se demostró que este flujo de trabajo analítico ofrece una alternativa a la creación de perfiles de candidatos a fármacos usando estudios in vivo.
Introducción
Los episodios moleculares intracelulares se encuentra muy relacionados con diversas patologías que sufren los humanos. La autofagia es un proceso intracelular que degrada y recicla las proteínas y los orgánulos celulares; y, la desregulación de la autofagia acarrea enfermedades humanas. Para comprender mejor estos procesos de las enfermedades y probar la eficacia de los fármacos en los mismos, es importante monitorear el estado autofágico de las células humanas bajo condiciones patológicas y terapéuticas.
Los modelos de cultivos 3D, como los esferoides o los orgánulos, pueden simular el complejo microentorno in vivo. Para evaluar la eficacia de un fármaco en las células del esferoide, es fundamental analizar de forma cuantitativa las imágenes microscópicas intracelulares. En esta nota de aplicación, se evalúo un proceso de autofagia intramolecular dinámico y analizó el efecto de un candidato a fármaco usando el microscopio confocal FV3000 de Olympus y el flujo de trabajo analítico 3D real del software de análisis NoviSight.
Beneficios
- Análisis cuantitativo de episodios intramoleculares en modelos de cultivos 3D
Resultados y debate
Una proteína LC3 es un marcador útil en un proceso autofágico. Por ejemplo, se ha demostrado que los puntos de fluorescencia de EGFP-LC3 aumentaron a medida que se acumularon autofagosomas. La formación de autofagosomas está regulada por varios compuestos que modulan las actividades autofágicas o el movimiento de los autofagosomas. Algunos de estos compuestos ahora es encuentran en ensayos clínicos. Uno de ellos, la cloroquina (CQ), originalmente un fármaco para combatir la malaria, se está sometiendo a un ensayo clínico para el tratamiento del cáncer, ya que inhibe la fusión de los autofagosomas y los liposomas.
Validación de un marcador de autofagia usando un sistema de cultivo 2D
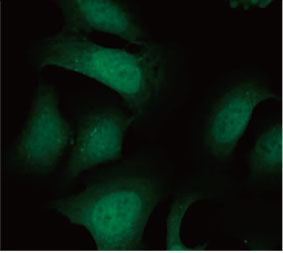
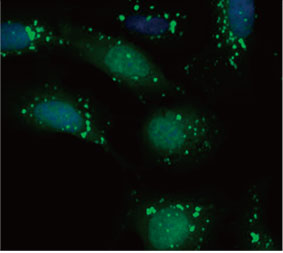
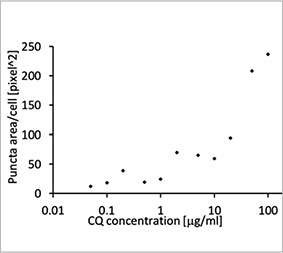
Se validó la utilidad de una sonda EGFP-LC3 para medir el efecto inhibitorio de la CQ en el proceso autofágico de un sistema de cultivo 2D. Se preparó una línea celular de osteosarcoma U2OS con expresión de EGFP-LC3. Las células se cultivaron como monocapas en pocillos de microplaca. Después de añadir la CQ, las células se fijaron y tiñeron con Hoechst 33342. Las imágenes de fluorescencia mostraron que el tratamiento con CQ evocaba la formación de puncta de EGFP-LC3 (Figuras 1A y B). La zona de puncta por cada célula se midió partiendo de la posición del núcleo de cada célula. El área de puncta aumentaba a medida que la concentración de CQ incrementaba, lo que confirmó la utilidad de la sonda EGFP-LC3 para medir un proceso autofágico.
(A)
| (B)
| (C)
|
Figuras 1A y 1B: (A) Células de U2OS con expresión de EGFP-LC3 cultivadas en condiciones normales. (B) Células de U2OS con expresión de EGFP-LC3 tratadas con CQ durante una hora. Figura 1C: El aumento de la zona punteada para cada célula se hizo evidente con el aumento de la concentración de CQ. | ||
Cuantificación de la zona punteada de LC3 en el modelo de esferoide cancerígeno
Para monitorear el estado autofágico en modelos de cultivos en 3D, se escogió la línea celular de HeLa establecida de un carcinoma cervical. Las células de HeLa son adecuadas para llevar a cabo un protocolo de cultivo 3D sin matriz y formar esferoides cancerígenos de tamaño uniforme. Se preparó una línea celular de HeLa con expresión de EGFP-LC3.
Las células se repartieron en microplacas de baja fijación con la base redonda. Después de un cultivo de cinco días, los esferoides fueron tratados con CQ durante siete horas. Después, los esferoides fueron fijados, su membrana fue permeabilizada, su núcleo fue teñido y finalmente fueron aclarados con SCALEVIEW-S4 (FUJIFILM WAKO).
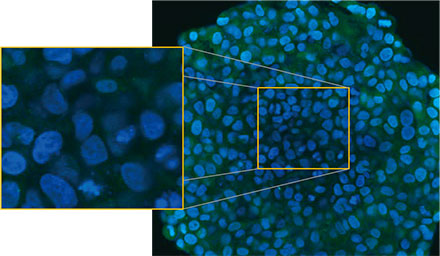
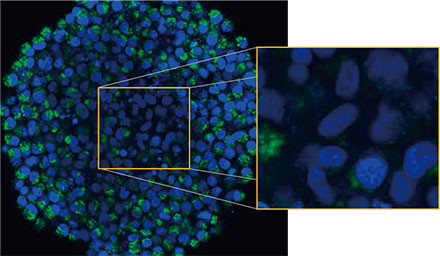
Los esferoides fueron observados usando el sistema confocal FLUOVIEW™ FV3000 de Olympus. Se usó un objetivo de inmersión en silicona (UPLSAPO30XSIR) por su alta apertura numérica (A.N.) y redujimos la aberración esférica. La Figura 2 (A)* muestra una imagen única de segmento Z del esferoide no tratado en el ecuador. La señal de EGFP apareció de forma uniforme en las células. Contrariamente, el tratamiento con CQ indujo la formación de señales de puncta del EGFP en las células (Figura 2 (B)*). Las formaciones de puncta se observaron en la zona periférica y en la zona central del esferoide. El tratamiento con CQ no provocó cambios aparentes en el tamaño del esferoide, lo que indicaba que el proceso autofágico intracelular podría requerir un mejor marcador para evaluar la eficacia de la CQ en esferoides cancerígenos en 3D.
Para analizar de forma cuantitativa los episodios intracelulares en los modelos de cultivos 3D, se usó el flujo de trabajo analítico 3D real de Olympus que combina las imágenes capturadas con el microscopio confocal FV3000 y el análisis con el software NoviSight™, el cual ofrece muchos beneficios con respecto a los métodos analíticos convencionales. En un análisis convencional, se usaría el valor característico integrado de los objetos reconocidos de diversas imágenes de segmento Z o se analizaría la imagen de proyección de varias imágenes de segmento Z. Estos métodos no son fiables, ya que pueden hacer que se cuenten objetos de interés varias veces o que se omitan objetos de interés solapados y oscurecidos por otros objetos. Pero, el flujo de trabajo analítico 3D real de Olympus incluye la adquisición de imágenes secuenciales de segmento Z con intervalos Z optimizados por el software FLUOVIEW™. El software reconstruyó el volumen 3D y reconoció los objetos de interés como agrupaciones de vóxeles. A continuación, se usó el software NoviSight para los análisis estadísticos. El flujo de trabajo de FLUOVIEW al software NoviSight es muy sencillo y ofrece una plataforma ideal para análisis de modelos de cultivos 3D.
(A)
| (B)
|
Figuras 2A y 2B: Esferoides de células HeLa con expresión de EGFP-LC3 (A) en condiciones normales y (B) tratados con CQ. Las imágenes ampliadas muestran la región del rectángulo amarillo de las imágenes originales de los esferoides.* | |
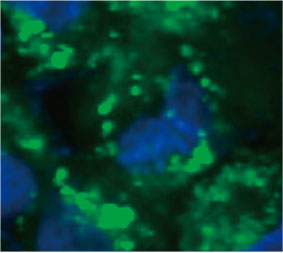
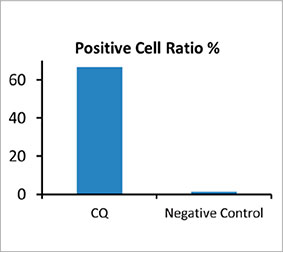
Para calcular el número de células de autofagia positiva en el esferoide, el software identificó el núcleo de cada célula. A continuación, se definieron las regiones que rodeaban cada núcleo como regiones de máscara. Finalmente, las puncta de las señales EGFP en las regiones de la máscara se reconocieron como objetos de puncta [Figura 2 (C)*]. Las células que tienen más de tres punctas se consideran células de autofagia positiva. El esferoide 3D sin tratamiento de fármaco tenía un 1,4 % de células de autofagia positiva, mientras que el esferoide tratado con CQ mostró un 66,6% de células de autofagia positiva [Figura 2 (D)*]. El análisis cuantitativo también sostiene que la CQ indujo la formación de punctas en los esferoides cancerígenos.
(C)
|
| (D)
|
Figura 2C - Izquierda: Imagen original de las células en el esferoide con CQ. Derecha: Misma imagen con reconocimiento de objeto para puncta (rectángulos rojos).* | ||
Conclusión
Se usó el microscopio confocal FLUOVIEW FV3000 de Olympus y el software NoviSight, como parte de un flujo de trabajo analítico 3D real, para evaluar el efecto de la CQ en el proceso autofágico intracelular en modelos de cultivos en 3D. Este estudio demuestra que los investigadores pueden usar este flujo de trabajo para crear perfiles de eficiencia relativos a los fármacos, de forma supeditada a la dosis, o analizar varios candidatos de fármacos de manera objetiva. Existen otros marcadores moleculares intracelulares que son cruciales para la eficacia o la toxicidad del fármaco, como las señales FISH en los cromosomas o la translocación de moléculas de señalización etiquetadas con fluorescencia. Este flujo de trabajo puede ayudar a evaluar cuantitativamente un amplio rango de episodios intracelulares en modelos de cultivos 3D y ofrecer una creación de perfiles alternativa para candidatos de fármacos en lugar de los estudios in vivo.
Referencias
- Maria C et al., (2019) Targeting autophagy to overcome human disease. International Journal of Molecular Sciences. 20(3): E725. doi: 10.3390/ijms20030725
- Yan C et al., (2013) Therapeutic targeting of autophagy in disease: Biology and Pharmacology. Pharmacological Reviews. 65: 1162–1197. doi: 10.1124/pr.112.007120
- Daniel JK et al., (2008) Guidelines for the use and interpretation of assays for monitoring autophagy in higher eukaryotes. Autophagy. 4(2): 151–175. doi: 10.4161/auto.5338
Author
Kazuhito Goda
*Si bien estas líneas celulares forman parte de las más importantes para la investigación médica, es imperativo reconocer la contribución de Henrietta Lacks a la ciencia que se produjo sin su consentimiento. Esta injusticia, a pesar de haber dado lugar a descubrimientos clave en inmunología, enfermedades infecciosas y cáncer, también ha generado importantes debates sobre la privacidad, la ética y el consentimiento en la medicina.
Para obtener más información sobre la vida de Henrietta Lacks y su contribución a la medicina moderna, haga clic aquí.
http://henriettalacksfoundation.org/
Productos usados para esta aplicación
se ha añadido correctamente a sus marcadores
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
Not Available in Your Country
Sorry, this page is not
available in your country.