La tecnología de aprendizaje profundo TruAI™ es un enfoque innovador de análisis de imágenes ya que permite a los investigadores abordar aplicaciones que eran imposibles o desafiantes, como el análisis sin marcado fiable o el seguimiento de las células vivas a intensidades muy bajas en diversos días.
Para estas aplicaciones, el análisis se realiza a través de la segmentación semántica: la capacidad de identificar si un píxel pertenece al fondo o al primer plano de una clase específica. Después de la segmentación semántica, se aplican los pasos de posprocesamiento como el watershed para realizar la segmentación final del objeto (normalmente el núcleo o células enteras).
Recientemente, la tecnología de aprendizaje profundo TruAI se ha mejorado con la introducción de la segmentación de instancia. Esta funcionalidad fusiona la segmentación semántica y la posterior división de objetos en un solo paso. La segmentación de instancia mejora los flujos de trabajo y le permite abordar aplicaciones complejas en un solo paso, como la segmentación de células en colonias celulares que requieren pasos de división posteriores al procesamiento.
La tecnología de aprendizaje profundo TruAI permite a los usuarios realizar el aprendizaje con sus propios datos desde el principio, por lo que los modelos de red neuronal profunda (DNN) de TruAi generados tienen un rendimiento increíblemente bueno. Los investigadores pueden desarrollar bibliotecas de DNN para diversas aplicaciones e incluso compartirlas con sus colaboradores.
Con todo, esto implica que el flujo de trabajo de deep-learning siempre consiste en tres pasos:
- Marcado con sus propios datos
- Formación
- Inferencia
A pesar de que el software es muy intuitivo, el paso de marcado es el más laborioso, especialmente para aplicaciones complejas en las que tienen que marcarse muchos objetos para alcanzar un buen rendimiento.
Segmentación simplificada de células y núcleos usando modelos de aprendizaje profundo formados previamente
Para mejorar este flujo de trabajo, la tecnología de aprendizaje profundo TruAI ahora incluye algunos modelos formados previamente para núcleos y células, que son los objetos más comunes que se segmentan en análisis de imágenes biológicas. Gracias a ello, los usuarios pueden ahorrar el tiempo dedicado al marcado y la formación (Figura 1).

Figura 1. La utilización de la tecnología de aprendizaje profundo TruAI con sus propios datos de aplicaciones para la formación y la inferencia ayuda a conseguir un rendimiento óptimo. Para un enfoque más rápido, los modelos TruAI formados previamente le permiten realizar el análisis en tan solo un clic.
Los modelos de TruAI formados previamente para núcleos y células se han formado para realizar la segmentación de instancia en imágenes de fluorescencia para diversos escenarios, como:
- Modalidades de microscopía (confocal de disco giratorio, confocal de barrido de puntos, campo amplio)
- Tinciones (DAPI, faloidina, MitoTracker, Lamin, etc.)
- Líneas celulares (HeLa, U2OS, SK-HEP-1, células vegetales, tejido, etc.)
- Contraste, relación señal-ruido y niveles de fondo
- Resolución de píxeles y magnificaciones
Cabe destacar que los modelos formados previamente son modelos generales y no pueden llevar a cabo la segmentación perfectamente en todas las situaciones posibles. Para ayudarle a entender mejor estas funcionalidades, hemos creado una lista de escenarios de segmentación celular.
20 ejemplos de segmentación de núcleos y células usando modelos de aprendizaje profundo formados previamente
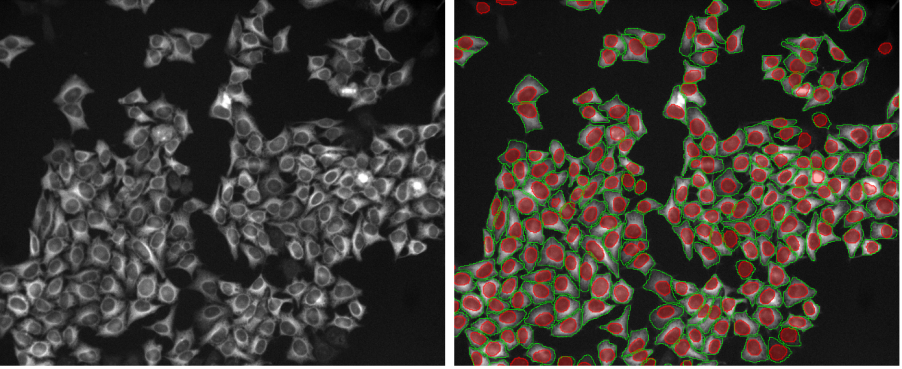
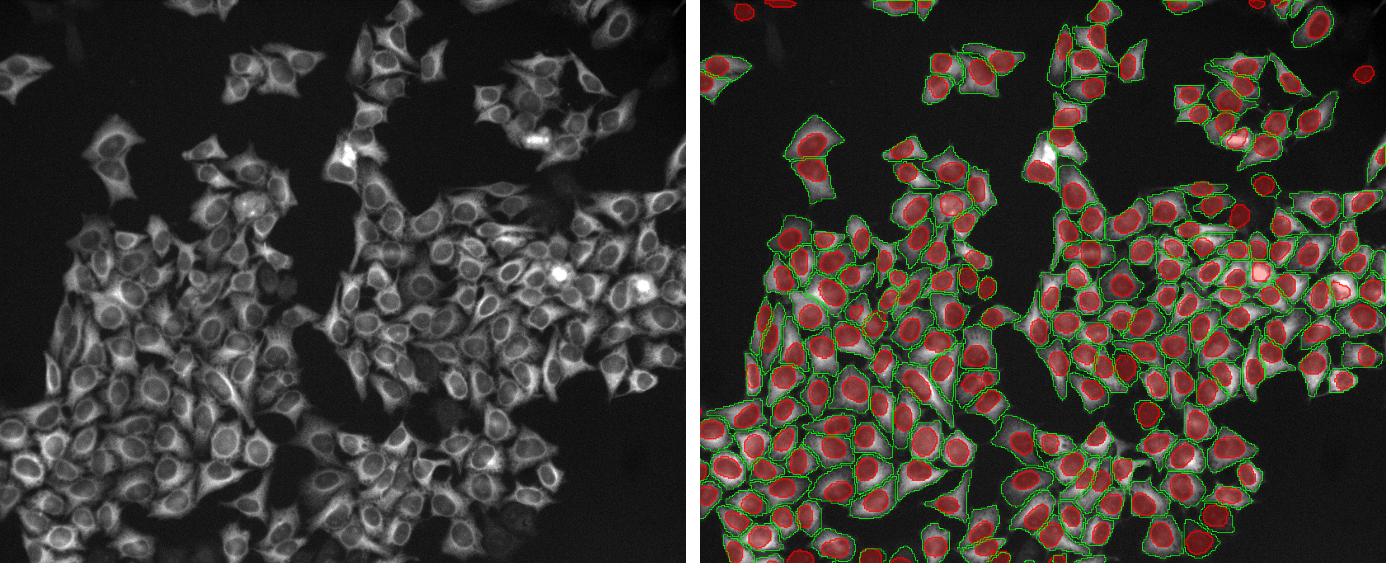
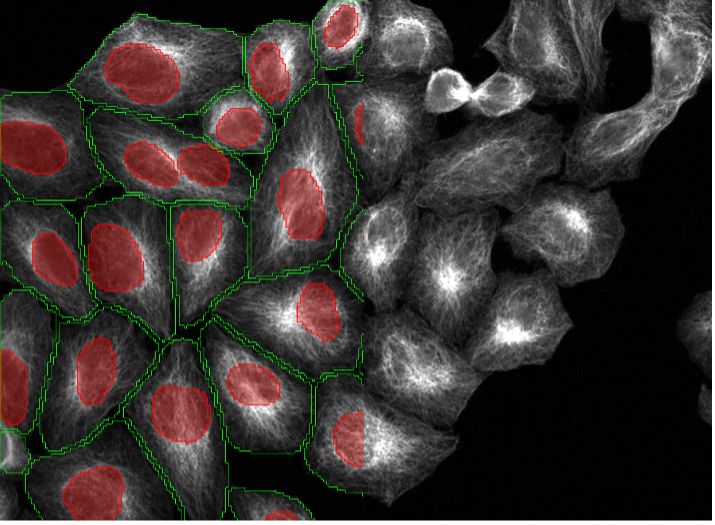
Las imágenes de ejemplo de más abajo (Figure 2, a–t) muestran el rendimiento de los modelos de TruAI formados previamente para el núcleo (rojo) y la célula (green) en diversos escenarios de segmentación. Esto incluye líneas celulares distintas, tinciones de marcador, magnificaciones, resoluciones, contraste, relaciones señal-ruido y desigualdades de fondo. Los dos modelos pueden aplicarse a la misma imagen (Figura 2, p-t). Los modelos pueden incluso aplicarse a núcleos no específicos (Figura 2, q–t) o tinciones celulares (Figura 2p, s).
1. Células U2OS, tinción nuclear, campo amplio 20x, confluencia media con células de distintos tamaños y morfologías
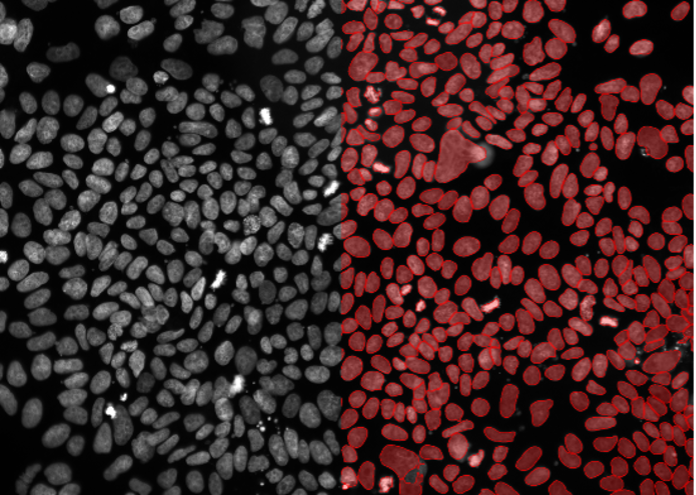
Figura 2a
2. Células de HeLa, tinción nuclear, campo amplio 10x, fondo desigual
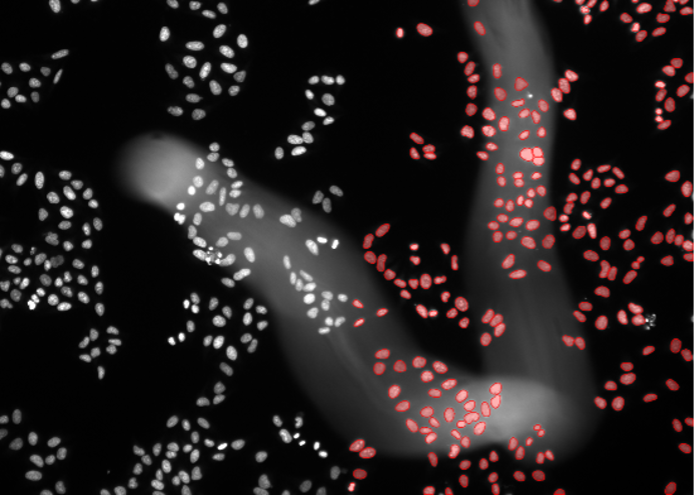
Figura 2b
3. Células madre pluripotentes, marcador nuclear, campo amplio 20x, confluencia muy alta

Figura 2c
4. Células de orgánulos de riñón, tinción nuclear, disco giratorio confocal 30x, alta profundidad de penetración
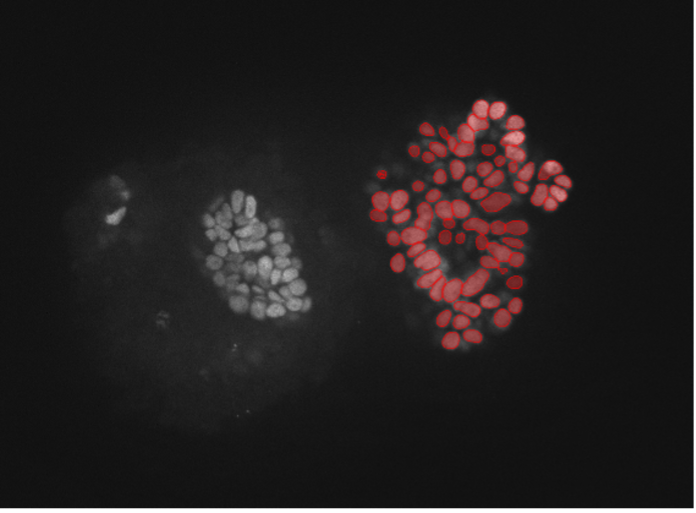
Figura 2d
5. Células de HeLa, tinción nuclear, disco giratorio confocal 40x, niveles altos de contraste y resolución

Figura 2e
6. Células de carcinoma humano A549, marcador nuclear, disco giratorio confocal 40x, baja relación señal-ruido
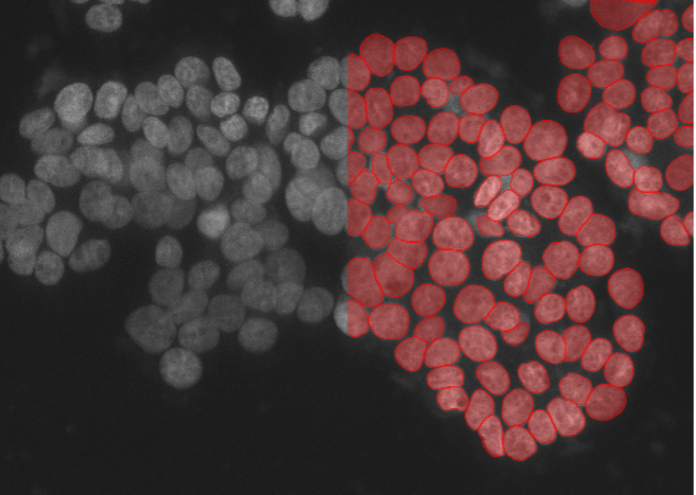
Figura 2f
7. Células de tumor de tejido humano, tinción nuclear, campo amplio 20x

Figura 2g
8. Células de tejido de riñón de ratón, tinción nuclear, campo amplio 20x, muestra gruesa

Figura 2h
9. Células Rat-1, tinción citosólica, campo amplio 20x, confluencia celular alta
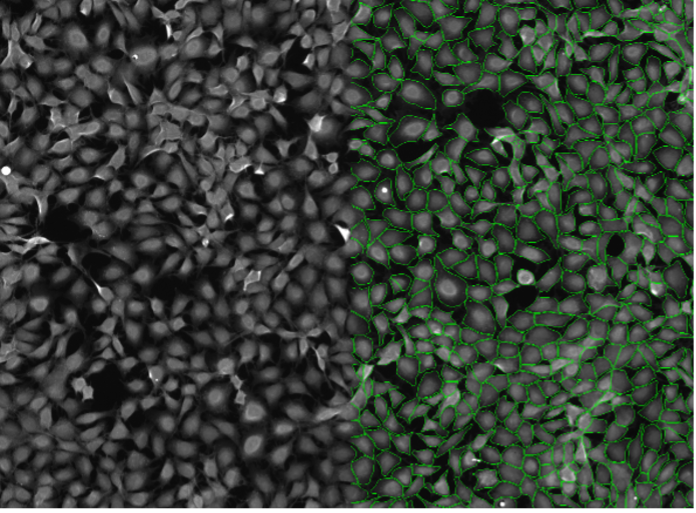
Figura 2i
10. Células SK-HEP-1, tinción de contacto celular, campo amplio 20x
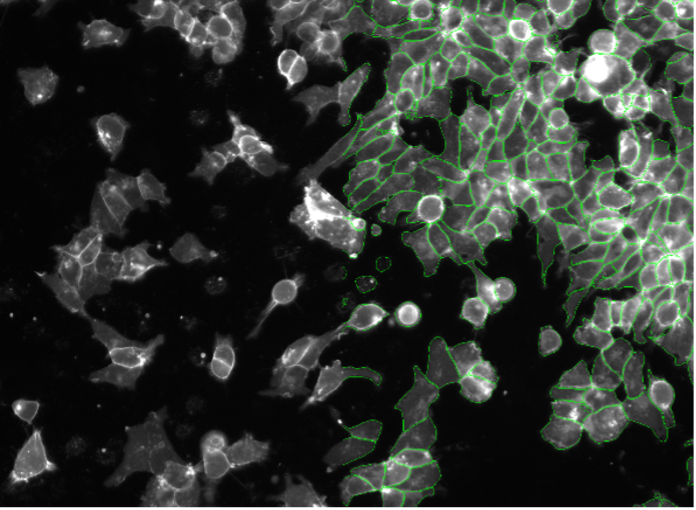
Figura 2j
11. Células vegetales, autofluorescencia, campo amplio 20x, baja relación señal-ruido
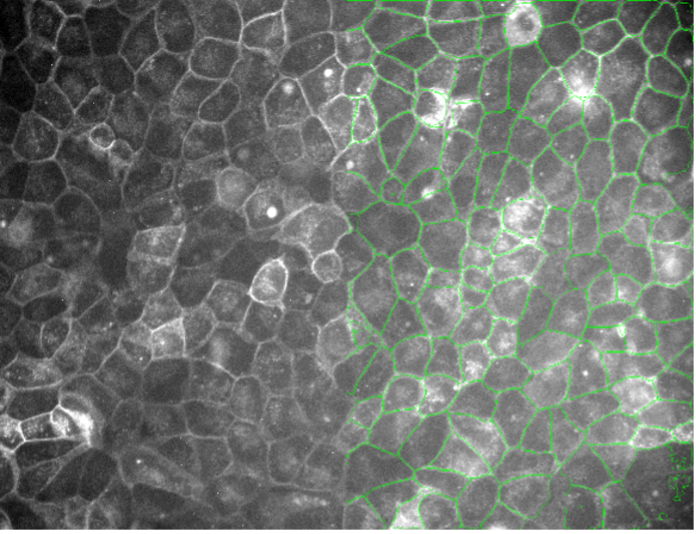
Figura 2k
12. Células de HeLa, tinción de membrana nuclear, campo amplio 20x
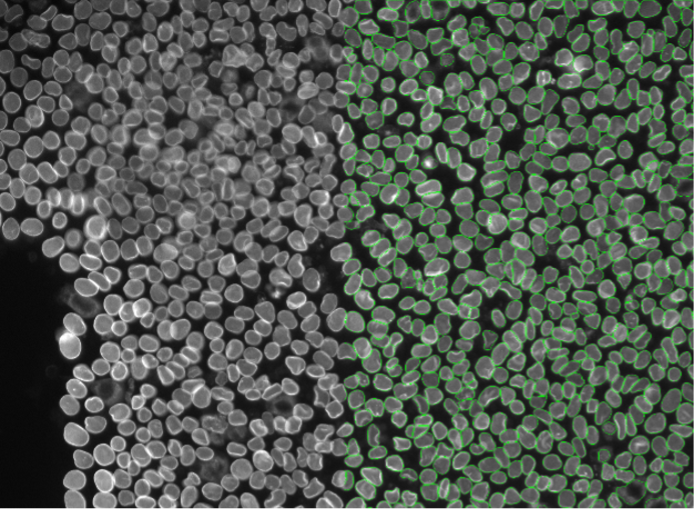
Figura 2l
13. Células de huFIB, tinción con tubulina, campo amplio 20x
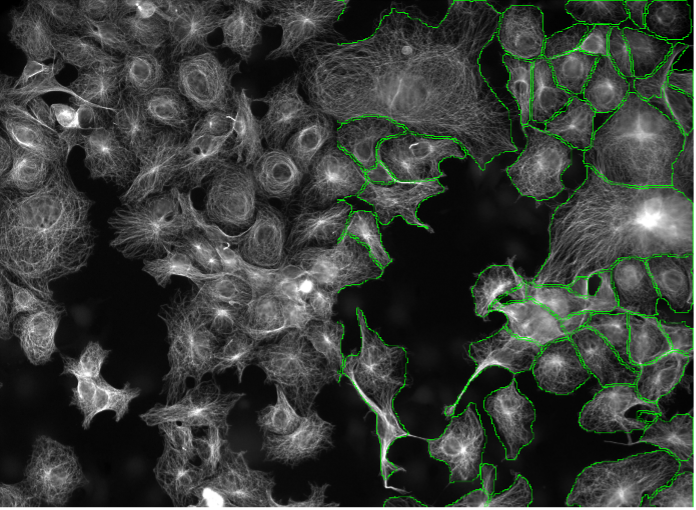
Figura 2m
14. Células Rat-1, tinción con actina, campo amplio 20x
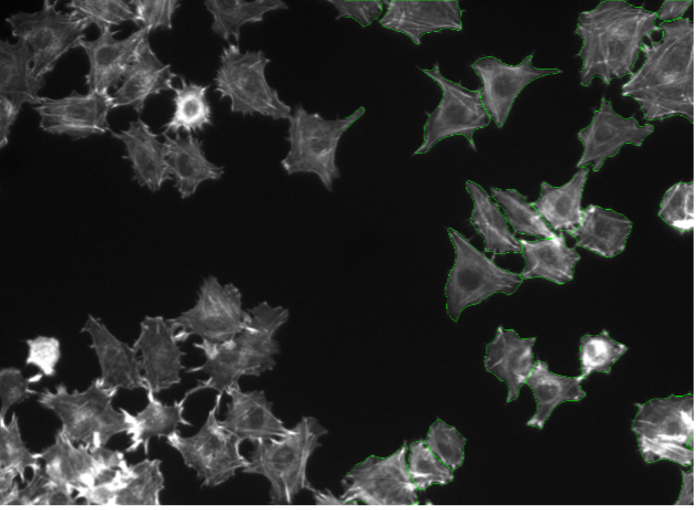
Figura 2n
15. Células BPAE, tinción con actina, barrido de puntos confocal 40x
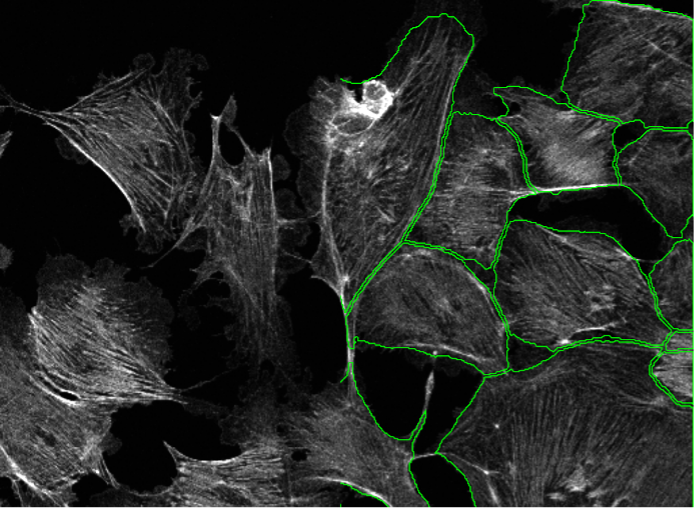
Figura 2o
16. Células Rat-1, tinción nuclear con sangrado débil en el citoplasma, campo amplio 20x
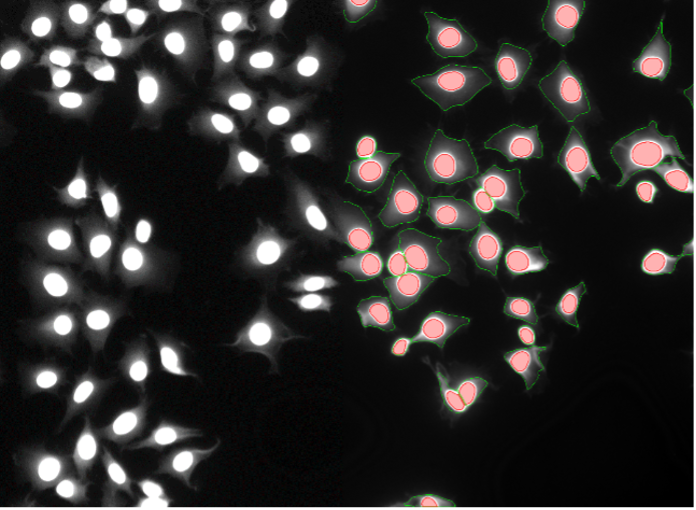
Figura 2p
17. Células de HeLa, tinción con tubulina, campo amplio 4x, resolución baja y relación señal-ruido baja

Figura 2q
18. Células Rat-1, tinción citosólica, campo amplio 20x
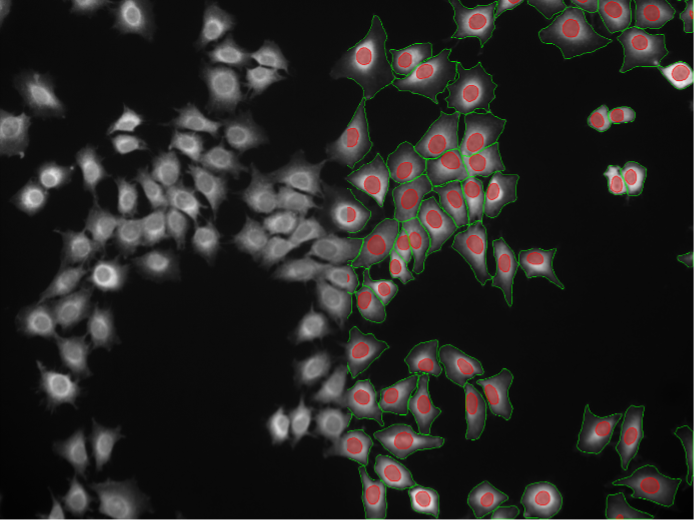
Figura 2r
19. Células COS-7, tinción del mitocondrias, barrido de puntos confocal 40x
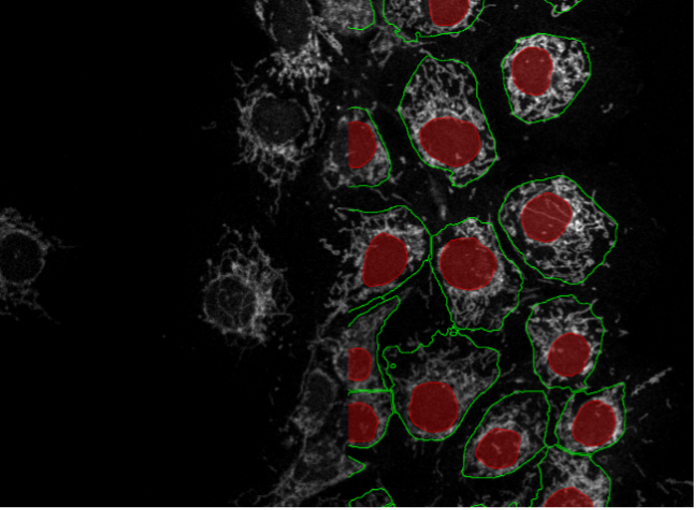
Figura 2s
20. Células BPAE, tinción con tubulina, barrido de puntos confocal 40x
Funcionalidades ampliadas de aprendizaje profundo para la segmentación de células y núcleos
La introducción de la segmentación de instancia y los modelos formados previamente amplía las funcionalidades del conjunto de herramientas TruAI. Ahora los usuarios pueden ahorrar mucho tiempo empezando con los modelos formados previamente y realizando la segmentación de células y núcleos con un solo clic. Para los casos en los que los resultados no son óptimos, los usuarios pueden convertir las predicciones TruAI en nuevas etiquetas y aplicar después las correcciones. Estas etiquetas corregidas sirven de base para la nueva formación, lo que ahorra tiempo y esfuerzos.
Nuestros clientes ya están viendo de primera mano los beneficios de las funcionalidades ampliadas de aprendizaje profundo. Robert Strauss, científico sénior en el Danish Cancer Society Research Center, comentaba "El reconocimiento de los núcleos formados previamente es absolutamente asombroso y ahora puedes analizar fácilmente muestras muy heterogéneas sin comprometer las fracciones celulares. Especialmente en áreas de alta densidad celular, la separación basada en TruAI es claramente superior a la intensidad o la detección de bordes, tanto a nivel de velocidad como de rendimiento".
Contenido relacionado
Segmentación de instancias simplificada de células y núcleos mediante el aprendizaje profundo
Seminario web: Mejore sus análisis de imágenes con la tecnología de aprendizaje profundo TruAI