El Hospital Nacional de Parapléjicos (HNP), ubicado en Toledo (España), es uno de los centros de referencia españoles para los pacientes con lesiones de médula espinal. Los objetivos del HNP son los siguientes: proporcionar servicios integrales de salud y rehabilitación a personas con una lesión de médula espinal; capacitar al personal cualificado, y efectuar investigaciones científicas en el campo neurocientífico.
Con el fin de respaldar la investigación neurocientífica, el HNP facilita instalaciones de investigación para la microscopía y el análisis de imágenes, la citometría de flujo, la proteómica, la resonancia magnética nuclear (RMN) en animales pequeños, y el alojamiento para animales de laboratorio. La instalación dedicada a la microscopía y el análisis de imágenes cuenta con una poderosa solución multimodal que soporta el escaneo de portaobjetos completos y el cribado de alto contenido (HCS) a través de un microscopio.
Esta solución combina el sistema microscópico automatizado IXplore™ Pro con el software de cribado de alto contenido scanR* y el software de procesamiento de imágenes cellSens™. Ésta puede adaptarse a una amplia variedad de aplicaciones y técnicas de procesamiento de imágenes, como el análisis avanzado de imágenes mediante la tecnología de aprendizaje profundo TruAI™.

Figura 1. Vista panorámica del Hospital Nacional de Parapléjicos en Toledo, España. Fotografía por cortesía de Juan Carlos Monroy.
La instalación de microscopía y análisis de imágenes del Hospital Nacional de Parapléjicos está dirigida por el Dr. José Ángel Rodríguez Alfaro y Dr. Javier Mazarío. Hace poco han compartido su experiencia con respecto al uso del sistema IXplore Pro con los softwares scanR y cellSens en los servicios centrales de la instalación.
P: ¿Por qué consideraron el sistema microscópico IXplore y su software scanR para su instalación?
R: En nuestra instalación contamos con una variedad de microscopios ópticos, que van desde microscopios confocales de escaneo láser automatizados hasta microscopios manuales de rutina. Anteriormente, hemos tenido un sistema de cribado de alto contenido (HCA) en caja que se malogró después de haber funcionado 10 años, y necesitábamos un reemplazo.
Cuando esto sucedió, nos encontrábamos buscando formas de exportar los datos que habían sido obtenidos con dicho sistema HCA a un software de análisis de citometría de flujo, el cual permitiría un análisis controlado más refinado de las mediciones.
Lo primero que llamó nuestra atención sobre el sistema scanR fue que este tipo de análisis citométrico controlado ya estaba integrado en el software. [Hasta ese momento], no habíamos encontrado ningún otro sistema HCA que pudiese llevar a cabo esta clase de análisis. Este fue uno de los factores clave que nos hizo preferir el sistema IXplore y el software scanR.
P: ¿Qué otras funciones les gustaron del sistema?
R: Nos encantó el hecho de que no era un sistema en caja y que gracias al software cellSens tendríamos un óptimo microscopio de campo amplio para escanear portaobjetos. Esta configuración dos en uno del sistema era perfecta para nosotros, ya que nos iba a permitir aumentar la variedad de servicios que ofrecía nuestra instalación.
La modularidad del sistema es bastante adecuada. Gracias a ello, podíamos agregar más funciones si se requería, como incorporar una incubadora destinada al estudio de células vivas.
Otro punto a favor era la posibilidad de incorporar a nuestra cartera una tecnología analítica de imágenes de punta por inteligencia artificial, como la TruAI, la cual no sólo trabaja con las imágenes cellSens y scanR, sino también con las imágenes adquiridas con otros sistemas microscópicos.
P: ¿Qué efectos han podido percibir en la instalación después de haber agregado el sistema IXplore? ¿Algo ha cambiado?
R: El sistema IXplore, combinado con el software scanR, se ha vuelto rápidamente el microscopio más usado en nuestra instalación. El tiempo de uso se multiplica por más de dos frente a los microscopios confocales, que antes eran considerados como los súper de la instalación.
Si bien se trata de un microscopio de epifluorescencia, la resolución de las imágenes es lo suficientemente buena como para evitar que los investigadores cambien a imágenes confocales en algunos estudios, especialmente si se prioriza la alta velocidad del sistema y no tanto la resolución Z. Ahora, ellos pueden capturar imágenes cuya adquisición con nuestros microscopios confocales de escaneo láser llevaba mucho tiempo.
Sin embargo, no sólo se trata de una velocidad más alta, sino también de la posibilidad de usar hasta cuatro soportes para portaobjetos en combinación con el mapa de enfoque, el cual permite configurar experimentos con necesidades de adquisición no supervisadas de noche.
La ventaja de ejecutar análisis de datos sobre la marcha, al mismo tiempo que son adquiridos con el software scanR, es otra estupenda característica en comparación con el sistema HCA anterior. Los resultados analíticos se obtienen en cuanto termina la captura de la imagen, lo cual permite pasar a la siguiente muestra.
La tecnología de aprendizaje profundo TruAI también ha demostrado ser una herramienta muy útil. La detección de núcleos en muestras de cultivos celulares de alta densidad ha mejorado drásticamente en comparación con las técnicas de segmentación estándar. Esto ha permitido a los usuarios obtener datos más precisos a partir de sus experimentos.
P: ¿Podrían darnos un ejemplo de lo que se investiga en su instalación con el sistema IXplore y el software cellSens? ¿Qué desean ver los investigadores en las imágenes?
R: A menudo se adquieren imágenes de alta resolución a partir de secciones transversales u horizontales de la médula espinal de ratones, ratas o incluso cerdos; los investigadores buscan los cambios que se producen en las poblaciones de células gliales, ya sea a nivel de la supervivencia de las neuronas o la expresión de diferentes marcadores después de una lesión de médula espinal (SCI), como también a nivel de los axones que crecen en el área de la lesión.
Sin embargo, no nos limitamos al tejido de la médula espinal. También, se escanean secciones del cerebro a alta resolución. En el siguiente ejemplo, se escaneó una sección cerebral completa de un ratón usando el contraste de interferencia diferencial (DIC). Esto tenía como fin encontrar la ubicación de la expresión genética del gen reportero fluorescente mCherry.
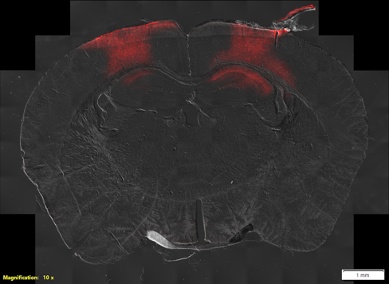
Figura 2. Imagen de portaobjetos completo del cerebro de un ratón usando la DIC y la fluorescencia. Imagen por cortesía del Grupo de Neurofisiología Experimental y Circuitos Neurales del HNP.
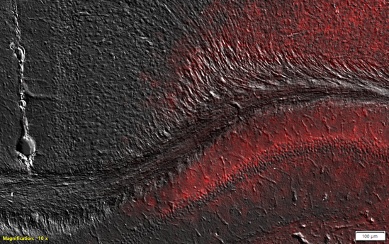
Figura 3. Primer plano de una región en la imagen anterior. Imagen por cortesía del Grupo de Neurofisiología Experimental y Circuitos Neurales del HNP.
P: ¿Pueden proporcionar un ejemplo del uso dado por su instalación al software scanR con fines de HCA? ¿Cuál es el enfoque en los datos?
R: El cribado de alto contenido (HCA) es usado con mucha frecuencia en los estudios de proliferación, diferenciación y migración celular bajo diferentes condiciones. En nuestra instalación, uno de los ensayos más frecuentes que se ejecuta con el software scanR es la detección de la expresión genética de los genes reporteros (GFP y mCherry) en función de diferentes tratamientos. Se combina la TruAI, para la segmentación de núcleos en los cultivos celulares de alta densidad, con el análisis controlado que cuantifica la cantidad de células a partir de un solo o doble marcado en la población; y se evalúa cómo funcionan los diferentes tratamientos.
El sistema también ha sido usado para detectar hibridación fluorescente (FISH) in situ en muestras de sangre y esperma. En este caso, no sólo se usó la TruAI para segmentar núcleos, sino también para detectar puntos de FISH dentro de los núcleos.
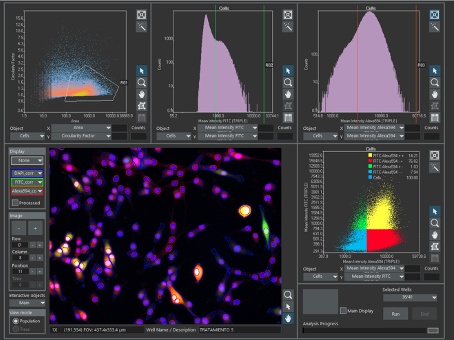
Figura 4. Ventana principal del software scanR que muestra la segmentación de células y núcleos, rodeada de diagramas de dispersión e histogramas. En los diagramas bidimensionales, cada punto representa una célula segmentada. En los histogramas unidimensionales, cada línea representa un valor de parámetro, que hace la correspondencia entre la altura de la línea y la cantidad de células para dicho valor. En el primer diagrama de dispersión, una puerta (R01) selecciona una subpoblación de células según su tamaño y forma, y es transferida a los siguientes diagramas analíticos. Las celdas al exterior de la R01 son descartadas. En el segundo y tercer diagrama analítico, se crean puertas de celdas verdes positivas (R02) y celdas rojas positivas (R03). En el diagrama analítico final, la R01, R02 y R03 son combinadas para obtener el porcentaje de células positivas individuales y dobles. Datos por cortesía del Grupo de Neurología Molecular del HNP.
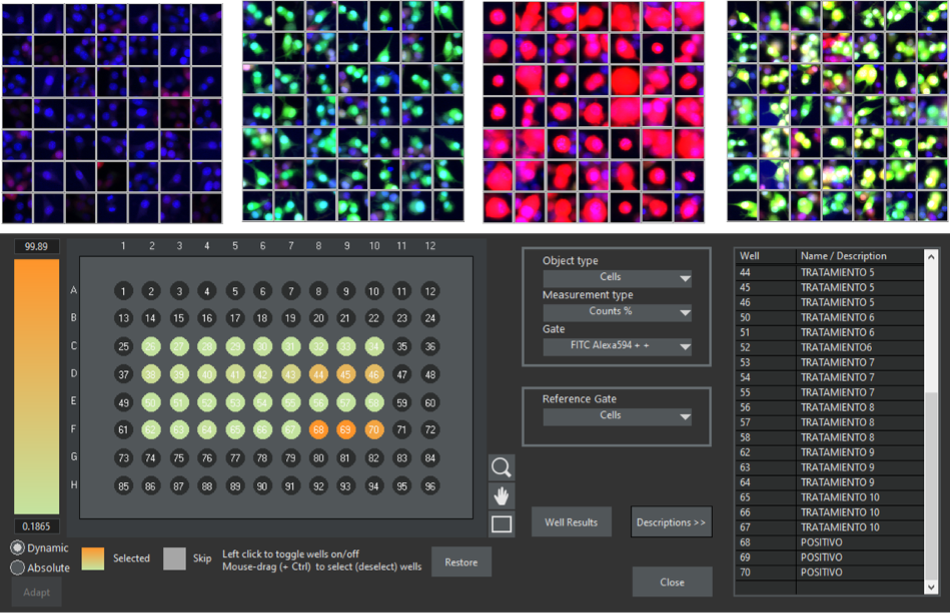
Figura 5. El software scanR permite crear galerías de imágenes a partir de las regiones controladas. Los ejemplos exponen galerías de imágenes de las poblaciones de células: doble negativa, verde individual positiva, rojo individual positiva y verde-rojo doble positiva. Los resultados pueden ser exportados a tablas, o ser visualizados como un mapa térmico en el marco de una placa de pocillos. Datos por cortesía del Grupo de Neurología Molecular del HNP.
P: ¿En su caso, la tecnología TruAI es usada para desarrollar los propios modelos de redes neuronales por aprendizaje profundo?
R: Sí. Disponemos de modelos formados para segmentar núcleos de células densamente empaquetadas, detectar neuronas en las secciones de la médula espinal, clasificar diferentes tipos de fibras musculares, reconocer los núcleos anormales tras determinados tratamientos, etc.
Esta es una de las cosas que más nos gusta del software: la posibilidad de formar fácilmente modelos de redes neuronales, no solo a partir de imágenes adquiridas con este sistema, sino también a partir de las imágenes obtenidas con otros microscopios que no son de Olympus. Y ahora que podemos usar OlyVIA, una aplicación de visualización gratuita que permite hacer anotaciones en las imágenes, esto resulta aún más conveniente, ya que los usuarios pueden trabajar desde sus propios PC en la oficina o incluso en casa.
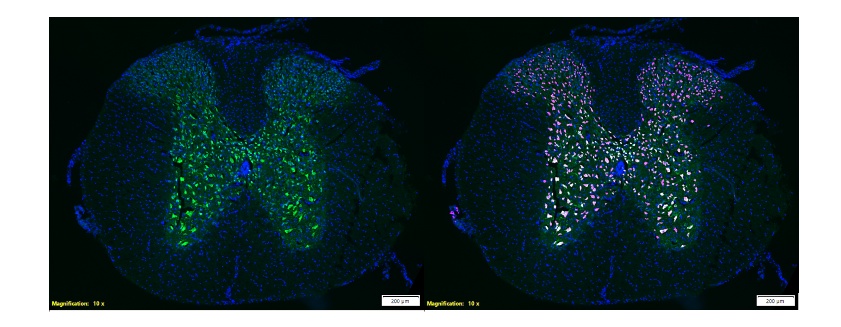
Figura 6. La imagen de la izquierda muestra el marcador neuronal NeuN (verde) de una sección de la médula espinal de un ratón. La imagen de la derecha muestra la misma sección con una capa que muestra la detección TruAI de células positivas NeuN (rosa). Imágenes por cortesía del Grupo de Neurología Molecular del HNP. 66
P: ¿Qué es lo que más les gusta del sistema?
R: Como se ha mencionado anteriormente, hay muchas cosas que nos gustan de este sistema. Tal vez, la más destacable de entre todas ellas, es que al combinar el software cellSens y scanR se puede pasar de escanear todo el portaobjetos al HCA usando un solo sistema microscópico. Esta configuración dos en uno cuadra perfectamente en la optimización de los recursos para nuestra instalación. Y, por supuesto, también está la TruAI que es totalmente compatible con los dos sistemas. Esta permite usar modelos de redes neuronales en ambos softwares, independientemente de cuál haya sido usado para la formación.
Obtenga más información sobre los sistemas microscópicos IXplore
La serie de microscopios IXplore deriva en microscopios invertidos adaptados a diferentes aplicaciones de investigación en las ciencias de la vida. Los microscopios invertidos motorizados IXplore Pro incluyen capacidades de campo claro, fluorescencia multicanal, apilamiento en Z y función mosaico.
El sistema IXplore es controlado con el software cellSens: plataforma dedicada a los microscopios con cámaras de Olympus, adaptables a una amplia variedad de aplicaciones y técnicas de imagen en microscopía de campo amplio. Entre ellas destaca la microscopía de rutina, confocal de disco giratorio, de superresolución, de fluorescencia de reflexión interna total (TIRF), de recuperación de fluorescencia después del fotoblanqueo (FRAP), etc.
Mediante el hardware seleccionado, el mismo microscopio IXplore también puede ser controlado con el software scanR, el cual está dedicado y optimizado para aplicaciones de cribado de alto contenido en placas de pocillos y portaobjetos de cámara.
Personas entrevistadas
Se agradece al Dr. José Ángel Rodríguez Alfaro y al Dr. Javier Mazarío, directores de la instalación de investigación dedicada a la microscopía y el análisis de imágenes del Hospital Nacional de Parapléjicos (Toledo, España), por compartir sus experiencias en esta entrevista. Para cualquier consulta con respecto a esta instalación/centro, envíe un correo electrónico a microscopia.hnp@sescam.jccm.es.

El Dr. José Ángel Rodríguez Alfaro y el Dr. Javier Mazarío, directores de la instalación de investigación dedicada a la microscopía y el análisis de imágenes del Hospital Nacional de Parapléjicos en Toledo (España).
*El sistema scanR no es vendido en Japón.
Contenido relacionado
Segmentación de instancias simplificada de células y núcleos mediante el aprendizaje profundo
Seminario web: Cribado de alto contenido: Facilitar el análisis personalizado

