La technologie d'apprentissage profond TruAI™ est une approche révolutionnaire de l'analyse des images, car elle permet aux chercheurs de s'attaquer à des applications jusqu’à présent impossibles ou difficiles à mettre en œuvre, telles que l'analyse sans marqueur ou le suivi de cellules vivantes avec une très faible luminosité sur plusieurs jours de manière fiable.
Pour ces applications, l'analyse est effectuée par segmentation sémantique, qui permet d'identifier si un pixel appartient à l'arrière-plan ou au premier plan d'une catégorie donnée. Après la segmentation sémantique, des étapes de post-traitement telles que la « ligne de partage » sont appliquées pour effectuer la segmentation finale des objets (généralement des noyaux ou des cellules entières).
Récemment, la technologie d'apprentissage profond TruAI a été améliorée grâce à l'introduction de la segmentation d'instances. Cette fonctionnalité fusionne la segmentation sémantique et la séparation ultérieure des objets en une seule étape. La segmentation d’instances améliore les procédures en vous permettant d'effectuer des opérations difficiles en une seule étape, comme la segmentation des cellules dans les colonies de cellules, qui nécessite des étapes de séparation en post-traitement.
La technologie d'apprentissage profond TruAI permet aux utilisateurs d'entraîner le réseau de neurone avec leurs propres données dès le début, de sorte que les modèles de réseaux neuronaux profonds (DNN) TruAI générés ont des performances extrêmement bonnes. Les chercheurs peuvent développer des bibliothèques de DNN pour différentes applications et même les partager avec leurs collaborateurs.
Mais cela implique que la procédure d'apprentissage profond se compose toujours de trois étapes :
- Marquage avec vos propres données
- Entraînement
- Inférence
Malgré les qualités pratiques de notre logiciel, l'étape de marquage est la tâche la plus longue, en particulier pour les applications difficiles où de nombreux objets doivent être marqués afin d’obtenir de bonnes performances.
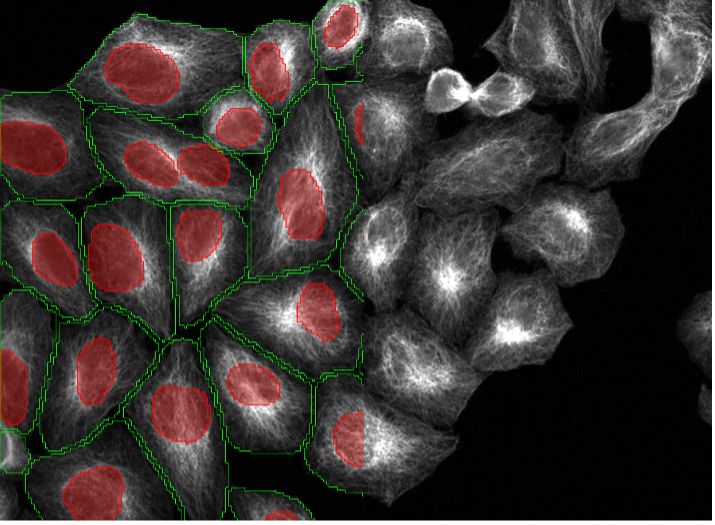
Segmentation simplifiée des cellules et des noyaux à l'aide de modèles d'apprentissage profond pré-entraînés
Pour améliorer cette procédure, la technologie d'apprentissage profond TruAI inclut désormais des modèles pré-entraînés pour les noyaux et les cellules, qui sont les objets les plus fréquement segmentés dans les analyses d’images biologiques. Les utilisateurs peuvent ainsi gagner le temps normalement consacré au marquage et à l’entraînement (figure 1).

Figure 1. L'utilisation de la technologie d'apprentissage profond TruAI avec vos propres données
pour l’entraînement et l'inférence permet d'assurer des performances optimales. Pour une approche
plus rapide, les modèles TruAI pré-entraînés vous permettent d'effectuer l'analyse en un seul clic.
Les modèles TruAI pré-entraînés pour les noyaux et les cellules sont entraînés pour effectuer la segmentation d'instances des images de fluorescence dans diverses situations, et notamment un éventail de :
- Modalités de microscopie (confocale à balayage ponctuel, confocale à disque rotatif, grand champ)
- Marquages flurescents (DAPI, phalloïdine, MitoTracker, lamine, etc.)
- Lignées cellulaires (HeLa, U2OS, SK-HEP-1, cellules végétales, tissus, etc.)
- Contrastes, rapports signal/bruit et niveaux de bruit de fond
- Résolutions de pixel et grossissements
Il est important de noter que les modèles pré-entraînés sont des modèles généraux et ne peuvent pas effectuer la segmentation parfaitement dans toutes les situations possibles. Pour vous aider à mieux comprendre leurs capacités, nous avons dressé une liste de situations de segmentation cellulaire.
Vingt exemples de segmentation de noyaux et de cellules à l'aide de modèles d'apprentissage profond pré-entraînés
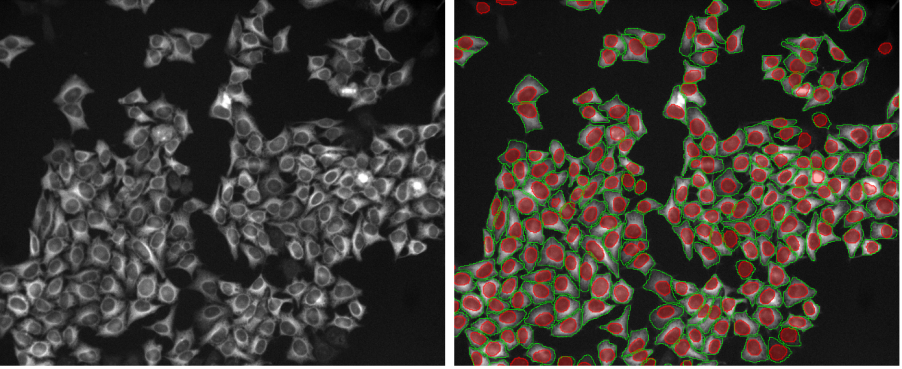
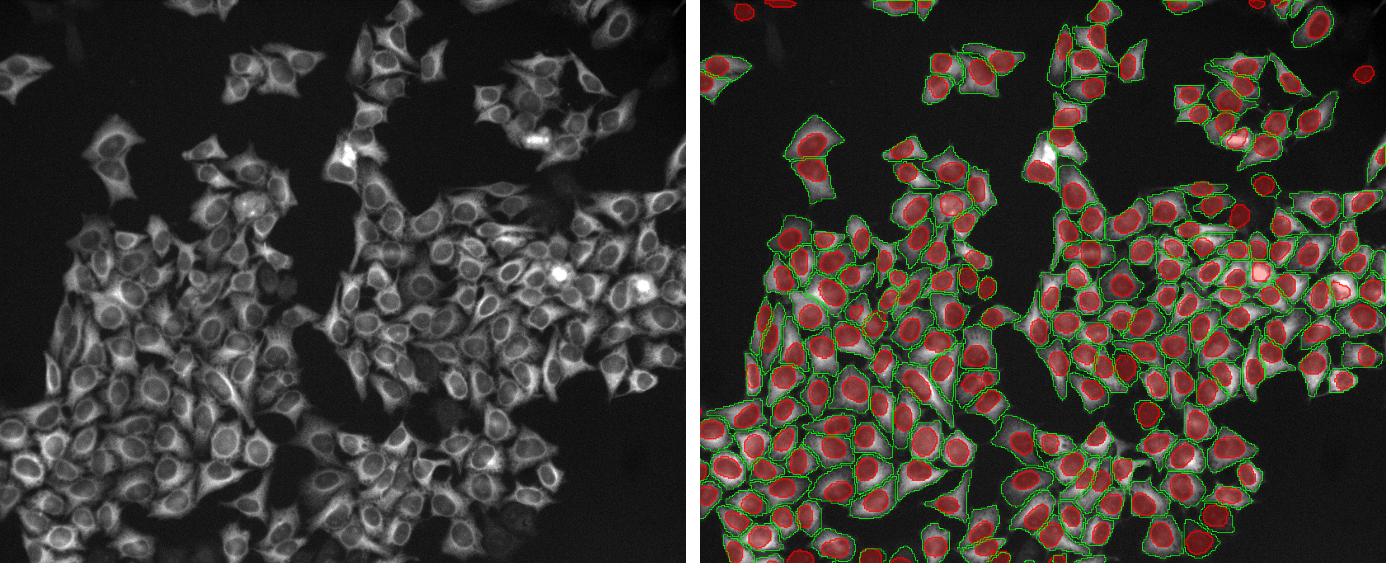
Les exemples d'images ci-dessous (figure 2, a–t) illustrent les performances des modèles TruAI pré-entraînés pour les noyaux (rouge) et les cellules (vert) dans divers scénarios de segmentation. Ceux-ci comprennent divers marqueurs, lignées cellulaires, grossissements, résolutions, contrastes, rapports signal/bruit et hétérogénéités de fond. Notez que les deux modèles peuvent être appliqués à la même image (figure 2, p-t). Les modèles peuvent même être appliqués à des noyaux non spécifiques (figure 2, q–t) ou à des marquages fluorescents de cellules (figure 2p, s).
1. Cellules U2OS, marquage fluorescent nucléaire, grand champ 20x, confluence moyenne avec des cellules de différentes tailles et morphologies
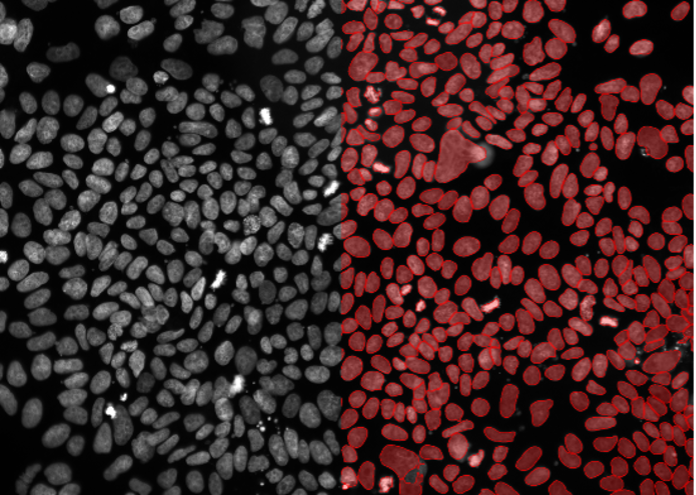
Figure 2a
2. Cellules HeLa, marquage fluorescent nucléaire, grand champ 10x, fond hétérogène
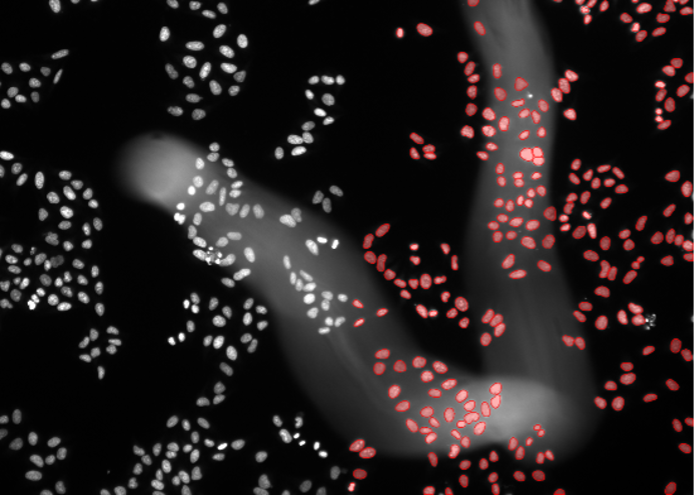
Figure 2b
3. Cellules souches pluripotentes, marqueur nucléaire, grand champ 20x, très forte confluence

Figure 2c
4. Cellules d’organoïdes rénaux, marquage fluorescent nucléaire, disque rotatif confocal 30x, grande profondeur de pénétration
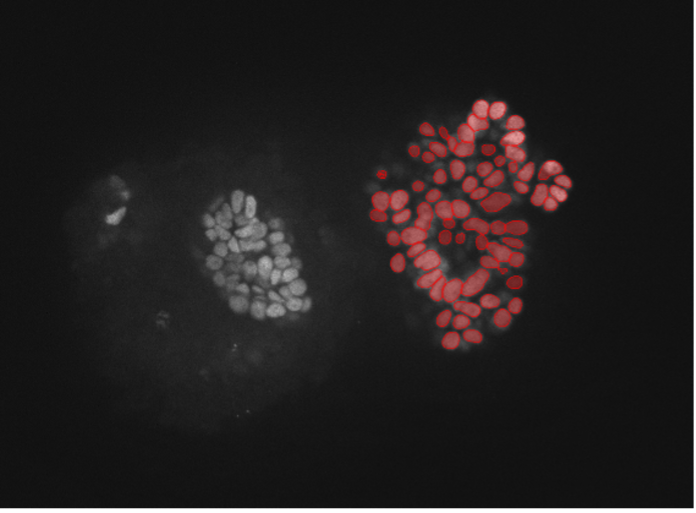
Figure 2d
5. Cellules HeLa, marquage fluorescent nucléaire, disque rotatif confocal 40x, contraste et résolution élevés

Figure 2e
6. Cellules de carcinome humain A549, marqueur nucléaire, disque rotatif confocal 40x, faible rapport signal/bruit
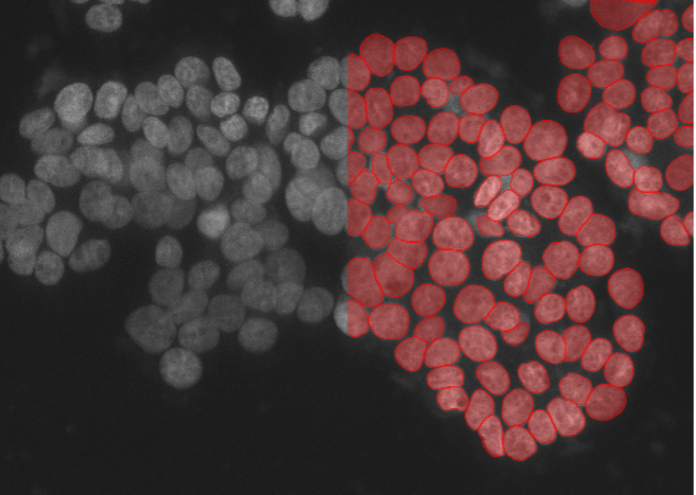
Figure 2f
7. Cellules tumorales de tissu humain, marquage fluorescent nucléaire, grand champ 20x

Figure 2g
8. Cellules de tissus rénaux de souris, marquage fluorescent nucléaire, grand champ 20x, échantillon épais

Figure 2h
9. Cellules Rat-1, marquage fluorescent cytosolique, grand champ 10x, forte confluence cellulaire
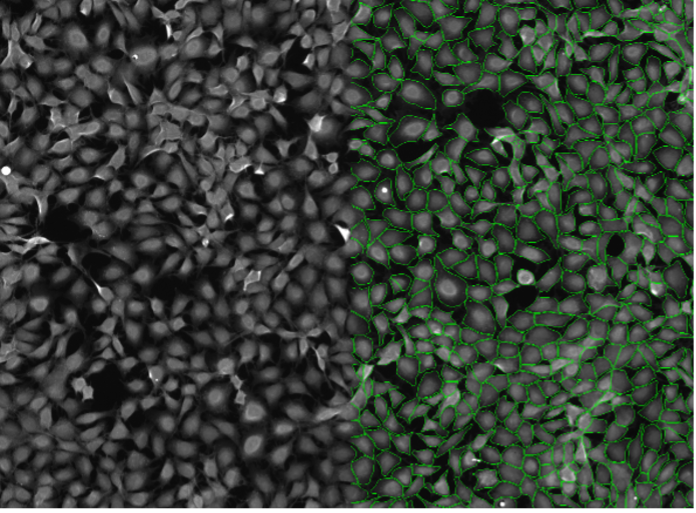
Figure 2i
10. Cellules SK-HEP-1, marquage fluorescent des zones de contact entre cellules, grand champ 20x
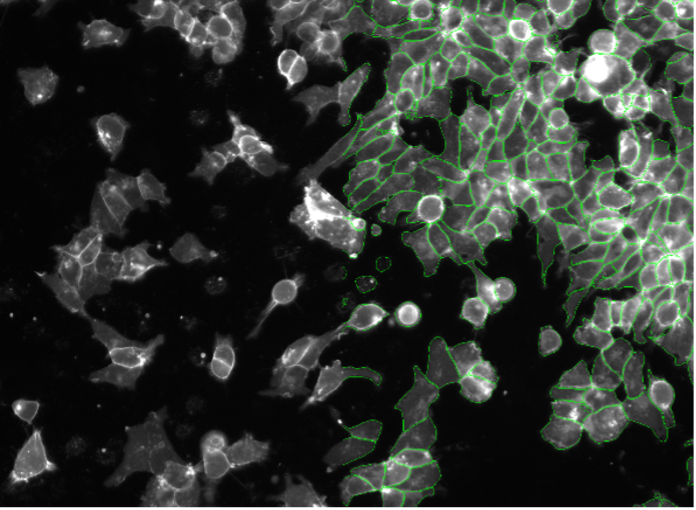
Figure 2j
11. Cellules végétales, autofluorescence, grand champ 20x, faible rapport signal/bruit
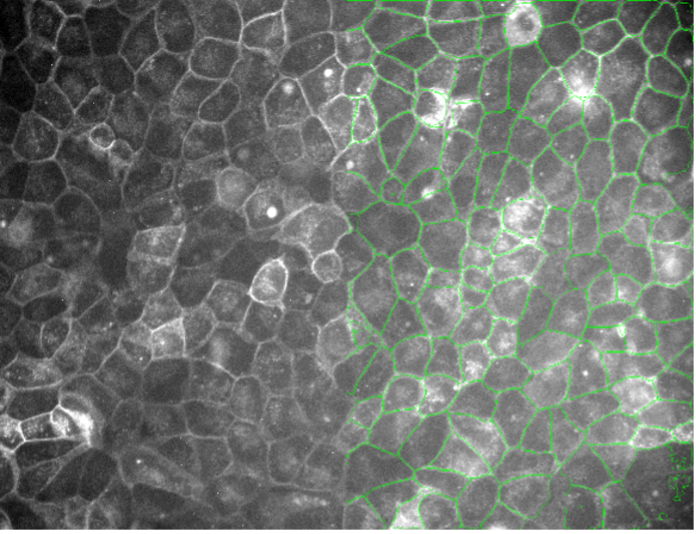
Figure 2k
12. Cellules HeLa, marquage fluorescent de la membrane nucléaire, grand champ 20x
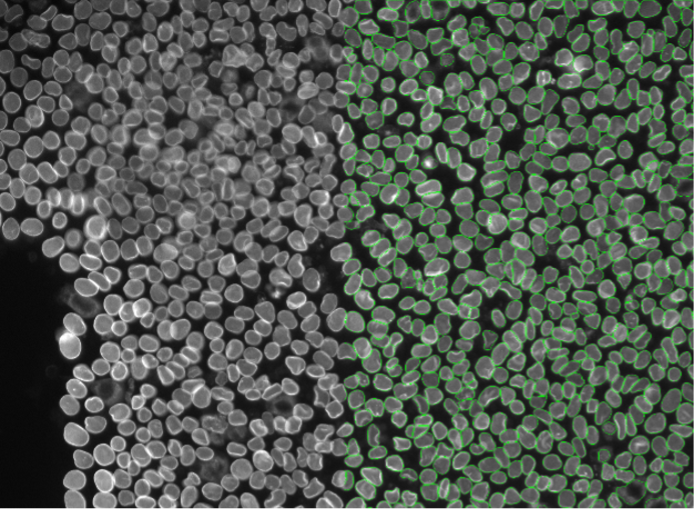
Figure 2l
13. Cellules huFIB, marquage fluorescent de la tubuline, grand champ 20x
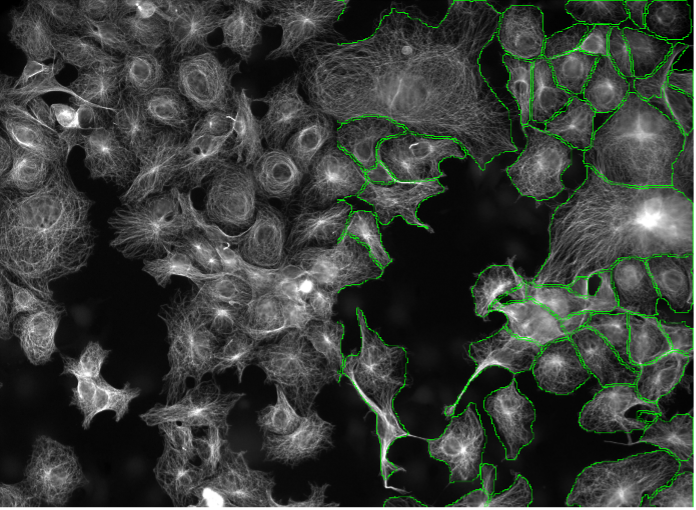
Figure 2m
14. Cellules Rat-1, marquage fluorescent de l'actine, grand champ 20x
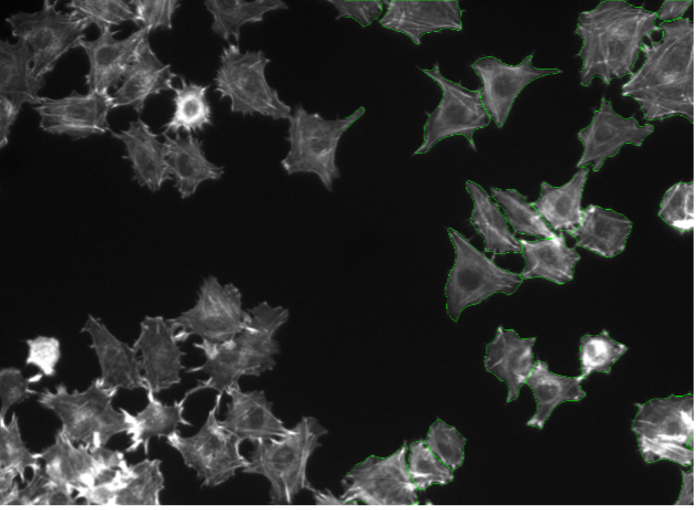
Figure 2n
15. Cellules endothéliales d’artère pulmonaire bovine, marquage fluorescent de l'actine, confocale à balayage ponctuel 40x
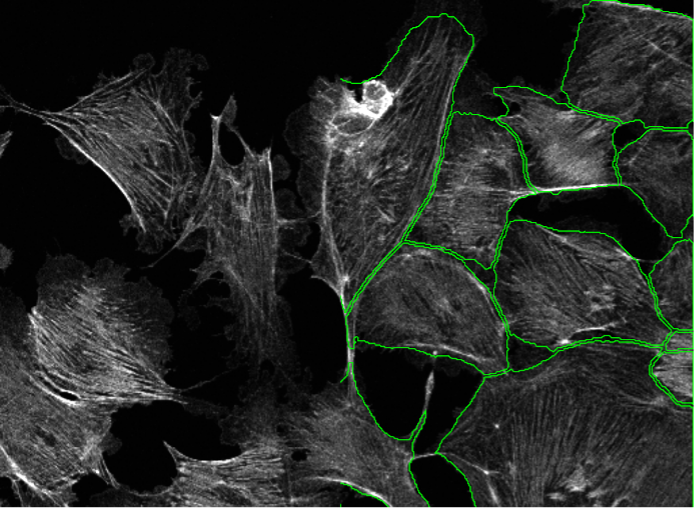
Figure 2o
16. Cellules Rat-1, marquage fluorescent nucléaire diffusant faiblement dans le cytoplasme, grand champ 20x
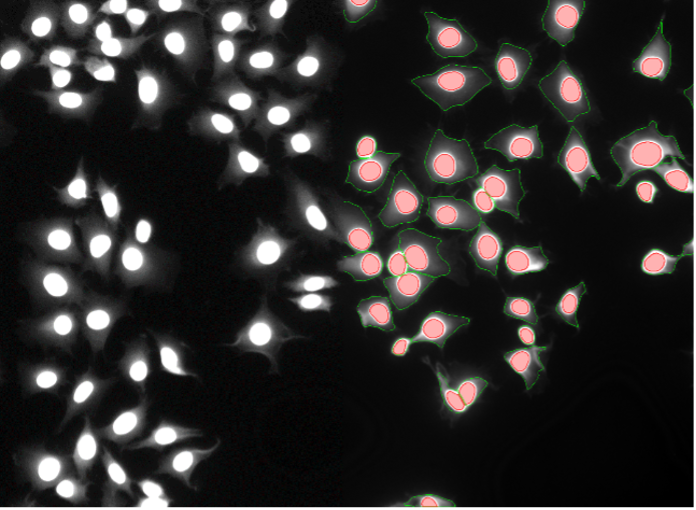
Figure 2p
17. Cellules HeLa, marquage fluorescent de la tubuline, grand champ 4x, faible résolution et faible rapport signal/bruit

Figure 2q
18. Cellules Rat-1, marquage fluorescent cytosolique, grand champ 20x
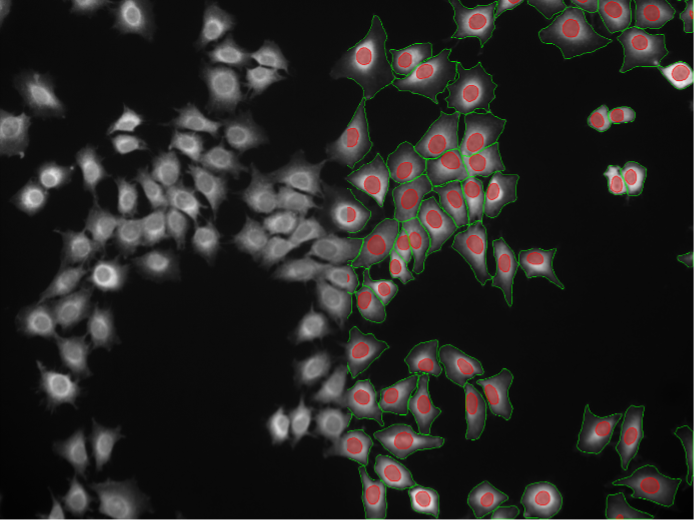
Figure 2r
19. Cellules COS-7, marquage fluorescent des mitochondries, confocale à balayage ponctuel 40x
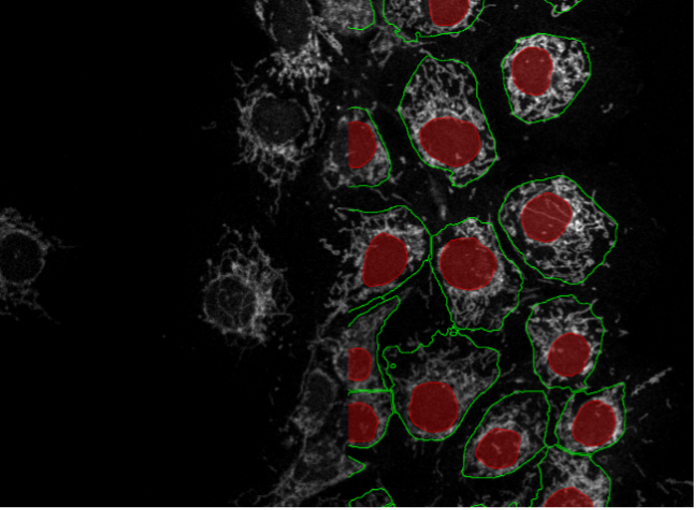
Figure 2s
20. Cellules endothéliales d’artère pulmonaire bovine, marquage fluorescent de la tubuline, balayage confocale à balayage ponctuel 40x
Capacités d'apprentissage profond étendues pour la segmentation des cellules et des noyaux
L'introduction de la segmentation d'instances et de modèles pré-entraînés élargit les capacités de l’outil TruAI. Les utilisateurs peuvent désormais gagner du temps en commençant leurs analyses avec des modèles pré-entraînés et en effectuant la segmentation des cellules et des noyaux en un seul clic. Dans les cas où les résultats ne sont pas optimaux, les utilisateurs peuvent convertir les prédictions TruAI en nouveaux marquages, puis appliquer des corrections. Ces marquages corrigés servent de base à un nouvel entraînement, ce qui permet de gagner du temps et d'économiser des efforts.
Nos clients constatent déjà les avantages des capacités étendues de l’apprentissage profond. Robert Strauss, scientifique chevronné au Danish Cancer Society Research Center, nous a confié : « La reconnaissance pré-entraînée des noyaux est absolument stupéfiante et permet désormais d'analyser facilement des échantillons très hétérogènes sans compromettre la moindre fraction cellulaire. En particulier dans les zones à forte densité cellulaire, la séparation effectuée par la technologie TruAI est nettement supérieure à la détection des intensités lumineuses ou des bords, à la fois en matière de vitesse et de performances. »
Contenu connexe
Webinaire : Améliorez l’analyse de vos images avec la technologie d'apprentissage profond TruAI™