FV3000と高性能対物レンズX Lineを用いたマウス透明脳の高精細広視野イメージング
高精細かつ広視野の3Dイメージング
神経細胞の接続を調べ上げるコネクトーム解析において、大規模のサンプルを対象に神経線維(突起)を正確に追跡する作業が必須となります。以前から、大きな薄い脳切片に対し、高精細な画像をずらしながら取得して連結する2Dタイリングが盛んに行われています。最近は、組織透明化技術の進歩により、分厚い脳サンプルに対して高精細な共焦点画像を異なる深度で何十枚も取得することが可能になり、神経線維のZ軸方向における追跡精度向上が達成されてきました。今まさに、そうした高精細三次元再構築をXYに拡大すべき3Dタイリング機能が活躍すると期待されています。高効率の3Dタイリングに求められるのは、連結に必要な画像周辺部における光学性能であり、特に像面の平坦性(フラットネス)は神経線維を隣り合うタイル間で追跡するために最も重要な性能と言えます。
オリンパスのUPLXAPO-X Lineは、優れた周辺光学性能(フラットネス)を備えた対物レンズ群です。先述の要求に応えるべく、今回は60倍油浸対物レンズUPLXAPO60XOをオリンパス共焦点レーザー走査型顕微鏡FLUOVIEW FV3000に搭載し、蛍光標識された神経細胞を高密度に含むマウス脳の透明サンプルを用いて海馬CA1領域の三次元再構築を行いました。
組織透明化溶液:SCALEVIEW-S
本実験では、理化学研究所の宮脇敦史博士らにより開発されたScaleS法に基づいた組織透明化試薬SCALEVIEW-Sを使用しました。本溶液を用いて生体試料を透明化することで、試料を薄切することなく、蛍光タンパク質で標識された組織内部構造を深部まで高精細に観察することができます。SCALEVIEW-Sは透明化性能が高く、内在する蛍光タンパク質を長期間明るく保つことができるため、脳全体など広範囲にわたるコネクトーム解析に適しています。

図1: 蛍光色素染色:ChemScale プロトコル
高NAの油浸対物レンズUPLXAPO60XO
新型対物レンズシリーズUPLXAPO-X Line*は、従来の対物レンズに比べて、性能が大幅に向上しています。特に平坦性(フラットネス)は優れており、405 nmのような短波長でも視野中心部から周辺部まで均質な画像を取得することができるようになりました。中でも、60倍油浸対物レンズUPLXAPO60XOはNAが1.42と高く、解像度および明るさが格段と向上しています。今回の実験では、マウンティング溶液SCALEVIEW-SMtの屈折率が高いため、対物レンズも屈折率の高い油浸レンズUPLXAPO60XOを採用し観察しました。(*2019年6月発売)
広い視野で撮影、高精細に観察することに成功
今回の実験では、Thy1-YFP-H lineマウスの固定脳スライス(1 mm厚)をサンプルとし、ChemScaleプロトコル(図1)に準じ、DAPI染色および透明化処理を行いました。透明化された脳組織をSCALEVIEW-SMt液中でマウントし、共焦点レーザー走査型顕微鏡FV3000を用いて488 nm励起で神経細胞、および405 nm励起で細胞核の蛍光画像を取得しました。まず、低倍率対物レンズPLAPON1.25Xを用いて組織全体を俯瞰した画像を取得しました(図2 a)。その画像を参考にして、目的の領域(今回は海馬CA1付近)を、高倍率油浸対物レンズUPLXAPO60XOを用いて詳細にイメージングしました(図2 b, c)。さらに、三次元解析ソフトを用いて、取得した画像を三次元再構築しました(動画)。その結果、高精細でかつ深さのある蛍光画像を広い範囲で観察でき、画像境界面においても各神経線維がスムーズに繋がっていることが確認されました。 |
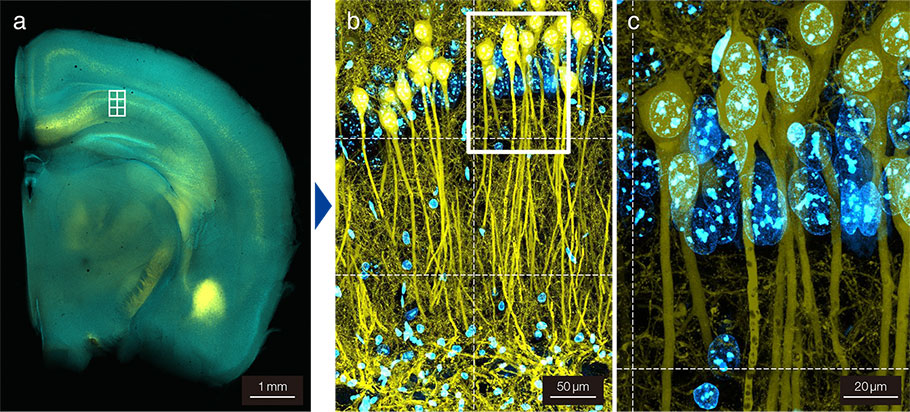
図2:透明化されたマウス脳の共焦点蛍光画像 サンプル条件 撮影条件 | Related Videos動画:海馬CA1領域における神経細胞および細胞核の三次元再構築蛍光画像 |
星田哲志先生からのコメント
高倍レンズを用いて広範囲に3Dイメージングすると、解像度が高く精細に観察できる一方、相対的に画像のズレが目立ちやすくなります。仮に視野周辺部に光学的湾曲等があれば、神経線維の繋ぎ合わせがうまくいかないなどの不具合が生じます。今回はフラットネス性の高い対物レンズUPLXAPO60XOを用いたことで、神経細胞の軸索や樹状突起などがきれいに繋がる良質の画像を描写できました。さらに、収差が激しく出やすい405 nm励起によるDAPIのタイリング画像でもシームレスに連結されており、全体的に歪みのない鮮明な画像が得られました。そのため、画像内にあるほとんどの細胞核のクロマチン構造まで三次元ではっきりと確認できました。
アプリケーションノート制作にご協力賜りました先生:
理化学研究所 脳神経科学研究センター 細胞機能探索技術研究チーム1
光量子工学研究センター 生命光学技術研究チーム2
星田哲志先生1,2 |
濱裕先生1 |
宮脇敦史先生1,2 |
参考文献:
Hama, H., et al. : Nature Neuroscience, 14, 1481(2011).
Hama, H., et al. : Nature Neuroscience, 18, 1518(2015).
Hama, H., et al. : Protocol Exchange (2016), doi:10.1038/protex.2016.019
濱 裕, 他. : 生体の科学, 68(1):pp.85-93 (2017)
日置 寛之, 他. : 日薬理誌, 149(4):pp.173-179 (2017)
日置 寛之, 他. : 生体の科学, 68(5):pp.416-417 (2017)
実験を可能にしたオリンパスの技術
独自の新技術による高性能対物レンズ X Line
マクロ~ミクロ観察
FV3000は、低倍率(1.25倍)から高倍率(150倍)まで、貼り合わせ画像の取得が簡単にできるマクロ-ミクロ機能を搭載しています。1.25倍の対物レンズを使用して組織全体のマクロ画像を撮影し、その上で容易に高解像観察の対象領域を見つけることが可能です。
このアプリケーションノートに関連する製品
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
このページはお住まいの地域ではご覧いただくことはできません。



