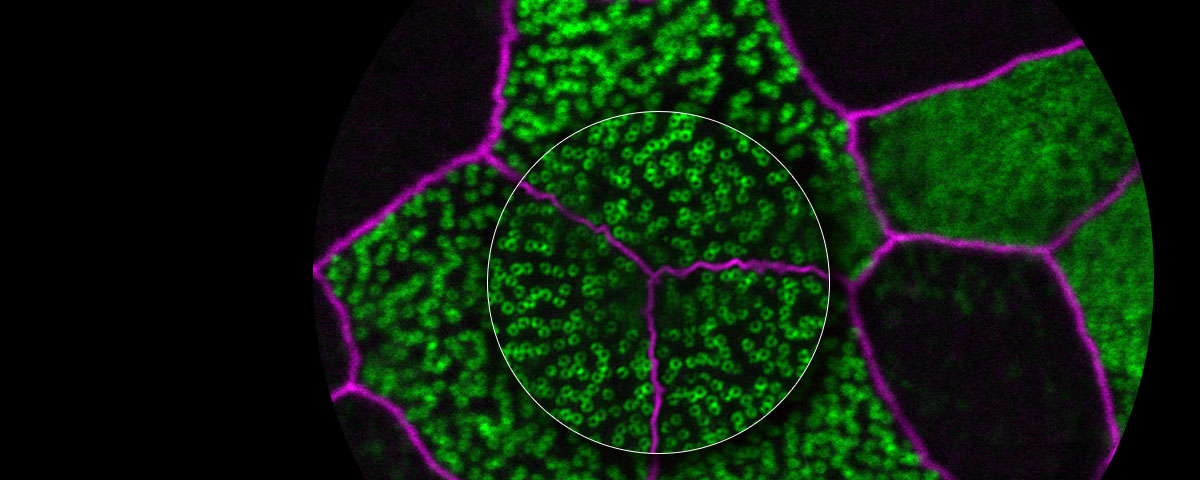
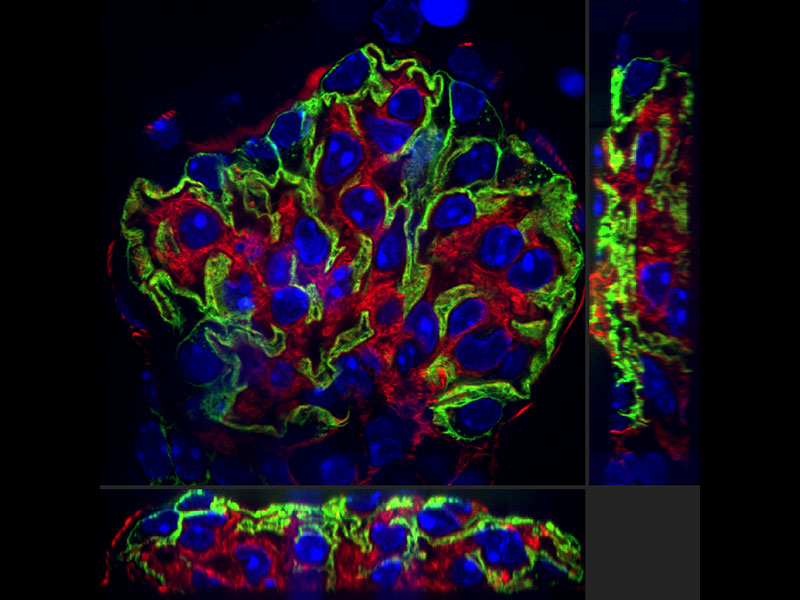
Super resolução para todos os tipos de imagem de célula viva
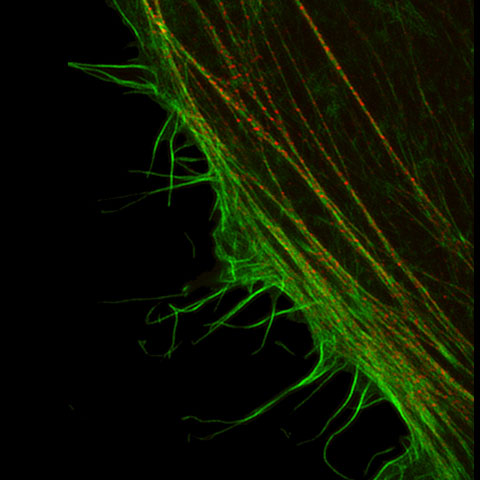
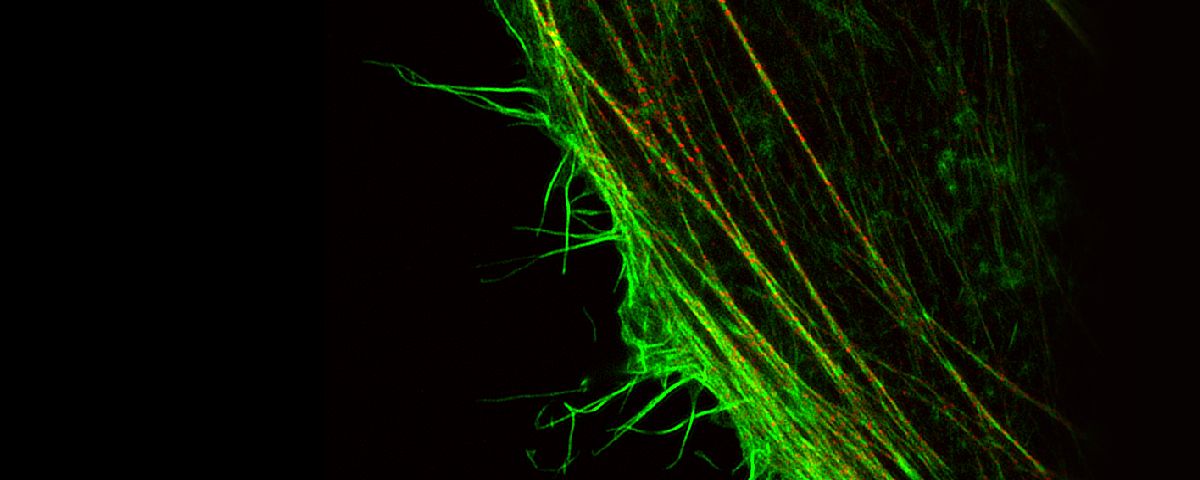
Os microscópios de fluorescência podem identificar proteínas específicas in vivo com sondas de fluorescência. A resolução de muitos desses microscópios é determinada pelo limite da difração de cerca de 200 nm, isto impede a observação de estruturas finas. Com a tecnologia de super resolução da Olympus, pode-se adquirir imagens com resolução abaixo de 120 nm na direção horizontal.
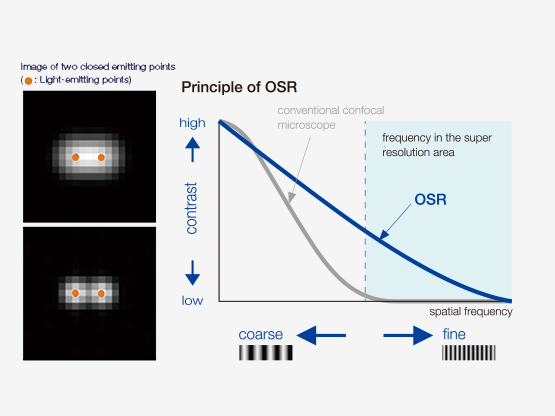
Como o OSR funciona?
| A detecção aperfeiçoada, as configurações específicas de hardware, o diâmetro da abertura confocal otimizado e o processamento avançado de sinal juntos fornecem imagens de super resolução. A tecnologia Olympus Super Resolution (OSR) consegue resolução lateral (XY) abaixo de 120 nm. Referências: |
Olympus Super Resolution (OSR)
SpinSR10
FV3000 (FV-OSR)
|
Outras vantagens
Nosso algoritmo de deconvolução forma imagens de super resolução claras e nítidas. A deconvolução iterativa restrita 3D elimina a distorção no eixo Z e forma imagens tridimensionais nítidas.
|
Depoimentos
Yasushi Okada, M.D. e Ph.D., Centro de Biologia Quantitativa, Riken | O tamanho da maioria dos organelos intracelulares e dos complexos supramoleculares é de aproximadamente 100 nm, com os microscópios convencionais nós não conseguimos observar as estruturas desses complexos. Atualmente, vários métodos de microscopia de super resolução foram desenvolvidos, porém eles não são capazes de realizar experimentos simples na área biológica pois precisam de corantes específicos, de condições precisas de observação e de sistemas óticos especiais. É possível acrescentar facilmente as funções do microscópio confocal com disco giratório no microscópio de super resolução da Olympus. Além disso, a combinação das objetivas de imersão em silicone ajudam a reduzir a aberração esférica e são capazes de formar imagens de super resolução em tempo real de planos profundos das células; eu espero que a tecnologia desta ferramenta possa ser usada na maioria das aplicações na área de biologia. |
Referências
S. Hayashi e Y. Okada, “Ultrafast super resolution fluorescence imaging with spinning disk confocal microscope optics,” Mol. Biol. Cell 26(9), 1743–1751 (2015).
S. Hayashi, “Resolution doubling using confocal microscopy via analogy with structured illumination microscopy,” Jpn. J. Appl. Phys. 55(8), 082501 (2016).
A. Nagasawa-Masuda e K. Terai, “Yap/Taz transcriptional activity is essential for vascular regression via Ctgf expression and actin polymerization,” PLoS ONE 12(4), e0174633 (2017).
H. Nakajima, et al., “Flow-Dependent Endothelial YAP Regulation Contributes to Vessel Maintenance,” Dev. Cell 40(6), 523-536.e6 (2017).
K. Tateishi, et al., “Three-dimensional Organization of Layered Apical Cytoskeletal Networks Associated with Mouse Airway Tissue Development,” Sci. Rep. 7, 43783 (2017).
E. Herawati, et al., “Multiciliated cell basal bodies align in stereotypical patterns coordinated by the apical cytoskeleton,” J. Cell Biol. 214(5) 571-586 (2016).
M.-T. Ke, et al., “Super-Resolution Mapping of Neuronal Circuitry With an Index-Optimized Clearing Agent,” Cell Rep. 14(11) 2718–2732 (2016).
Sorry, this page is not
available in your country.
Sorry, this page is not
available in your country.
.jpg?rev=2087)