使用 scanR 系统的 TruAI™ 深度学习技术监控干细胞分化过程中的细胞周期动力学
引言
在哺乳动物的早期发育过程中,胚胎干细胞 (ESC) 经历细胞分化并产生所有胚胎生殖层,这些层最终在成年生物体中分化为多种细胞类型。 ESC 的特点是具有难以置信的自我更新和增殖能力,由于间隙期(G1 和 G2)截短,因此其分裂周期较短。 在细胞分化过程中,会发生标志性的细胞事件: 细胞改变形态,生长至约为其原有大小的十倍,并改变细胞核与细胞质的比率,同时细胞变得扁平和拉长; 命运特异性基因表达程序被激活,并发生全局染色质修饰。 重要的是,细胞周期减慢。 分化的体细胞具有较长的G1期和G2期、严格的检查点控制和井然有序的分裂周期(Padgett 和 Santos,2020 年)(图 1)。
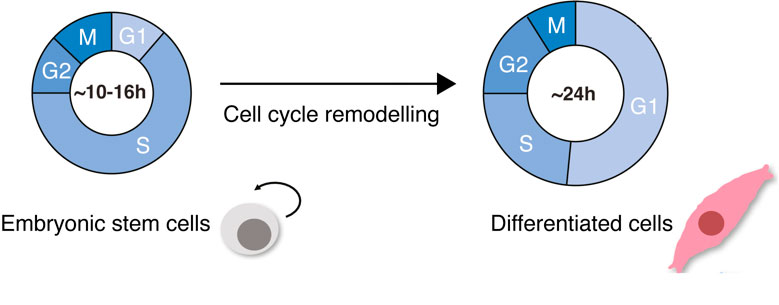
细胞周期重建不仅仅是 ESC 分化过程中发生的一种现象。 细胞周期动力学的变化在生物学领域经常发生。 例如,当细胞在受到病毒感染后或出现恶性肿瘤期间经历再生时。 因此,研究细胞分化背景下的细胞周期动力学变化对于理解细胞周期控制的潜在保守机制非常重要,并将对我们理解健康和疾病状态之间的平衡产生影响。
通过测量参与细胞周期不同阶段的基因编码蛋白的活性,可以监控分化过程中单个细胞发生的变化(Araujo 等人, 2016 年; Sakaue-Sawano 等人, 2008 年)。 其中一种探针是基于荧光泛素化的细胞周期指示剂 FUCCI (CA),由 Miyawaki 及其同事开发(Sakaue-Sawano 等人, 2017 年),它监控 Cdt1 和联会蛋白(两种重要的细胞周期蛋白)的降解,产生 G1、S 和 G2 期(红色、绿色和黄色)的清晰三色分离,可在显微镜的 YFP 和 mCherry 通道中观察到(图 2)。
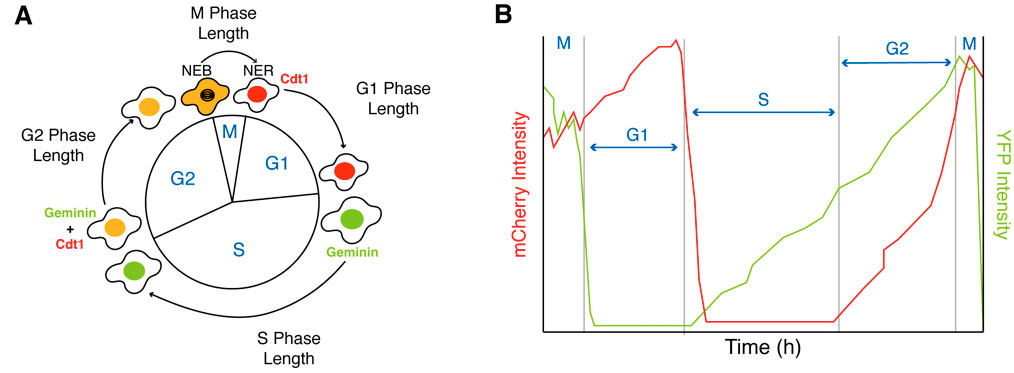
图 2. 使用 FUCCI(CA) 探针测量细胞周期的 G1、S、G2 和 M 期。 (A) FUCCI(CA) 探针示意图。 (B) 基于 Cdt1(mCherry 通道)和联会蛋白(YFP 通道)表达水平的 G1、S、G2 和 M 期持续追踪的示例。
然而,在细胞分化过程中监控细胞周期动力学在成像和数据分析两方面都面临挑战。 长时间活细胞成像需要稳定的温度和二氧化碳控制,通过柔和的光照尽量降低光毒性,并通过可靠的自动聚焦确保在整个时间序列采集过程中获得优质图像。
对分析而言,最大的挑战是在于 ESC分化过程中运动、分裂并显著改变其形状的同时在低荧光强度和高融合条件下对单个细胞进行分割和后续追踪。
所有这些难题都可以通过配备 cellVivo 培养箱、用于自动聚焦的 Z 轴防漂移补偿 (ZDC) 硬件、用于分割的 TruAI™ 深度学习技术以及用于追踪的动力学模块的高内涵scanR 荧光显微镜来解决。
物镜
在本应用说明中,我们会向您展示,运用 TruAI 技术,可以通过FUCCI(CA) 探针的长时间成像,显著改善对单个 ESC 的探测和追踪。 这对于追踪没有标记细胞核的细胞效果尤其显著,可以避免不必要的照明和光毒性。
为了实现这一目标,我们利用 TruAI 技术生成了一个深层神经网络 (DNN) 模型,该模型能够在所有时间点准确地识别多能 ESC 和分化细胞的位置,即使是在荧光强度很弱的情况下。 使用这个技术可以长期对细胞群的 G1、S 和 G2 期进行可靠的监控。 随后,通过应用 scanR 动力学模块,我们可以追踪数以千计的细胞,评估每个细胞的运动轨迹,并获得单细胞水平在细胞周期变化方面的定量动态信息。
为了展示scanR 系统作为研究工具的多功能性,所有数据的展示(包括直方图、散点图、图库、踪迹和动力学轨迹)仅使用 scanR 软件就可完成。
实验设置
用 50 ng/ml 的骨形态发生蛋白 4 (BMP4) 处理表达 FUCCI(CA) 探针的 H1 人类胚胎干细胞 (hESC),以促进其向中内胚层谱系分化。 然后使用10 X UPLANSAPO 物镜的 scanR 显微镜在 mCherry 和 YFP 通道中每隔 15 分钟对这些 H1 细胞进行一次成像,一共持续 6 天。
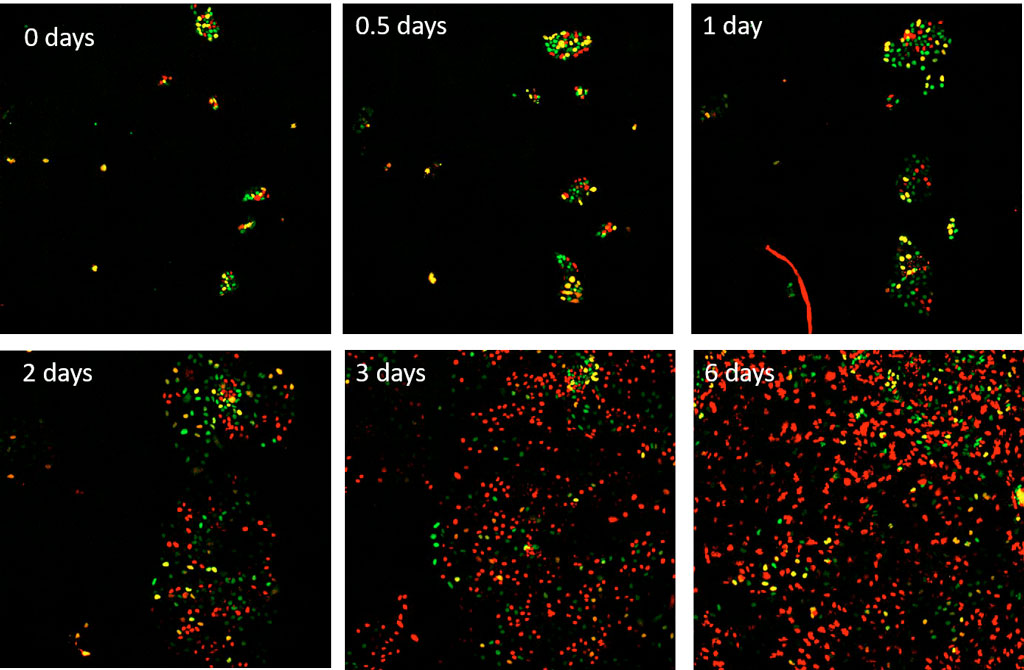
图 3. 在成像之初,先用 BMP4 刺激已经表达 FUCCI(CA) 探针的 hESC H1 细胞。 红色、绿色和黄色分别对应于 G1、S 和 G2 期。
开发一个用于探测活细胞成像条件的 DNN模型
培养数日后,表达 FUCCI(CA) 探针的单个细胞可能会表达出明显的荧光强度变化。 具体地讲,在从 G1 向 S 期过渡时,总的荧光强度可能非常低(图 2b)。 这可能会导致追踪算法失效,因为会在难以检测到细胞的时间点中断追踪。
为了实现对尽可能多的细胞进行数日追踪,需要开发一种能够在非常弱的强度下检测细胞的 DNN 模型。 在之前的白皮书中,我们证明了,通过使用成对的荧光细胞长曝光和强照明图像与短曝光和弱照明图像进行对比,可以实现这一目标(Woerdemann,2020 年)。 简要地讲,长曝光图像用于细胞分割并生成虚拟图层(mask)。 该虚拟图层被用作在真实数据中训练 DNN 模型,使其能够检测低曝光水平下的细胞。
或者,也可以执行手动标注来训练 DNN 模型。 要完成这项任务,可以使用奥林巴斯的 cellSens™ 软件。 cellSens 软件生成的 DNN 模型可以导入 scanR 软件。 在本应用说明中,我们使用了后一种手动方法,并在不同时间点提供了数百条不同强度的细胞注释,以训练 DNN 模型。
分割: TruAI 深度学习技术与传统方法的比较
一旦生成DNN模型,DNN 模型将应用于单个 mCherry 和 YFP 荧光通道。 这会为每个通道创建一个像素 AI 概率图。 像素中 AI 概率越高,该像素包含细胞的可信度就越高。 随后,对两个通道的 AI 概率图进行求和,并通过对该求和图层使用强度阈值来执行分割(图 4e 和 4f)。
为了将此结果与传统方法进行比较,使用滚球算法对 mCherry 和 YFP 荧光通道进行了背景校正、求和,并使用强度阈值(图 4c)或边缘探测方法(图 4d)对求和结果进行了分割。 上述所有图像处理步骤均在 scanR 分析软件上进行。
scanR 系统的统计工具用于创建表 1,其结果是指 6 天时间内单一视场的结果。
表 1. 每种方法分割的细胞数量比较
| 时间点 | 分割方法 | ||
|---|---|---|---|
| 强度阈值 | 边缘探测 | TruAI 技术 | |
| 0 小时 | 78 | 103 | 93 |
| 24 小时 | 209 | 188 | 240 |
| 48 小时 | 421 | 281 | 475 |
| 72 小时 | 847 | 654 | 986 |
| 96 小时 | 1480 | 1330 | 1590 |
| 160 小时 | 1340 | 1460 | 1610 |
|
所有帧的总和
0–160 小时 | 504131 | 573179 | 659199 |
荧光图像与 AI 概率图像的简单比较(图 4a 和 4b)表明,TruAI 方法在检测所有类型的细胞时都具有更高的灵敏度。 表 1 中汇总的结果以及整个时间段都证明了这一点。 传统强度阈值法无法检测弱荧光细胞及确定其正确形状(图 4c)。 边缘探测方法改善了检测到的细胞的轮廓,但仍会遗漏许多暗淡的细胞(图 4d)。 TruAI 深度学习技术能够可靠地探测暗淡的细胞,并且它们的边界十分清晰(图 4e 和 4f)。
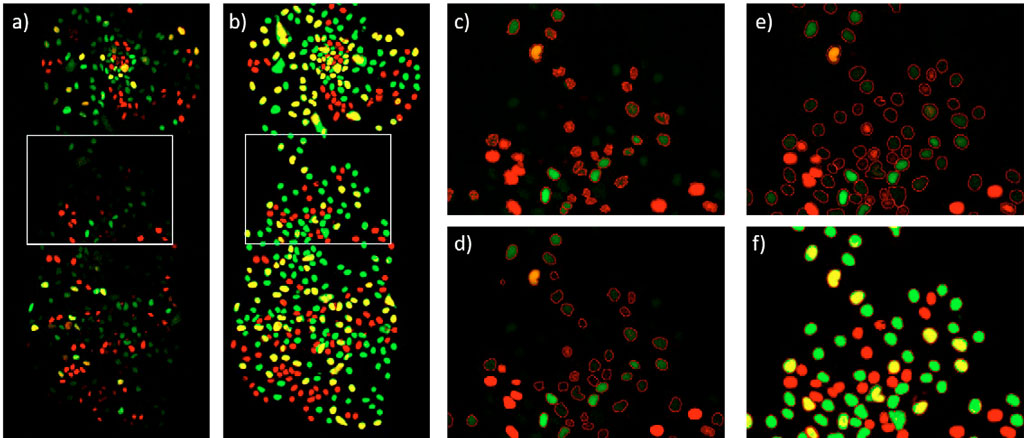
图 4: 48 小时的 ESC 细胞群。a) mCherry(红色,G1 期)和 YFP(绿色,S 期)荧光通道。 G2 期被视为 mCherry 和 YFP(黄色)的组合。b) mCherry(红色)和 YFP(绿色)通道中的 TruAI 概率。 两个通道中 AI 概率高的细胞显示为黄色。c) 在 mCherry 和 YFP 荧光通道总和中使用强度阈值进行分割 d) 在 mCherry 和 YFP 荧光通道总和中使用边缘探测法进行分割 e) 在 TruAI 概率强度总和中进行分割,结果与荧光图像叠加 f) 和 e) 基本相同,不同的是其结果与TruAI 概率叠加。
长时间细胞群分析
散点图(图 5)显示的是使用 TruAI 技术分割的细胞,在mCherry 和 YFP通道中不同时间间隔时的荧光强度变化。 仅包含 mCherry的细胞对应于G1期,仅包含YFP的细胞对应于S期,包含双通道荧光强度的细胞对应于G2 期。 图 5 显示大多数细胞在早期处于 S 期,后期大多数细胞处于 G1 期。 这一细胞群的变化分别与多能细胞和分化细胞所预期的细胞周期构成是一致(图 1)。
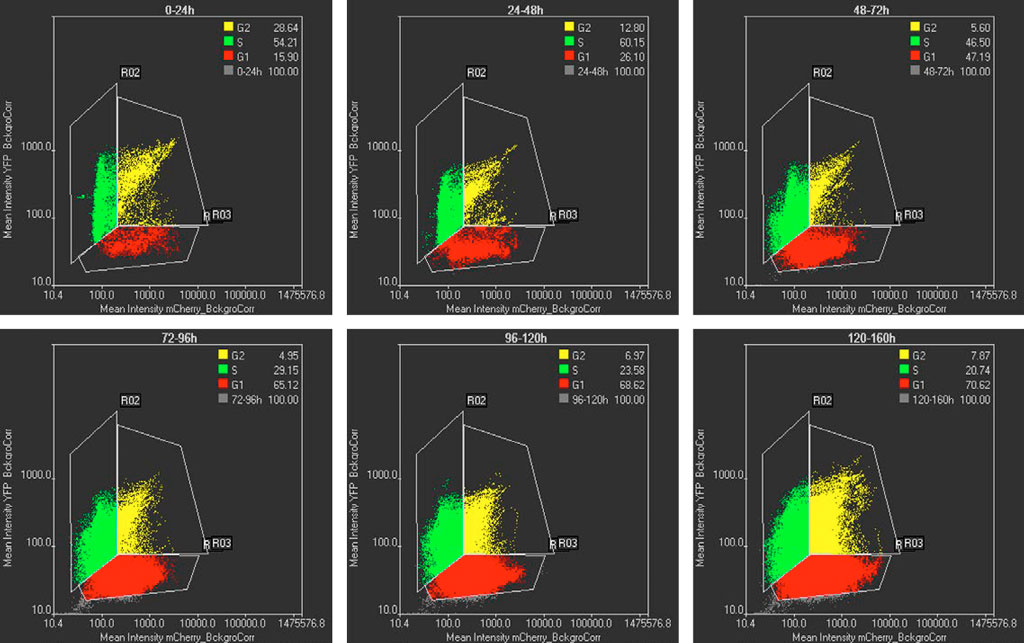
图 5: 不同时间间隔下 mCherry 与 YFP 强度对比的散点图。 散点图中的每个点表示用 TruAI 技术分割的单个细胞。 G1、S 和 G2 细胞分别用红色、绿色和黄色表示。 插入部分显示了 G1-S-G2 细胞群的百分比。 时间间隔显示在每个图形的顶部。
细胞追踪: TruAI 技术与传统方法的比较
利用 scanR 动力学模块,在相同的追踪设置下对用三种方法(强度阈值、边缘探测和 TruAI 技术)分割的细胞进行了追踪。 scanR 系统的统计工具用于创建表 2,其结果是指 6 天时间内单一视场的结果。
表 2. 每种方法追踪的细胞数量比较
| 时间点 | 追踪的细胞数 | ||
|---|---|---|---|
| 强度阈值 | 边缘探测 | TruAI 技术 | |
| 24 小时或更长 | 302 | 1030 | 1965 |
| 48 小时或更长 | 16 | 126 | 426 |
| 72 小时或更长 | 0 | 27 | 72 |
| 96 小时或更长 | 0 | 0 | 5 |
表中显示,使用 TruAI 技术进行分割明显改善了后续的追踪结果。 对于该数据集,考虑到 24 小时每个视场的平均细胞数为 200(表 1),可对 70 多个细胞进行 3 天的追踪,甚至可以进行 4 天或更长时间的追踪。 能够在如此长的时间内追踪如此多的细胞很重要,因为这样就可以通过具有统计学意义的方式进行多次细胞分裂过程中的细胞周期动力学的研究。
细胞轨迹分析
对于 scanR 系统生成的每个轨迹,都可以提取多种参数。
- 要确定最长的轨迹,可以在直方图中选择“寿命”参数,并且可以为超过 24 小时的轨迹创建门(图 6a)。
- 要进一步过滤轨迹,可在散点图中显示参数“初始(时间)”和“最终(时间)”(图 6b)。 这有助于我们确定只跟踪前几天的细胞(周期时间短的 ESC 细胞)、仅追踪最后几天的细胞(细胞周期时间长的分化细胞)以及追踪整个时间段内的细胞(分化过程中受到监控的细胞)。 确定这些轨迹后,就可以绘制 mCherry 和 YFP 的强度随时间变化的曲线,以获得有关细胞周期动力学的定量信息(图 6c、d、e)。
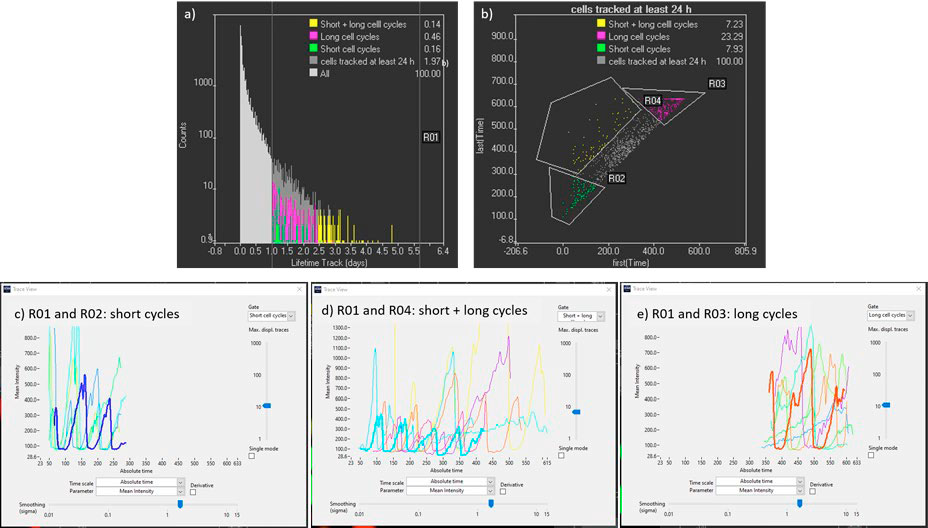
图 6. a) 用于过滤长轨迹的轨迹寿命直方图 (R01)。b) 带有轨迹初始和最终时间点的散点图,用于确定从开始到结束过程中追踪的细胞(R01 和 R04)。c)、d) 和 e) 追踪时间超过 24 小时的 mCherry 细胞的动力学轨迹,分别包含早期起点和终点、早期起点和晚期终点以及晚期起点和终点。 时间间隔为 15 分钟。
从图 6c 到 6e,可以观察到 mCherry(G1 期)信号持续时间随时间增加,从 ESC 细胞的约 2 小时增加到细胞分化后的 24 小时。 对于细胞周期动力学的深入分析,可以选择区域 R01 和 R04 的轨迹,其中 从ESC 向分化细胞的转化是通过四次或更多次细胞分裂完成的(图 6d)。
图 7 显示了追踪一个超过五个细胞周期而分化的 ESC 细胞。 在图 7a 中,ESC的位置显示在时间点 52(13 小时)处,同时显示了其至最大时间点 422(105 小时)的轨迹。 在图 7b 中,显示了时间点 280(70 小时)的图像。 请注意,这个细胞的荧光强度极弱,使用传统方法检测不到。 在图 7c 中,显示了相同的图像,但使用了 TruAI 概率图像。 被追踪细胞的概率强度与其它细胞的概率强度一样高。 图 7d 和 7e 分别代表被追踪细胞每 间隔15 分钟的荧光和 TruAI 概率图像,以画廊的形式体现。 请注意,直至G1 期(红色)结束时,红色荧光强度始终非常弱。 图 7f 表示 mCherry(顶部)和 YFP(底部)荧光强度变化,并从中可以提取 G1、S、G2 和 M 期对应的细胞周期。
从这些数据中,我们观察到,随着时间的推移,该特定细胞在 G1 期的时间增加,在 S 和 G2 期的时间减少。 M 期总是很短; 其确切位置看似 mCherry 信号的减弱,同时表现为 YFP 信号的一个剧烈衰减。
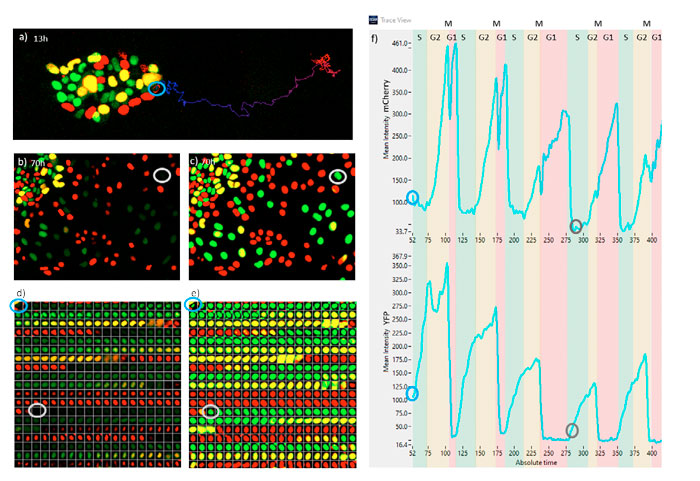
图 7. a) 13 小时的荧光图像。b) 70 小时的荧光图像。c) 70 小时的 TruAI 图像。d) 被追踪细胞每隔 15 分钟的时间序列荧光图像。e) 被追踪细胞每隔 15 分钟的时间序列TruAI图像。f)不同细胞周期时段的 mCherry 和 YFP 荧光信号轨迹。 时间间隔为 15 分钟。 a–f 中的蓝色和灰色标注分别对应同一细胞在 13 小时和70 小时的图像。
结语
这种新颖的深度学习方法,与 scanR 高内涵筛选系统的动力学模块相结合,分析ESC 分化过程中细胞周期动力学的变化,能够采集到有关单个细胞在分化过程中细胞周期动力学变化情况的可重现并具有统计学意义的定量数据。 在数天的分化过程中,数千个形状和形态上发生变化的细胞可以通过测量荧光强度的方式得以识别和追踪。 这将成为研究细胞周期动力学的强大工具,主要用于研究早期发育和重编程过程中细胞周期动力学的研究,以及正在经历细胞周期调控发挥中心作用的细胞转化(恶性肿瘤)的研究。
参考文献
Padgett, J., and Santos, S.D.M. 2020. “From clocks to dominoes: lessons on cell cycle remodeling from embryonic stem cells.” FEBS Letters. 10.1002/1873-3468.13862.
Araujo, A.R., Gelens, L., Sheriff, R.S.M., and Santos, S.D.M. 2016. “Positive feedback keeps duration of mitosis temporally insulated from upstream cell cycle events.” Molecular Cell 64, 362–375.
Sakaue-Sawano et al. 2008. “Visualizing Spatiotemporal Dynamics of Multicellular Cell-Cycle Progression.” Cell 132, 487–498.
Sakaue-Sawano et al. 2017. “Genetically Encoded Tools for Optical Dissection of the Mammalian Cell Cycle.” Molecular Cell 68, 626-640.
Woerdemann, M., and Genenger, M. 2020. “TruAI™ Technology with Deep Learning for Quantitative Analysis of Fluorescent Cells with Ultra-Low Light Exposure.” 奥林巴斯应用说明。 www.olympus-lifescience.com/en/resources/white-papers/ultra-low_light_exposure_analysis/
作者
Joe Pagdett 和 Silvia Santos
Quantitative Stem Cell Biology Lab, The Francis Crick Institute, 1 Midland Road, NW1 1AT, London, UK
Manoel Veiga Gutierrez
Olympus Soft Imaging Solutions GmbH, Johann-Krane-Weg 39, 48149 Muenster, Germany
适于这类应用的产品
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
对不起,此内容在您的国家不适用。