基于深度学习技术的TruAI用于微孔板的鲁棒性、无标记核检测和分割
引言
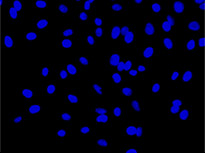
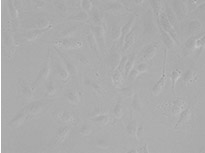
用彩色标记物标记细胞,尤其是使用荧光染料标记细胞,对于观察和分析生物学特征和过程非常有用。在这些标记技术发展之前,都是通过无标记方式对生物样品进行显微镜观察分析。由于机器学习方法在图像分析中获得巨大发展,近年来无标记观察的重要性又得到大幅度提升。基于深度学习的TruAI对透射光图像分析提供新的途径,因此有可能取代荧光标记方法进行细胞和细胞器的结构分析*1 (图1)。

图1
从左到右:TruAI预测的核位置(蓝色),绿色荧光蛋白(GFP)标记核的组蛋白2B(绿色)和原始明场透射图像(灰色)。
任何无标记方法都不能完全替代荧光,这是因为直接将标记物附着到目标分子上所获得的信息仍然具有价值。但是,以较少标记获得相关样品信息存在着明显的优势:
- 降低样品制备的复杂性
- 减少光毒性
- 为其他标记物保留荧光通道
- 成像更快
- 通过避免转染或化学标记提高活细胞的活力
无标记分析的局限性很大程度上在于缺乏有效解决透射光图像分析固有问题的方法。这些限制包括:
- 低对比度,尤其是明场图像
- 与荧光显微镜相比,光路上的灰尘和其他缺陷会对图像质量产生负面影响
- 改进技术条件提高对比度,如相差或微分干涉(DIC)
- 与荧光相比,背景水平更高
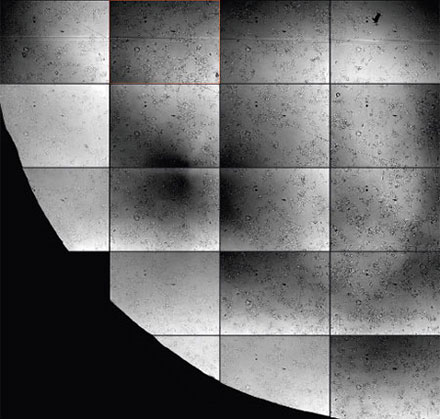
对于微孔板中的活细胞成像,由于缓冲液的表面弯曲和其他噪声影响,要想获得具备足够质量的透射图像进行分析可能尤其具有难度(图2*2和3)。微孔板的难题包括:
- 相差通常不可能
- DIC仅可用于玻璃培养皿
- 明场图像在孔边界处存在明显阴影
- 冷凝物的影响,可能需要取下盖子
- 悬浮液中的颗粒物会提高背景
|
|
图2*2 | 图3 |
TruAI背后的技术
透射明场成像是无标记分析应用的自然方法,但是其也造成长期以来尚未解决的图像分析和分割难题。为了应对这些挑战,奥林巴斯将图像分析方法TruAI集成到cellSens软件和scanR中,TruAI是基于深度卷积神经网络系统工作的。这种神经网络架构近期已被视为最强大的对象分割技术(Long等人,2014: 用于语义分割的完全卷积网络)。这种神经网络系统对于各种挑战性图像分析任务具有非常好的适用性,它将成为透射明场图像进行无标记分析的最佳选择。
在训练阶段,TruAI自动学习如何预测诸如细胞或细胞器位置和曲线等相应参数。在训练阶段,向网络发送图像和“真实”数据(即,对感兴趣的对象进行目标注释)。一旦对网络进行训练之后,就可以将其应用于新图像并以高精度预测目标物。
通常,在机器学习中,正确的目标物(如,细胞边界)由人类专家提供。这个步骤可能非常乏味并且耗费时间,这是因为神经网络需要大量训练数据才能充分发挥其潜力(图4)。因此,拥有方便且易于使用的标注正确目标物的工具至关重要。
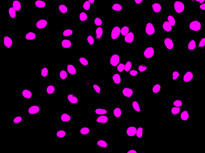
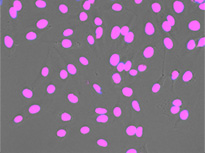
另一方面,作为一种更简单的方法,显微镜通过在训练阶段获取参考图像的方式自动生成训练神经网络所需的基准真相。例如,若要教导神经网络在困难条件下在明场图像中对核进行鲁棒性检测和分割,可以用荧光标记物标记核。显微镜可以自动获取大量的多通道图像(明场和荧光)。在荧光通道上,可以通过自动阈值化轻松检测真实数据(图6和7)。这些对象是训练神经网络的真是数据,由此产生的网络系统让研究人员仅使用明场图像就可以正确找到核(图5*2)。
图4
通过常规机器学习从透射图像获得的核检测结果。通过手动方式将核标记成黄色和将背景标记成淡蓝色进行训练。检测精度低。
图5*2
TruAI从透射图像获得的核检测结果。高度精确检测成为可能。
由于这种生成真实数据的方法几乎不需要人工干预,因此可以在短时间内获取大量训练图像对。这让神经网络有可能在训练过程中适应各种变化和失真,从而获得可以很好应对这些挑战性问题的神经网络模型。
| |
图6 | 图7 |
TruAI的无标记分割训练
为了演示一个典型用例,使用以下参数对在缓冲液填充水平、凝结效果和弯液面成像干扰方面有所差异的整个96孔板进行成像:
- UPLSAPO物镜(10X倍率,NA = 0.6)
- 缓冲液中的贴壁HeLa细胞(固定)
- GFP通道:组蛋白2B GFP作为细胞核的标记物(真实数据)
- 明场通道:三个步进为6μm的Z切片(在训练中包括散焦图像)
通过使用常规方法对荧光图像进行自动分割,可以生成神经网络训练的真实数据。在训练期间在示例数据集中加入略微散焦的图像,让神经网络可以在以后更好地应对轻微的焦点变化。如图8*2所示,使用组对真实数据和明场图像训练神经网络。挑选5个孔,每个孔选择40个位置作为训练数据。训练阶段使用NVIDIA GTX 1080Ti显卡(GPU)花费了大约160分钟。
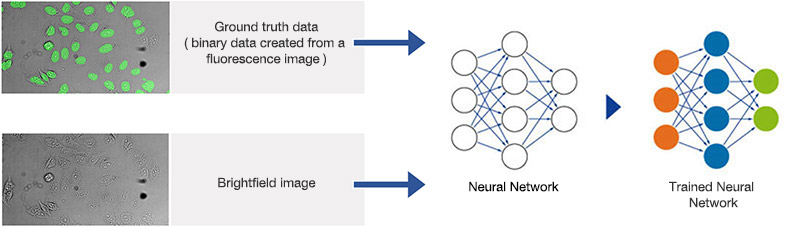
图8*2
显示神经网络训练过程的示意图。
无标记核检测和分割
在检测阶段,将经过训练的神经网络模型应用于明场图像,如图9*2所示。其可以针对每个像素预测是否属于一个细胞核。结果为概率图像,可以通过对概率进行颜色编码并生成叠加图像的方式令其可视化,如图10*2和11*2所示。
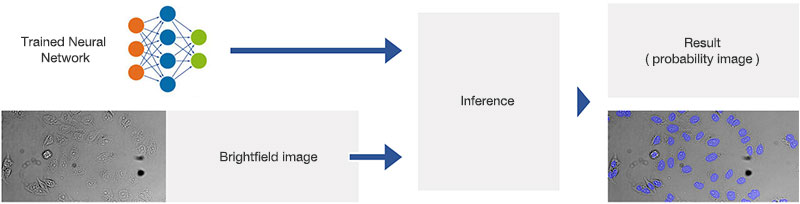
图9*2
经过训练神经网络的应用示意图(推断)。
图10*2和图11*2中的图像显示,从明场图像中学会预测细胞核的神经网络可以在明场图像中找到细胞核准确位置,从而清楚证明了基于AI方法的价值:
- 核的高精度检测和分割
- 最适合细胞计数和诸如面积或形状等几何测量
- 每个位置的处理时间少于1秒(使用NVIDIA GTX 1080 Ti GPU时)
| |
图10*2 | 图11*2 |
结果验证
尽管TruAI预测可能非常精确且可靠,但仍需仔细验证预测以确保不会产生错误识别或其他错误,这一点至关重要。从这个意义上讲,其类似于传统图像分析思路,但由于其与所使用的训练数据相关,如果不仔细进行验证就很难预测错误。
奥林巴斯cellSens成像软件非常适合对TruAI结果进行系统验证。图12*2在单张图片中,将软件结果与基于荧光的分析结果进行了比较。结果表明,奥林巴斯的TruAI结果与荧光结果非常吻合。
为了进行更详细的验证,可以使用奥林巴斯scanR软件。例如,可在scanR软件中轻松比较孔板的总细胞数(图13)。
但是,使用TruAI得出的总细胞数比基于荧光成像的细胞数高出3%(113万个细胞对110万个细胞核)。
造成这种差异的原因之一是部分细胞核表达的GFP信号很弱,无法被荧光检测,但是TruAI可以检测出这些细胞核。但是,另一原因可以通过检测尺寸超过340 μm2的较大对象确定。
这些总值在荧光图中显示为22,000(2%)个异常大的对象(> 340 μm2),而TruAI结果为7,000(0.6%)。
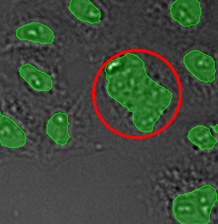
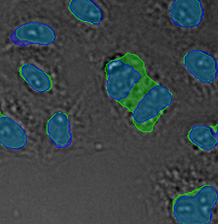
图14*2显示为一系列异常大的对象,表明TruAI对紧密接触核的分割效果更好。
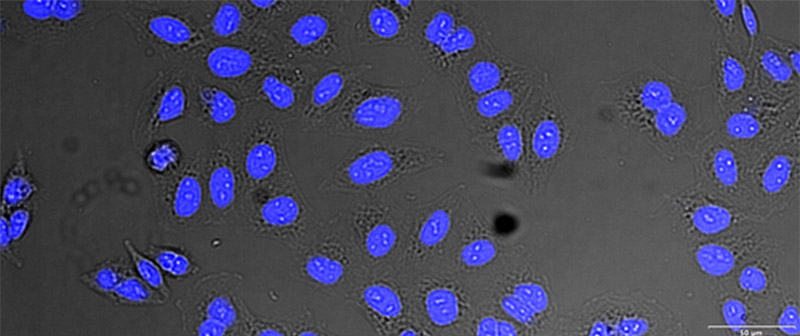
(A)
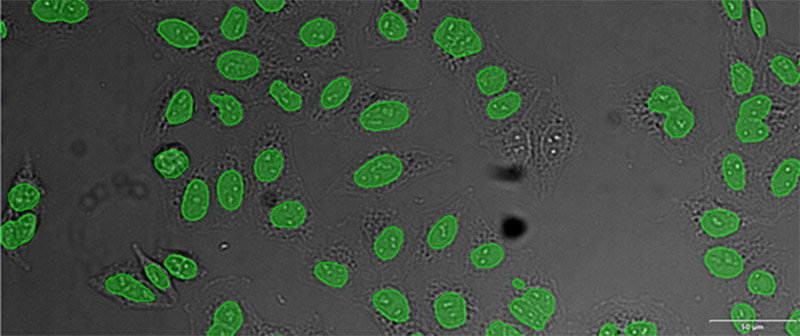
(B)
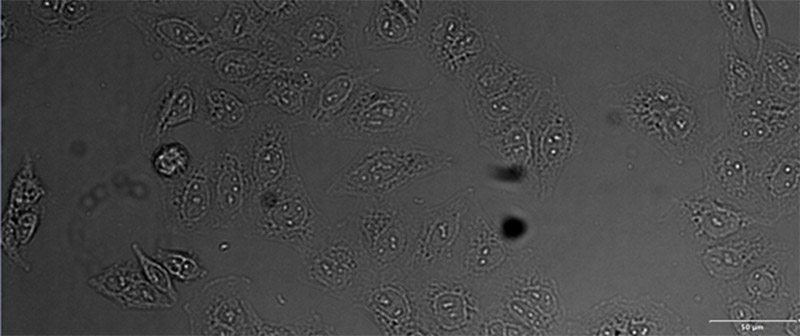
(C)
(D)
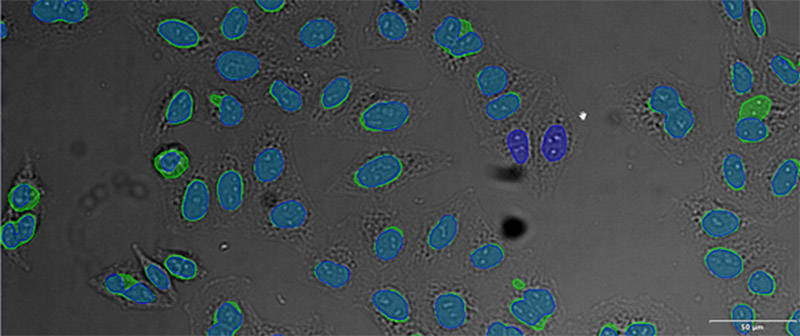
图12*2
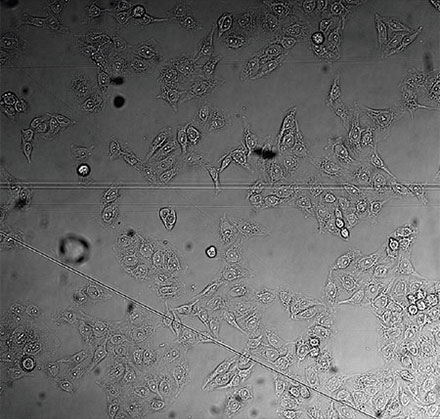
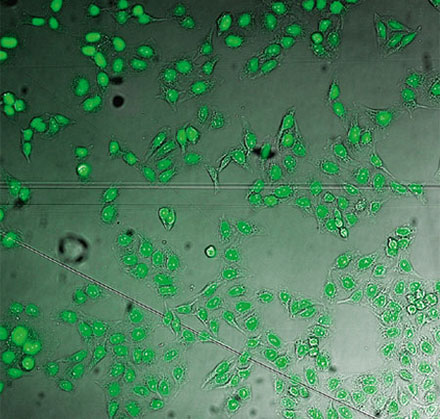
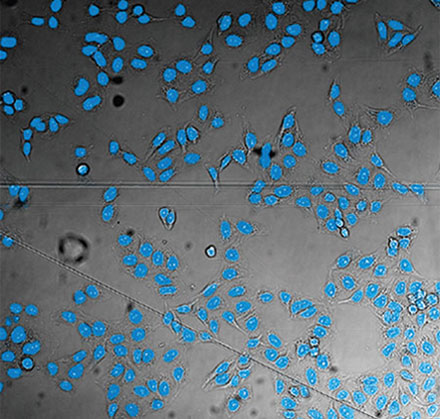
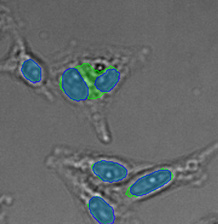
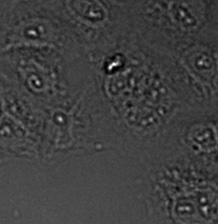
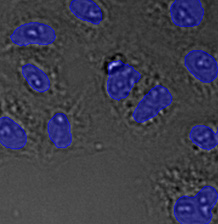
验证数据集的示例图像。(A)GFP核标记,(B)明场图像,(C)明场图像核位置的AI预测,(D)GFP标记(绿色)和AI结果(蓝色)的叠加
|
|
图13
scanR系统:参考方法细胞计数(使用常规方法在GFP通道上计数)(左)和TruAI(使用神经网络在明场通道上计数)(右)的对比。1– 5孔板已被用于训练,且不得考虑对其进行验证。
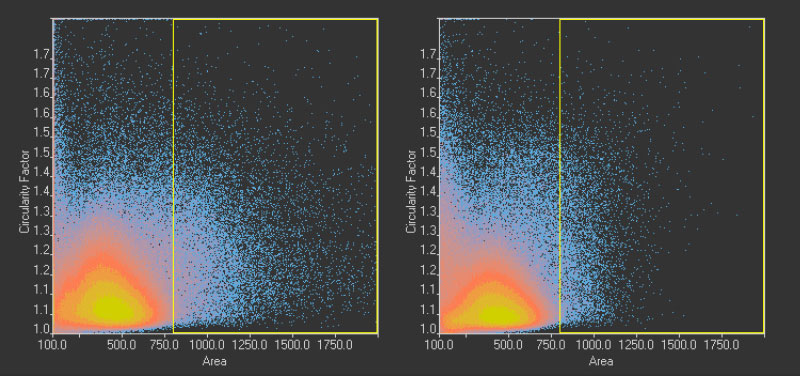
图14*2
显示通过TruAI(右)在GFP通道中检测到110万个核和在明场通道检测到113万个核的圆度与面积分布的散点图。黄色矩形表示异常大的对象。
(A) | (B) | (C) | (D) |
 |  |  |  |
 |  |  |  |
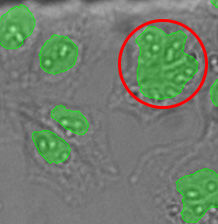 | 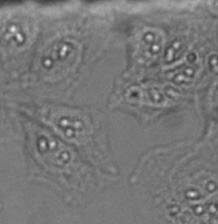 | 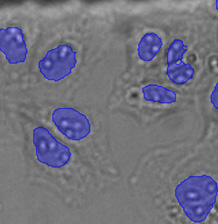 | 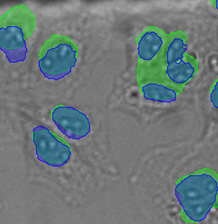 |
图15*2
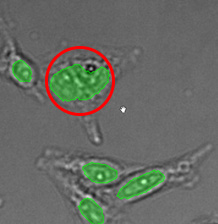
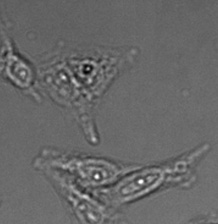
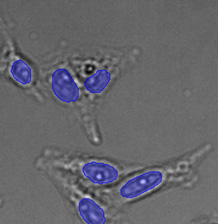
所有验证数据中异常大的对象的三个示例。(A)GFP核标记,(B)明场图像,以及(C)明场图像核位置的TruAI检测。
结论
奥林巴斯cellSens软件的TruAI扩展功能能够仅通过明场透射图像即可获得微孔板中的核位置和标识物。经过短暂培训之后,软件即可具备该功能。经过自动对象检测后,可轻松地编辑数据进行训练。所生成的用于分析透射光图像的网络与传统荧光标记方法相当,甚至更为出色。
使用奥林巴斯TruAI可为诸多活细胞分析工作流程带来显著优势。除提高准确性外,使用明场图像还避免了使用遗传修饰或核标记物的需要。这样不仅可以节约样品制备时间,还节省了其他标记物的荧光通道。此外,更短的明场成像曝光时间还意味着降低了光毒性并缩短了成像时间
作者
Matthias Genenger博士,
Mike Woerdemann博士
产品经理
Olympus Soft Imaging Solutions
GmbH
德国明斯特
*1(Christiansen等人在Silico标记中:预测未标记图像中的荧光标记,细胞杂志,2018年)
引言
用彩色标记物标记细胞,尤其是使用荧光染料标记细胞,对于观察和分析生物学特征和过程非常有用。在这些标记技术发展之前,都是通过无标记方式对生物样品进行显微镜观察分析。由于机器学习方法在图像分析获得巨大发展,近年来无标记观察的重要性又得到大幅度提升。基于深度学习的TruAI对透射光图像分析提供新的途径,并有可能取代荧光标记方法进行细胞和细胞器的结构分析*1 (图1)。

图1
从左到右:TruAI预测的核位置(蓝色),绿色荧光蛋白(GFP)标记核的组蛋白2B(绿色)和原始明场透射图像(灰色)。
任何无标记方法都不能完全替代荧光,这是因为直接将标记物附着到目标分子上所获得的信息仍然具有价值。但是,以较少标记获得相关样品信息存在着明显的优势:
- 降低样品制备的复杂性
- 减少光毒性
- 为其他标记保留荧光通道
- 成像更快
- 通过避免转染或化学标记提高活细胞的活力
无标记分析的局限性很大程度上在于缺乏有效解决透射光图像分析固有问题的方法。这些限制包括:
- 低对比度,尤其是明场图像
- 与荧光显微镜相比,光路上的灰尘和其他缺陷会对图像质量产生负面影响
- 改进技术条件提高对比度,如相差或微分干涉(DIC)
- 与荧光相比,背景水平更高
|
图2
细节图显示了明场透射成像中可能出现的强烈背景和不均匀性。注意不良颗粒物造成的散焦问题。
TruAI背后的技术
透射明场成像是无标记分析应用的自然方法,但是其也造成长期以来尚未解决的图像分析和分割难题。为了应对这些挑战,奥林巴斯将图像分析方法TruAI集成到cellSens软件中,TruAI是基于深度卷积神经网络系统工作的。这种神经网络架构近期已被视为最强大的对象分割技术(Long等,2014:用于语义分割的完全卷积网络)。这种神经网络对于各种挑战性图像分析任务具有非常好的适用性,,它将成为透射明场图像进行无标记分析的最佳选择。
在训练阶段,TruAI自动学习如何预测诸如细胞或细胞器位置和曲线等相应参数。在训练阶段,向网络发送图像和“真实”数据(即,对感兴趣的对象进行目标注释)。一旦对网络进行训练之后,就可以将其应用于新图像并以高精度预测目标物。
通常,在机器学习中,注释(如,细胞边界)由人类专家提供。这个步骤可能非常乏味并且耗费时间,这是因为神经网络需要大量训练数据才能充分发挥其潜力(图3)。因此,拥有方便且易于使用的注释工具至关重要。
也可以使用TruAI这种更简单的方法。例如,若要教导神经网络在困难条件下在明场图像中对核进行鲁棒检测和分割,可以用荧光标记物标记核。显微镜可以自动获取大量的多通道图像(明场和荧光)。在荧光通道上,可以通过自动阈值化轻松检测真实数据。这些对象是训练神经网络的真实数据,由此产生的网络让研究人员仅使用明场图像就可以正确找到核(图4*2)。

图3
通过常规机器学习从透射图像获得的核检测结果。通过手动方式将核标记成黄色和将背景标记成淡蓝色进行训练。检测精度低。

图4*2
TruAI从透射图像获得的核检测结果。高度精确检测成为可能。
由于这种生成真实数据的方法几乎不需要人工干预,因此可以在短时间内获取大量训练图像对。这让神经网络有可能在训练过程中适应各种变化和失真,从而获得可以很好应对这些挑战性问题的神经网络模型。
无标记分割训练
为了演示一个典型用例,使用以下参数对在缓冲液填充水平、凝结效果和弯液面成像干扰方面有所差异的整个96孔板进行成像:
- LUCPLFLN20XPH物镜(NA = 0.45)
- 缓冲液中的贴壁HeLa细胞(固定)
- 荧光通道:DAPI作为核标记物(真实数据)
- 透射通道:明场
通过使用常规方法对荧光图像进行自动分割,可以生成神经网络训练的真实数据。在训练期间在示例数据集中加入略微散焦的图像,让神经网络可以在以后更好地应对轻微的焦点变化。如图5*2所示,使用组对基准真相和明场图像训练神经网络。训练阶段使用NVIDIA GTX 1060显卡(GPU)花费了大约180分钟。
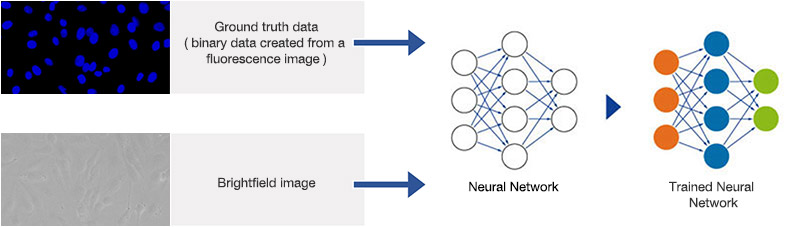
图5*2
显示神经网络训练过程的示意图。
无标记核检测和分割
在检测阶段,将经过训练的神经网络模型应用于明场图像,如图6*2所示。其可以针对每个像素预测是否属于一个细胞核。结果为概率图像,通过对概率进行颜色编码并生成叠加图像。
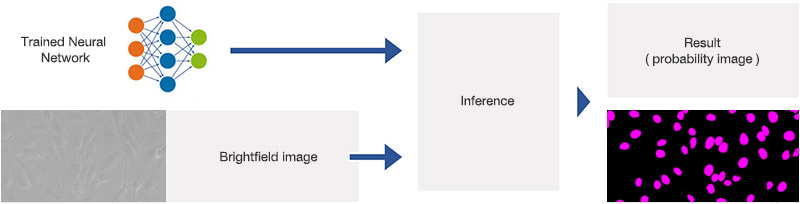
图6*2
经过训练神经网络的应用示意图(推断)。
从明场图像中学会预测细胞核的神经网络可以在明场图像中准确找到细胞核位置,从而清楚证明了基于AI方法的价值:
- 核的高精度检测和分割
- 最适合细胞计数和诸如面积或形状等几何测量
- 每个位置的处理时间少于1秒(使用NVIDIA GTX 1060 GPU时)
结果验证
尽管深度学习预测可能非常精确且可靠,但仍需仔细验证预测以确保不会产生伪影或其他错误,这一点至关重要。从这个意义上讲,其类似于传统图像分析思路,但由于其与所使用的训练数据相关,如果不仔细进行验证就很难预测错误。
奥林巴斯cellSens成像软件非常适合对AI结果进行系统验证。图7在单张图片中,将软件结果与基于荧光的分析结果进行了比较。其表明奥林巴斯的AI结果与荧光检测结果非常吻合。
但是,使用深度学习方法得出的总细胞数比基于荧光成像的细胞数高出3%(2523个细胞对24.59亿个细胞核)。
造成这种差异的原因之一是部分细胞核表达的GFP信号很弱,无法被荧光检测,但是TruAI可以检测出这些细胞核。但是,另一原因可以通过检测尺寸超过400 μm2的较大对象确定。
这些总值在荧光图中显示为126(5.1%)个异常大的对象(> 400 μm2),而TruAI结果为24(0.95%)。
图8*2显示为一系列异常大的对象,表明TruAI对紧密接触核的分割效果更好。
|
|
|
|
图7
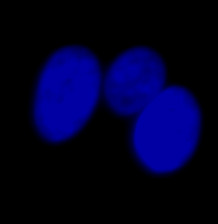
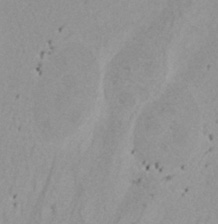
验证数据集的示例图像。(A)GFP核标记,(B)明场图像,(C)明场图像核位置的AI预测,(D)GFP标记(绿色)和AI结果(蓝色)的叠加
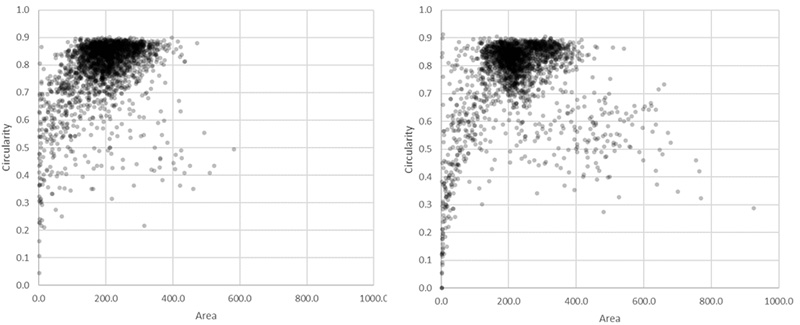
图8*2
显示通过AI(右)在GFP通道中检测到110万个核和在明场通道检测到113万个核的圆度与面积分布的散点图。黄色矩形表示异常大的对象。
(A) | (B) | (C) | |
 |  |  | |
 |  |  | |
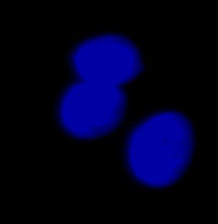 | 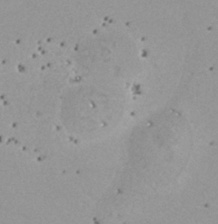 |  | |
| 图9 整个验证数据中异常大的对象的三个示例。(A)GFP核标记,(B)明场图像,以及(C)明场图像核位置的TruAI检测。 | |||
结论
奥林巴斯cellSens软件的深度学习扩展功能能够仅通过明场透射图像即可获得微孔板中的核位置和标识物。经过短暂培训之后,软件即可具备该功能。经过自动对象检测后,可轻松地编辑数据进行训练。所生成的用于分析透射光图像的网络与传统荧光标记方法相当,甚至更为出色。
使用奥林巴斯TruAI可为诸多活细胞分析工作流程带来显著优势。除提高准确性外,使用明场图像还避免了使用遗传修饰或核标记物的需要。这样不仅可以节约样品制备时间,还节省了其他标记物的荧光通道。此外,更短的明场成像曝光时间还意味着降低了光毒性并缩短了成像时间
作者
Matthias Genenger博士,
Mike Woerdemann博士
产品经理
Olympus Soft Imaging Solutions
GmbH
德国明斯特
*1(Christiansen等人在Silico标记中:预测未标记图像中的荧光标记,细胞杂志,2018年)
*2 虽然它已经成为医学研究中最重要的细胞系之一,但我们必须认识到Henrietta Lacks对科学的贡献是在未经她同意的情况下发生的。这一不公正现象在导致免疫学、传染病和癌症方面重大发现的同时,也引发了关于医学中的隐私、伦理和同意方面的重要对话。
要了解更多关于Henrietta Lacks的生平和她对现代医学的贡献,请点击这里。
http://henriettalacksfoundation.org/
对不起,此内容在您的国家不适用。