Monitoramento da dinâmica do ciclo celular durante a diferenciação de células-tronco com a tecnologia de aprendizagem profunda TruAI™ do sistema scanR
Introdução
Durante o desenvolvimento dos mamíferos, as células-tronco embrionárias (ESC) passam por especificação celular que dá origem aos folhetos germinativos embrionários, que geram a diversidade de tipos celulares no organismo adulto. As ESCs são caracterizadas pela incrível capacidade de autorenovação e proliferação, com ciclos de divisão curtos devido às fases de GAP truncadas (G1 e G2). Durante a diferenciação, eventos celulares marcantes acontecem: as células mudam de morfologia, aumentam de tamanho em cerca de dez vezes, mudam a proporção entre núcleo e citoplasma e se tornam planas e alongadas; os programas de expressão de genes específicos ao destino são ativados e ocorrem modificações globais de cromatina. O mais importante, contudo, é a desaceleração do ciclo celular. Células somáticas diferenciadas têm fases G1 e G2 longas, controle de checkpoint rigoroso e ciclos de divisão bem regulados (Padgett e Santos 2020) (Figura 1).
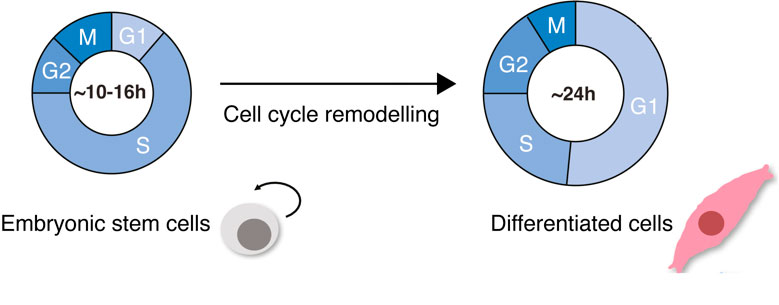
O remodelamento do ciclo celular não é apenas um fenômeno que ocorre na diferenciação da ESC. Alterações nas dinâmicas do ciclo celular ocorrem frequentemente na biologia. Por exemplo, quando as células passam por regeneração após infecções virais e transformações malignas. Estudar a mudança de dinâmica do ciclo celular no contexto da diferenciação celular é, por este motivo, importante para compreender os mecanismos potencialmente conservados de controle do ciclo celular e colaborará com a compreensão do equilíbrio entre os estados de saúde e doença.
Para monitorar as mudanças durante a diferenciação em células individuais, podemos medir a atividade de proteínas geneticamente codificadas envolvidas em diferentes fases do ciclo celular (Araujo et al. 2016; Sakaue-Sawano et al. 2008). Um sensor desse tipo é o indicador de ciclo celular fluorescente baseado em ubiqutinação FUCCI(CA), desenvolvido por Miyawaki e colegas (Sakaue-Sawano et al. 2017), que monitora a degradação de Cdt1 e geminina, duas proteínas importantes do ciclo celular e produz uma separação contrastante de três cores das fases G1, S e G2 (vermelho, verde e amarelo) que pode ser observada nos canais YFP e mCherry de um microscópio (Figura 2).
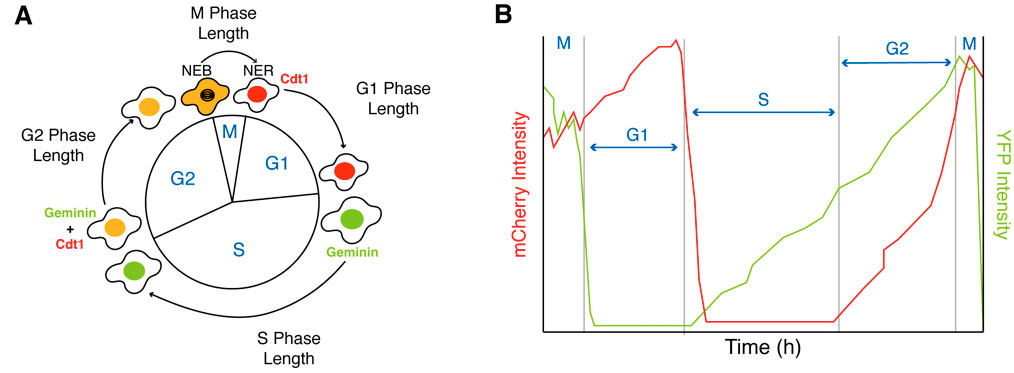
Figura 2. Medição das fases G1, S, G2 e M do ciclo celular que usa o sensor FUCCI(CA). (A) Esquema do sensor FUCCI(CA). (B) Exemplos de traços da duração da fase G1, S, G2 e M baseada nos níveis de expressão do Cdt1 (canal mCherry) e geminina (canal YFP).
No entanto, monitorar a dinâmica do ciclo celular durante a diferenciação apresenta desafios para a formação de imagem e a análise de dados. A formação de imagem de células vivas de longo prazo ao longo de dias requer uma temperatura estável, controle de CO2, baixa iluminação para minimizar a fototoxicidade e rotinas de autofoco robustas para garantir imagens de boa qualidade durante todo o processo de time-lapse (câmera rápida).
O maior desafio da análise é a segmentação e o rastreamento posterior de células individuais em condições de baixa intensidade de fluorescência e de alta confluência durante o movimento, a divisão e as mudanças drásticas de forma das ESCs durante a diferenciação.
Todos esses desafios podem ser superados com o microscópio scanR de epifluorescência de alto teor da Olympus, equipado com uma incubadora cellVivo, hardware de compensação de desvio Z (ZDC) para foco automático, tecnologia de aprendizagem profunda TruAI ™ para segmentação e um módulo cinético para rastreamento.
Objetivos
Nesta nota de aplicativo, mostramos que aplicar a tecnologia TruAI pode melhorar significativamente a detecção e o rastreamento de ESCs individuais por meio de formação de imagens de longo prazo com base no sensor FUCCI(CA). Especificamente, essa tecnologia é poderosa em células que não possuem um marcador nuclear para rastreamento, evitando iluminação e fototoxicidade desnecessárias.
Para isso, a tecnologia TruAI é usada para gerar um modelo de rede neural profunda (DNN) que pode identificar bem a posição de ESCs pluripotentes e células diferenciadas em todos os pontos de tempo, mesmo quando a intensidade de fluorescência é muito baixa. Ela permite um monitoramento robusto das fases G1, S e G2 das populações celulares ao longo do tempo. Posteriormente, podemos aplicar o módulo cinético scanR para rastrear milhares de células, avaliar seus traços por longos períodos de tempo e obter informações dinâmicas quantitativas sobre mudanças do ciclo celular no nível de uma única célula.
Para demonstrar a versatilidade do sistema scanR como ferramenta de pesquisa, todos os dados mostrados, como gráficos, gráficos de dispersão, galerias de objetos, trilhas e traços cinéticos, são gerados apenas com o software scanR.
Configuração experimental
Células-tronco embrionárias humanas H1 (hESC) que expressam o sensor FUCCI(CA) são tratadas com 50 ng/ml de proteína morfogênica óssea 4 (BMP4) para gerar a diferenciação na linhagem de mesendoderma. Para fotografar essas células H1, usamos o microscópio scanR com uma lente UPLANSAPO 10X nos canais mCherry e YFP a cada 15 minutos por 6 dias.
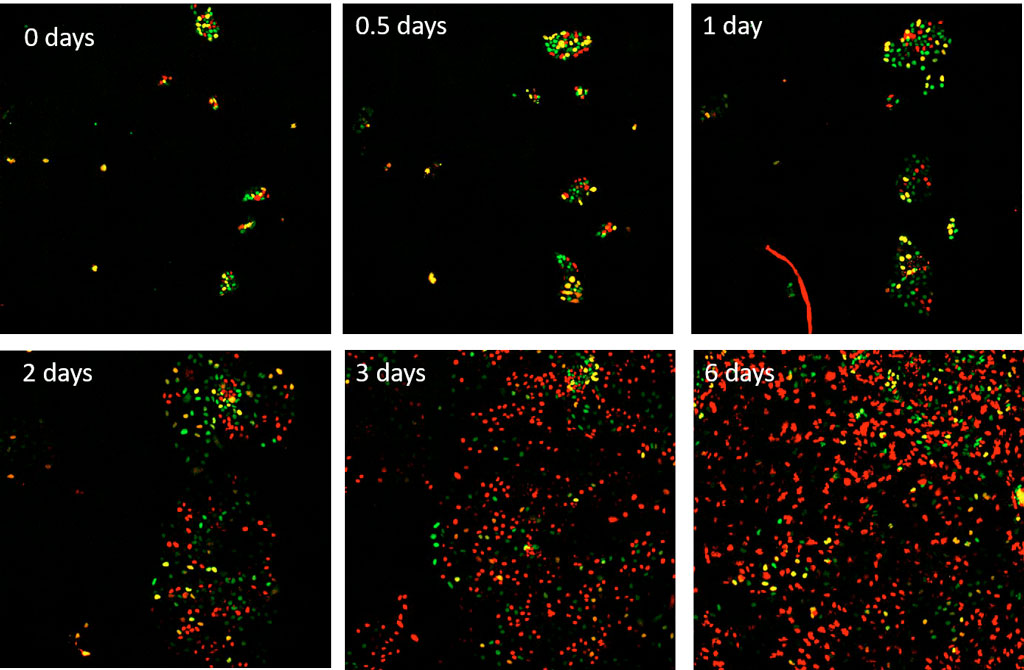
Figura 3. Séries temporais ao longo de 6 dias de células hESC H1 expressando o sensor FUCCI(CA), estimuladas com BMP4 no início da formação de imagem. Vermelho, verde e amarelo correspondem respectivamente às fases G1, S e G2.
Desenvolvimento de uma DNN para detectar células para condições de formação de imagem de células vivas
Uma única célula que expressa o sensor FUCCI(CA) pode exibir grande variação na intensidade da fluorescência durante um monitoramento de vários dias. Em especial, a intensidade total pode ser muito baixa na transição da fase G1 à S (Figura 2b). Isso pode causar a falha dos algoritmos de rastreamento, pois as trilhas são interrompidas nos pontos de tempo em que é difícil detectar as células.
Para concluir o rastreamento do maior número possível de células ao longo de vários dias, é necessário desenvolver um modelo de DNN capaz de detectar células em intensidades muito fracas. Em um artigo anterior, mostramos que isso é possível com pares de imagens de longa exposição e iluminação intensa de células fluorescentes em comparação com exposição curta e iluminação fraca (Woerdemann 2020). Resumidamente, as imagens de longa exposição são usadas para segmentação das células e uma máscara é gerada. Essa máscara é usada como referência de verdade para treinar o modelo de DNN para detectar células nos níveis de baixa exposição.
Como alternativa, pode-se fazer anotações manuais no conjunto de dados a ser analisado para treinar o modelo de DNN. Para esta tarefa, o software CellSens ™ da Olympus pode ser usado. Os modelos de DNN desenvolvidos no software cellSens podem ser importados no software scanR. Nesta nota de aplicativo, usamos o segundo método manual e fornecemos a ordem de centenas de anotações de células de várias intensidades em pontos de tempo diferentes para treinar o modelo de DNN.
Segmentação: Comparação da tecnologia de aprendizagem profunda TruAI com os métodos clássicos
O modelo de DNN desenvolvido é aplicado aos canais individuais de fluorescência mCherry e YFP. Isso cria um mapa de probabilidade de IA de pixel para cada canal. Quanto maior a probabilidade de IA em um pixel, maior será a confiança de ele pertence a uma célula. Posteriormente, os mapas de probabilidade de IA de ambos os canais são somados e a segmentação é realizada aplicando um limite de intensidade a essa soma (Figura 4e e 4f).
Para comparar os resultados com métodos clássicos, os canais de fluorescência mCherry e YFP passam por uma correção de fundo que usa um algoritmo de bola rolante e somados. Essa soma é segmentada usando um limiar de intensidade (Figura 4c) ou um método de detecção de borda (Figura 4d). Todas as etapas de processamento de imagem descritas acima são conduzidas no software de análise scanR.
As ferramentas estatísticas do sistema scanR são utilizadas para criar a Tabela 1, na qual os resultados se referem a um único campo de visão durante um período de 6 dias.
Tabela 1. Comparação do número de células segmentadas por método
| Pontos de tempo | Métodos de segmentação | ||
|---|---|---|---|
| Limiar de intensidade | Detector de borda | Tecnologia TruAI | |
| 0 h | 78 | 103 | 93 |
| 24 h | 209 | 188 | 240 |
| 48 h | 421 | 281 | 475 |
| 72 h | 847 | 654 | 986 |
| 96 h | 1480 | 1330 | 1590 |
| 160 h | 1340 | 1460 | 1610 |
|
Soma de todos os quadros
0–160 h | 504131 | 573179 | 659199 |
Uma comparação simples da imagem de fluorescência com a imagem de probabilidade de IA (Figura 4a e 4b) revela que a abordagem TruAI tem maior sensibilidade ao mesmo tempo que detecta todos os tipos de células. Isso é corroborado pelos resultados resumidos na Tabela 1, juntamente com todo o time-lapse. O método de limite de intensidade clássico não consegue detectar células fluorescentes fracas nem determinar a forma correta (Figura 4c). O método de detecção de borda melhora o contorno das células detectadas, mas ainda perde muitas células pouco iluminadas (Figura 4d). A tecnologia de aprendizagem profunda TruAI é confiável para detectar células pouco iluminadas e os limites são bem definidos (Figuras 4e e 4f).
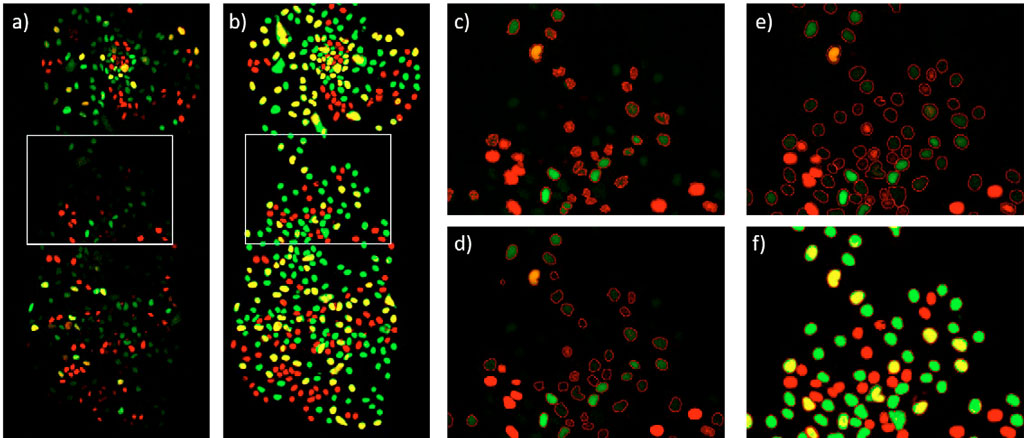
Figura 4. Colônia de células ESC no ponto de tempo 48 h. a) Canais fluorescentes mCherry (vermelho, fase G1) e YFP (verde, fase S). A fase G2 é vista como uma combinação de mCherry e YFP (amarelo). b) Probabilidade TruAI no mCherry (vermelho) e YFP (verde). As células com alta probabilidade de IA em ambos os canais são mostradas em amarelo. c) Segmentação com um limite de intensidade na soma dos canais de fluorescência mCherry e YFP d) Segmentação com um detector de borda na soma dos canais de fluorescência mCherry e YFP e) Segmentação na soma das intensidades de probabilidade TruAI, sobrepostas com a fluorescência imagem f) igual a e) mas sobreposta com a probabilidade TruAI.
Análise das populações de células ao longo do tempo
As intensidades mCherry e YFP das células segmentadas com a tecnologia TruAI são mostradas em um gráfico de dispersão em diferentes intervalos de tempo (Figura 5). As células que têm apenas intensidade mCherry correspondem à fase G1, as células que têm apenas intensidade YFP correspondem à fase S e as células com intensidades em ambos os canais correspondem às células no estado G2. A Figura 5 mostra que, em momentos iniciais, a maioria das células está no estado S, enquanto em momentos posteriores, a maioria das células está no estado G1. Esta mudança de população corresponde aos perfis de ciclo celular esperados para células pluripotentes e diferenciadas, respectivamente (Figura 1).
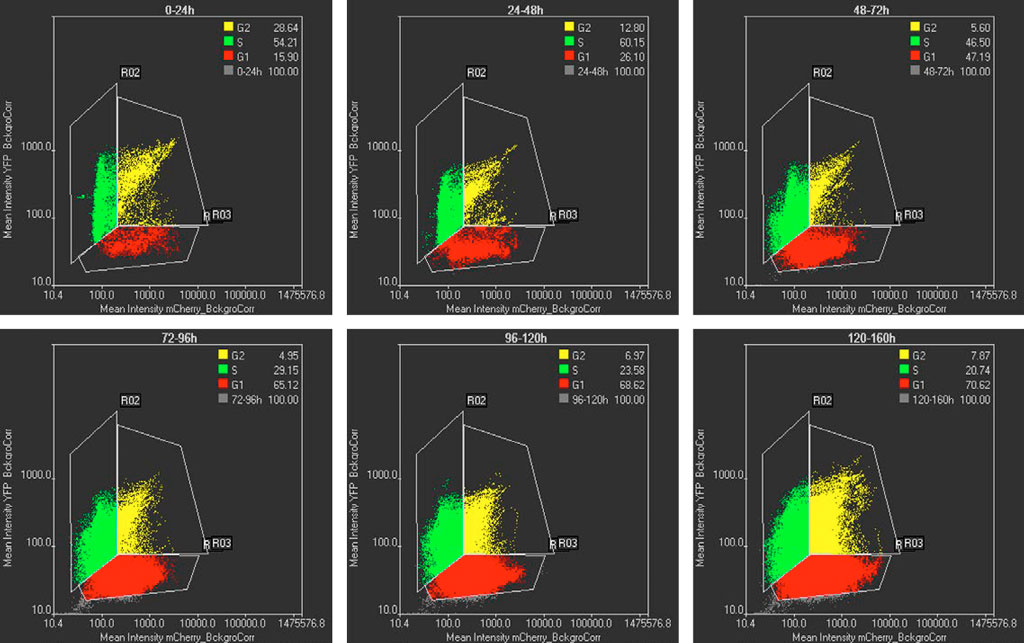
Figura 5. Gráficos de dispersão de intensidades mCherry em relação a YFP em diferentes intervalos de tempo. Cada ponto no gráfico de dispersão representa uma célula segmentada pela tecnologia TruAI. As células G1, S e G2 são representadas em vermelho, verde e amarelo, respectivamente. As porcentagens das populações G1-S-G2 são mostradas nas inserções. Os intervalos de tempo são mostrados em cima de cada gráfico.
Rastreamento de células: Comparação da tecnologia TruAI com os métodos clássicos
As células segmentadas com os três métodos (limite de intensidade, detector de borda e tecnologia TruAI) foram rastreadas com mesmas configurações de rastreamento do módulo cinético scanR. As ferramentas estatísticas do sistema scanR são utilizadas para criar a Tabela 2, na qual os resultados se referem a um único campo de visão durante um período de 6 dias.
Tabela 2. Comparação do número de células rastreadas por método
| Pontos de tempo | Número de células rastreadas | ||
|---|---|---|---|
| Limiar de intensidade | Detector de borda | Tecnologia TruAI | |
| 24 h ou mais | 302 | 1030 | 1965 |
| 48 h ou mais | 16 | 126 | 426 |
| 72 h ou mais | 0 | 27 | 72 |
| 96 h ou mais | 0 | 0 | 5 |
A tabela mostra que usar a tecnologia TruAI para segmentação melhora claramente os resultados de rastreamento subsequentes. Para esse conjunto de dados, considerando um número médio de 200 células por campo de visão no tempo de 24 h (Tabela 1), mais de 70 células podem ser rastreadas ao longo de 3 dias e é possível rastrear células por 4 dias ou mais. A capacidade de rastrear esse grande número de células por bastante tempo é importante porque permite estudar a dinâmica do ciclo celular em muitas divisões celulares de maneira estatisticamente significativa.
Análise das trilhas celulares
É possível extrair uma ampla gama de parâmetros de cada trilha gerada pelo sistema scanR.
- Para identificar as trilhas mais longas, o parâmetro “tempo de vida” pode ser representado em um histograma e um “gate” para trilhas com mais de 24 h pode ser criado (Figura 6a).
- Para filtrar ainda mais as trilhas, os parâmetros “Primeiro (tempo)” e “Último (tempo)” podem ser representados em um gráfico de dispersão (Figura 6b). Isso pode ajudar a identificar as células rastreadas apenas durante os primeiros dias (células ESC com ciclo curto), as células rastreadas apenas nos últimos dias (células diferenciadas com ciclo longo) e as células rastreadas do início ao fim do “time-lapse” (células nas quais o processo de diferenciação é monitorado). Uma vez que essas trilhas são identificadas, as intensidades de mCherry e YFP podem ser representadas ao longo do tempo para extrair informações quantitativas sobre a dinâmica do ciclo celular (Figura 6 c, d, e).
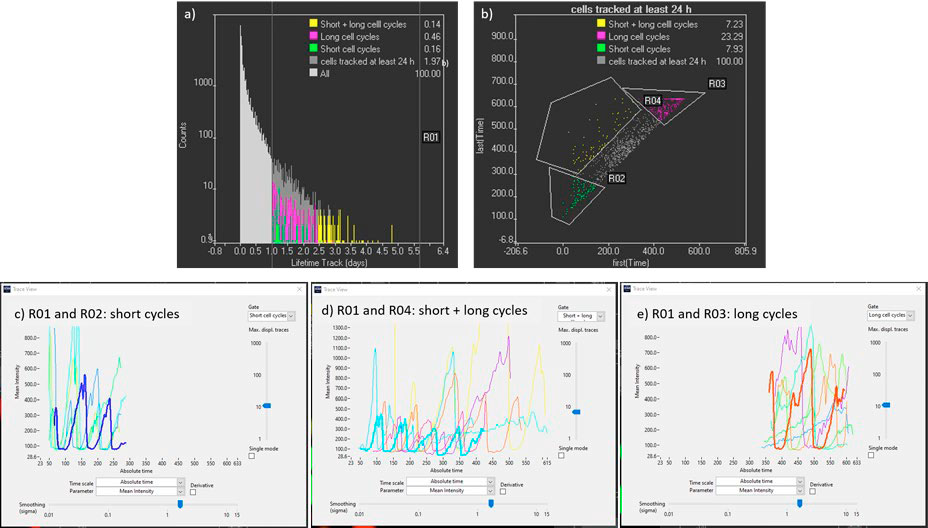
Figura 6. a) Histograma do tempo de vida da trilha usado para filtrar trilhas longas (R01). b) Gráfico de dispersão com ponto de tempo inicial e final das trilhas para identificar células rastreadas do início ao final do “time-lapse” (R01 e R04). c), d) e e) Traços cinéticos de células mCherry rastreados por mais de 24 h com pontos iniciais e finais antecipados, com pontos inicial antecipado e final atrasado e com pontos iniciais e finais atrasados, respectivamente. Os pontos de tempo são separados em intervalos de 15 minutos.
Da Figura 6c a 6e, observa-se que a duração do sinal mCherry (estado G1) aumenta com o tempo, de aproximadamente 2 horas para as células ESC a 24 horas, depois da diferenciação das células. Para uma análise aprofundada da dinâmica do ciclo celular, é possível selecionar as trilhas da região R01 e R04, nas quais a transformação de ESC em células diferenciadas é monitorada em quatro ou mais divisões celulares (Figura 6d).
A Figura 7 representa o monitoramento de uma célula ESC em diferenciação ao longo de cinco ciclos celulares. A Figura 7a mostra a posição da ESC na colônia no ponto de tempo 52 (13 h) com a trilha até o ponto de tempo 422 (105 h). A Figura 7b mostra um instantâneo no tempo 280 (70 h). Observe que a intensidade dessa célula é extremamente baixa e não foi detectada pelos métodos clássicos. A Figura 7c mostra o mesmo instantâneo, mas com a imagem de probabilidade TruAI. A intensidade da probabilidade da célula rastreada é tão alta quanto das outras células. As Figuras 7d e 7e representam uma galeria da célula rastreada em pontos de tempo a cada 15 minutos para fluorescência e probabilidade TruAI, respectivamente. Observe que ao terminar a fase G1 (cor vermelha), a intensidade total é sempre muito baixa. A Figura 7f representa as oscilações de intensidade de fluorescência mCherry (superior) e YFP (inferior) a partir das quais os períodos do ciclo celular dos estados G1, S, G2 e M podem ser extraídos.
A partir desses dados, observamos que esta célula em particular passa mais tempo no estado G1 e menos tempo nas fases S e G2 conforme o tempo avança. O estado M é sempre muito curto, a posição exata parece uma queda no sinal mCherry e um decaimento drástico no sinal YFP.
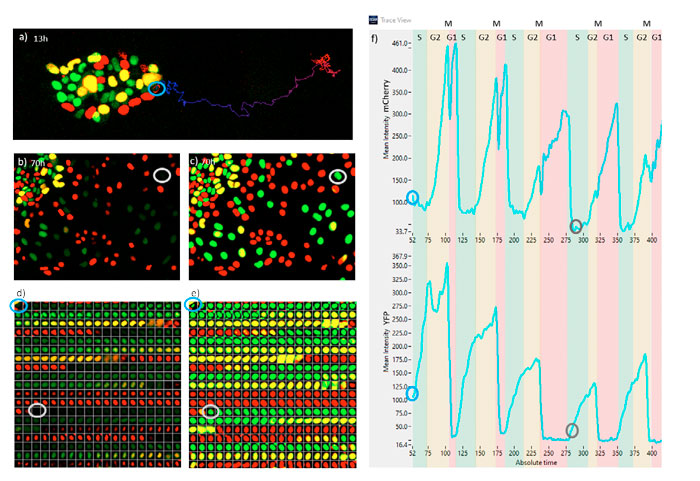
Figura 7. a) Instantâneo da fluorescência no ponto de tempo de 13 h. b) Instantâneo de fluorescência no ponto de tempo de 70 h. c) Instantâneo TruAI às 70 h. d) Sequência do “time-lapse” de fluorescência da célula rastreada em intervalos de 15 minutos. e) Sequência do “time-lapse” TruAI da célula rastreada em intervalos de 15 minutos. f) Traços de fluorescência de mCherry e YFP da célula rastreada, indicando os períodos das diferentes fases do ciclo celular. As etapas de tempo correspondem a 15 min. Os círculos em azul e cinza de a–f indicam a mesma célula em 13 h ou 70 h, respectivamente.
Conclusão
Essa nova abordagem de aprendizagem profunda de análise da dinâmica do ciclo celular durante a diferenciação de ESC combinada com o módulo cinético do sistema de triagem de alto teor scanR permite a aquisição de dados altamente reprodutíveis, quantitativos e estatisticamente significativos sobre as mudanças na dinâmica do ciclo celular de células únicas durante a diferenciação. Milhares de células mudando de forma e morfologia podem ser reconhecidas e rastreadas ao longo de dias de diferenciação usando medições de fluorescência. Essa ferramenta é poderosa para estudar a dinâmica do ciclo celular no desenvolvimento inicial e na reprogramação, bem como em células em transformação celular (malignidade), onde a regulação do ciclo celular é o principal acontecimento.
Referências
Padgett, J., e Santos, S.D.M. 2020. “From clocks to dominoes: lessons on cell cycle remodeling from embryonic stem cells”. FEBS Letters. 10.1002/1873-3468.13862.
Araujo, A.R., Gelens, L., Sheriff, R.S.M., e Santos, S.D.M. 2016. “Positive feedback keeps duration of mitosis temporally insulated from upstream cell cycle events”. Molecular Cell 64, 362–375.
Sakaue-Sawano et al. 2008. “Visualizing Spatiotemporal Dynamics of Multicellular Cell-Cycle Progression”. Cell 132, 487–498.
Sakaue-Sawano et al. 2017. “Genetically Encoded Tools for Optical Dissection of the Mammalian Cell Cycle”. Molecular Cell 68, 626–640.
Woerdemann, M., e Genenger, M. 2020. “TruAI™ Technology with Deep Learning for Quantitative Analysis of Fluorescent Cells with Ultra-Low Light Exposure”. Nota de aplicativo da Olympus. www.olympus-lifescience.com/en/resources/white-papers/ultra-low_light_exposure_analysis/
Autores
Joe Pagdett e Silvia Santos
Quantitative Stem Cell Biology Lab, The Francis Crick Institute, 1 Midland Road, NW1 1AT, Londres, Reino Unido
Manoel Veiga Gutierrez
Olympus Soft Imaging Solutions GmbH, Johann-Krane-Weg 39, 48149 Muenster, Alemanha
Produtos usados nesta aplicação
foi adicionado com sucesso aos seus favoritos
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
Not Available in Your Country
Sorry, this page is not
available in your country.