Bei der Instanzsegmentierung wird ein Bild in einzelne Instanzen eines Objekts, z. B. einzelne Zellen oder Zellkerne, zerlegt. Da diese Aufgabe bisher eine Nachbearbeitung erforderte, haben wir unsere TruAI Deep-Learning-Technologie erweitert, um die Instanzsegmentierung deutlich zu vereinfachen.
In diesem Beitrag erläutern wir diese einfache Instanzsegmentierungsmethode anhand verschiedener Bildbeispiele. Lesen Sie weiter, um mehr über die Vorteile für die Analyse von Mikroskopiebildern und damit verbundene neue Möglichkeiten zu erfahren.
Instanzsegmentierung bei Mikroskopiebildern
Die automatisierte Analyse von Mikroskopiebildern erfordert in der Regel eine Segmentierung, bei der das Bild in seine Objekte, Einzelteile und den Hintergrund aufgeteilt wird. Relativ einfach zu segmentieren sind Bilder mit gut differenzierten Zellkernen (siehe Abbildung 1).
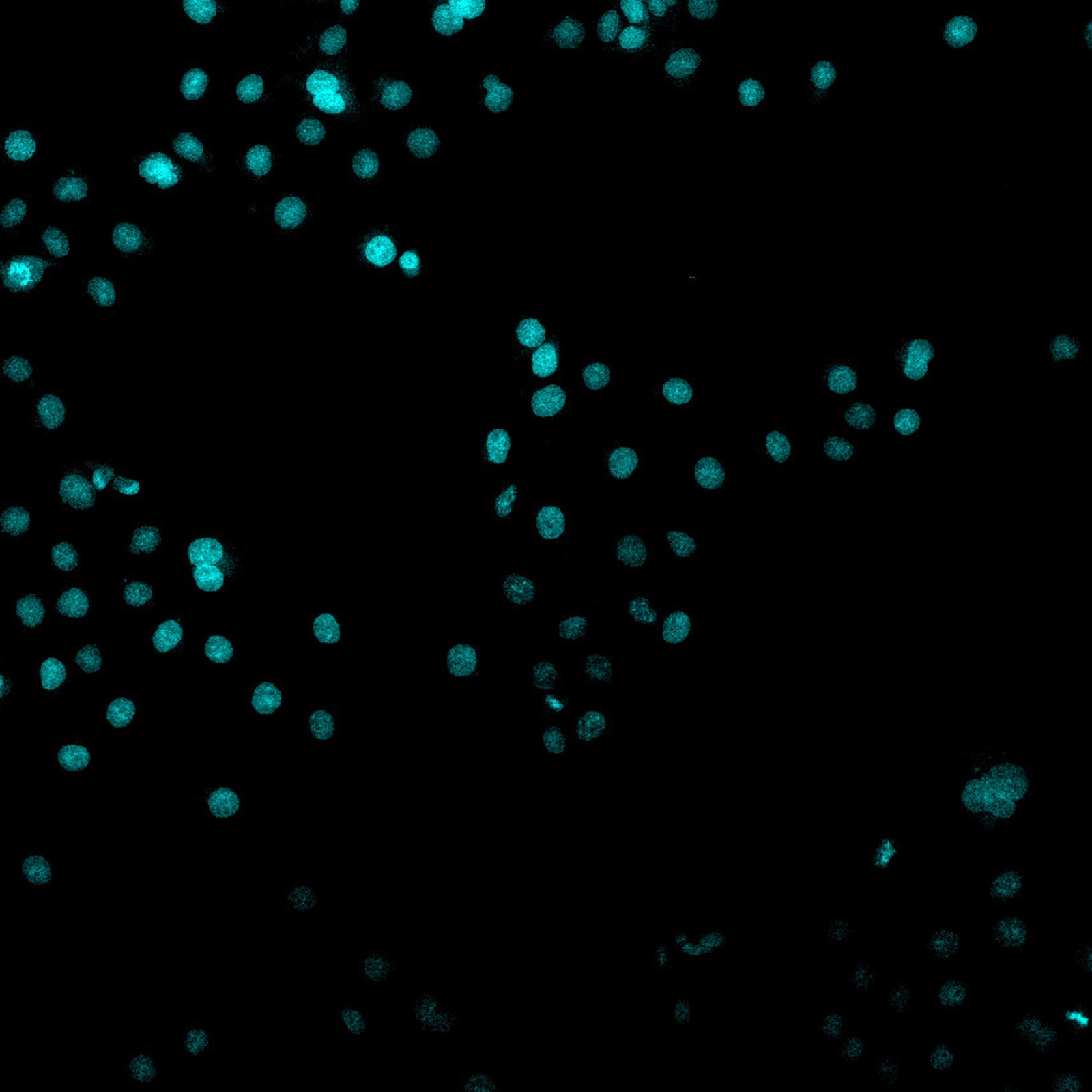
Abbildung 1: BPAE-Zellprobe mit mittels DAPI-Färbung sichtbar gemachten Zellkernen. Die Probe wurde mit dem konfokalen Mikroskop FLUOVIEW FV3000 bei 20facher Vergrößerung aufgenommen. Die Probe ist teilweise gebleicht, was zu unterschiedlichen Fluoreszenzsignalen führt. Um die Fähigkeiten der TruAI Technologie zu zeigen, wurde eine niedrige Integrationszeit gewählt.
Auch hier ist es sinnvoll, die TruAI Technologie einzusetzen, um verrauschte Zellkerne und Zellkerne über einen breiten Bereich von Signalpegeln zu segmentieren. Diese Standardanwendung ist mit der TruAI Technologie problemlos möglich (siehe Abbildung 2). Das Bild wird in zwei Klassen unterteilt: Vordergrund und Hintergrund. Die TruAI Technologie kann bei Bedarf bis zu 16 verschiedene Vordergrundklassen erkennen.
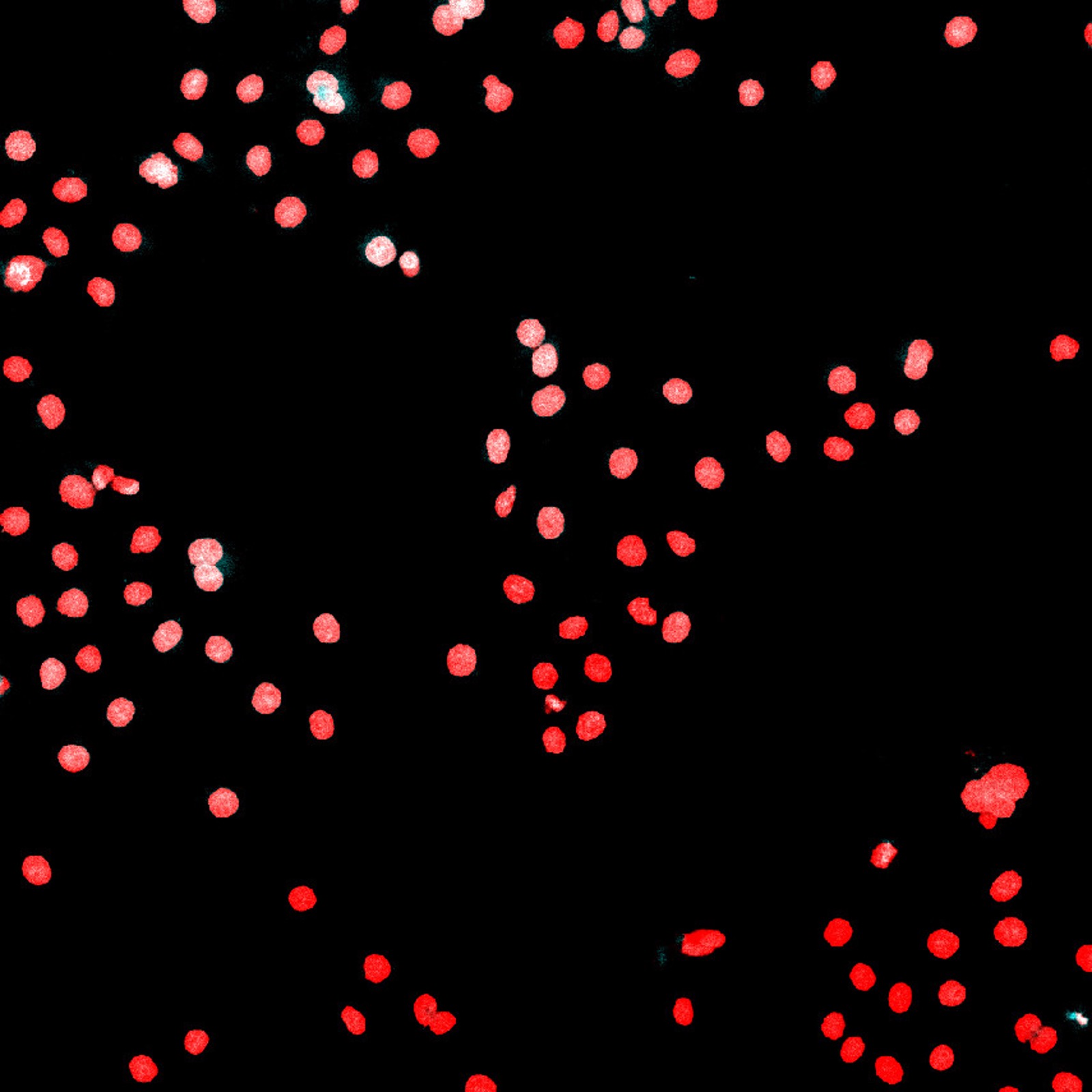
Abbildung 2: Das Bild aus Abbildung 1, überlagert mit der TruAI Wahrscheinlichkeitskarte, in der die Zellkernpixel rot dargestellt sind.
Zellkerne, die sich überlagern oder berühren, müssen unter Umständen in einfachen Nachbearbeitungsschritten in einzelne Zellkerne separiert werden, da das neuronale Netz alle Pixel entweder als Zellkern oder als Hintergrund erkennt. Wenn sich die Zellkerne berühren, gibt es keine Hintergrundpixel, um sie als separate Objekte zu unterscheiden. Die Segmentierung einzelner Zellkerne wird als Instanzsegmentierung bezeichnet.
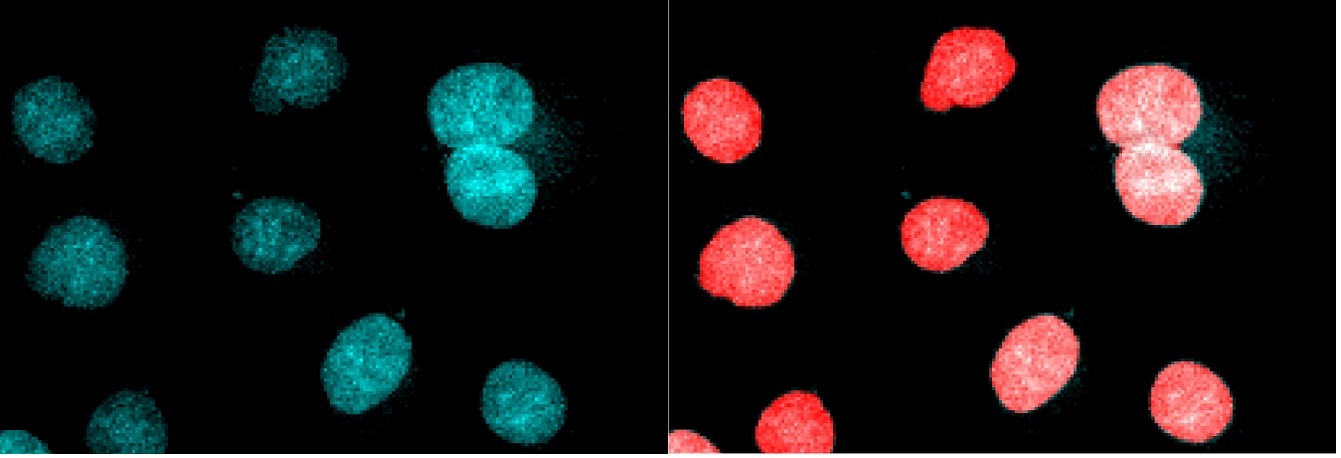
Abbildung 3: Detailansicht von Abbildung 1 (links) und Abbildung 2 (rechts). Die Segmentierung der Zellkernpixel ist sehr gut, aber sich berührende Zellkerne müssen durch Nachbearbeitungsschritte getrennt werden.
Die Segmentierung ist auch für komplexere Analyseaufgaben nützlich, z. B. bei konfluenten Zellen (siehe Abbildung 4). Anhand der Segmentierung kann beispielsweise eine von Zellen bedeckte Fläche gemessen werden.
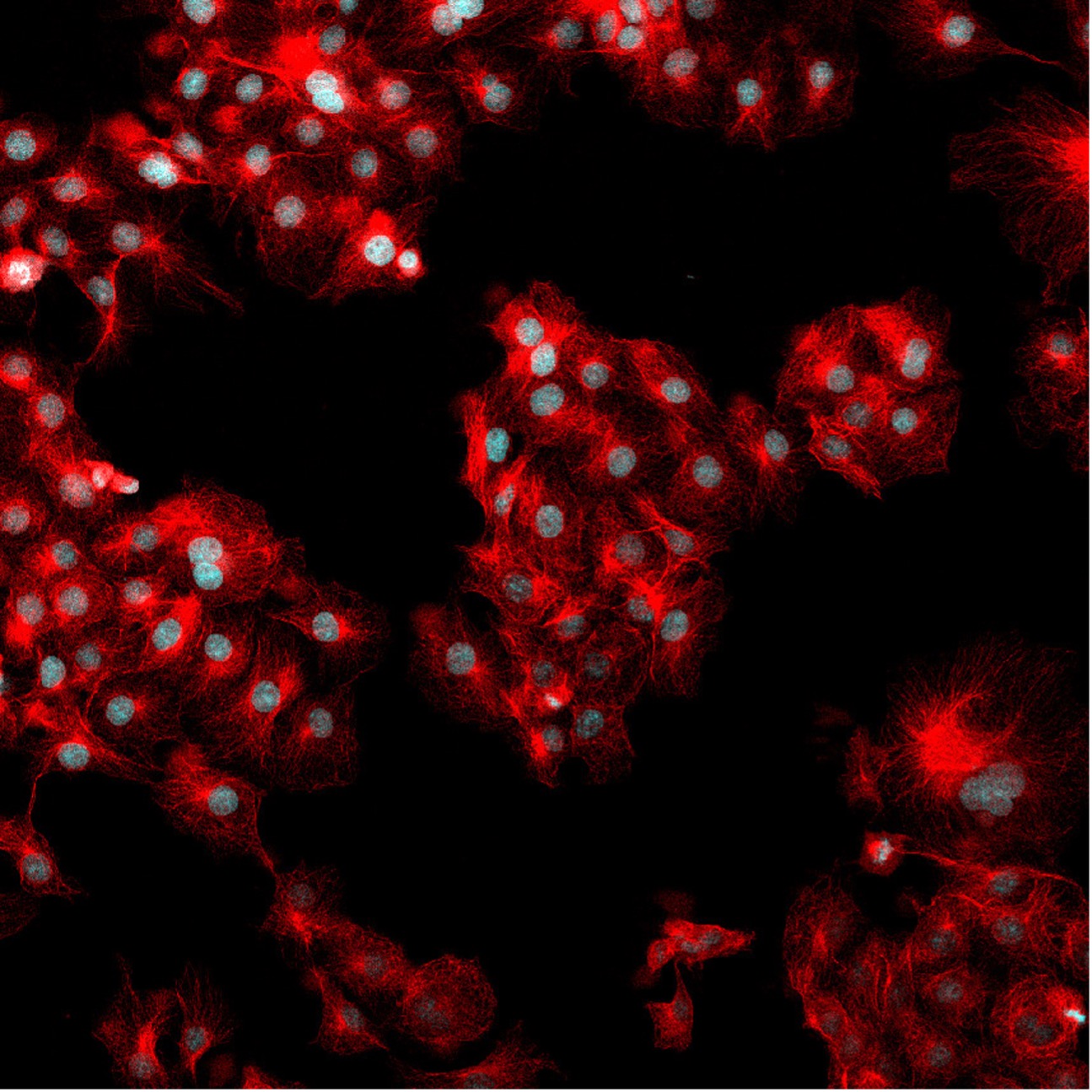
Abbildung 4: Dasselbe Sehfeld wie in Abbildung 1, aber jetzt auch mit dem Fluoreszenzsignal für 488 Phalloidin zur Markierung von F-Aktin.
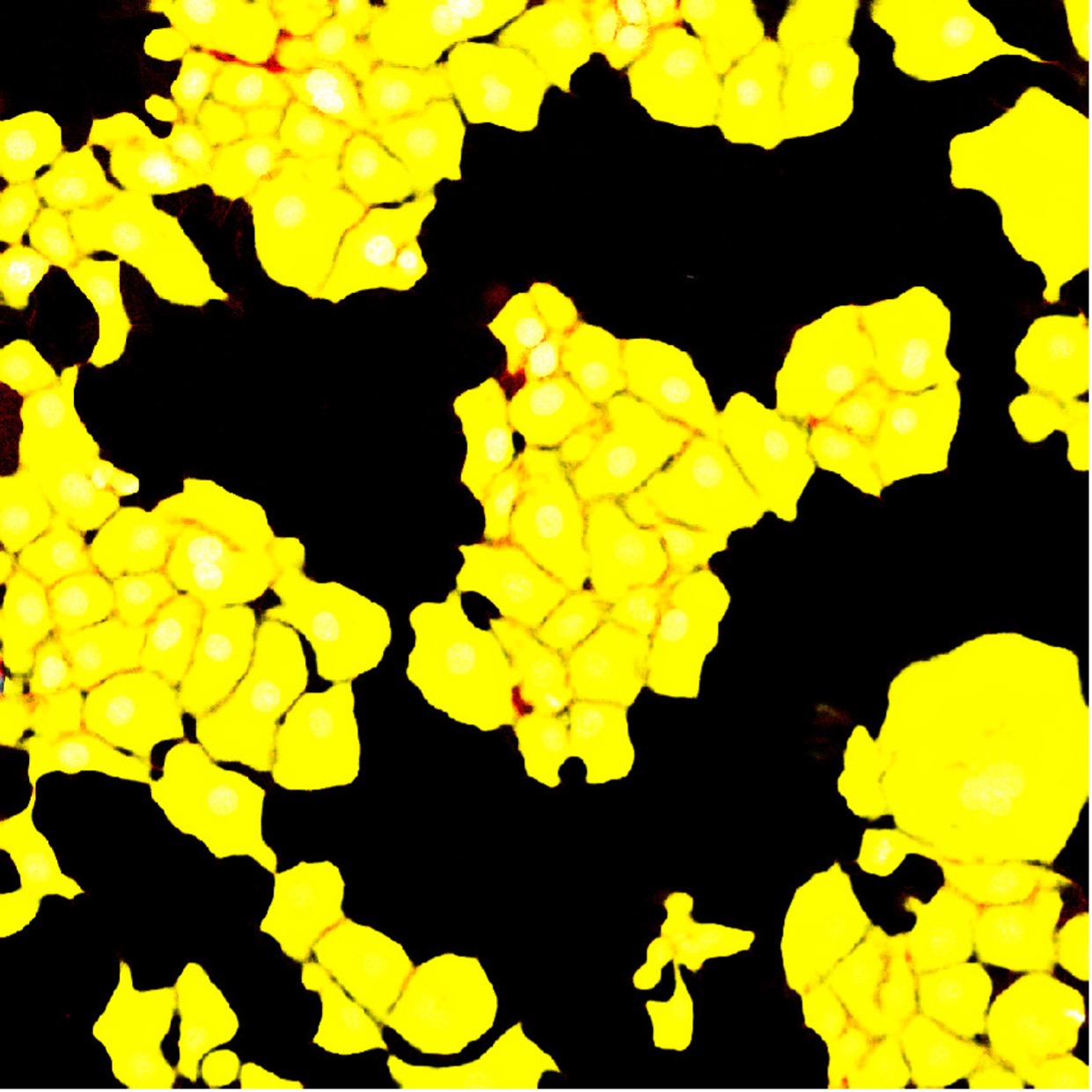
Abbildung 5: Das Ergebnis der Segmentierung des F-Aktin-Kanals mit einem neuronalen Standardnetz zur Segmentierung. Obwohl das neuronale Netz mit 1-Pixel-Grenzen zwischen sich berührenden Zellen trainiert wurde, kann das Segmentierungsergebnis einzelne Zellen nicht zuverlässig trennen.
Die Aufteilung des segmentierten Zellbereichs in einzelne Zellen (Instanzsegmentierung) kann jedoch eine aufwändige Nachbearbeitungsaufgabe verlangen (siehe Abbildung 6).
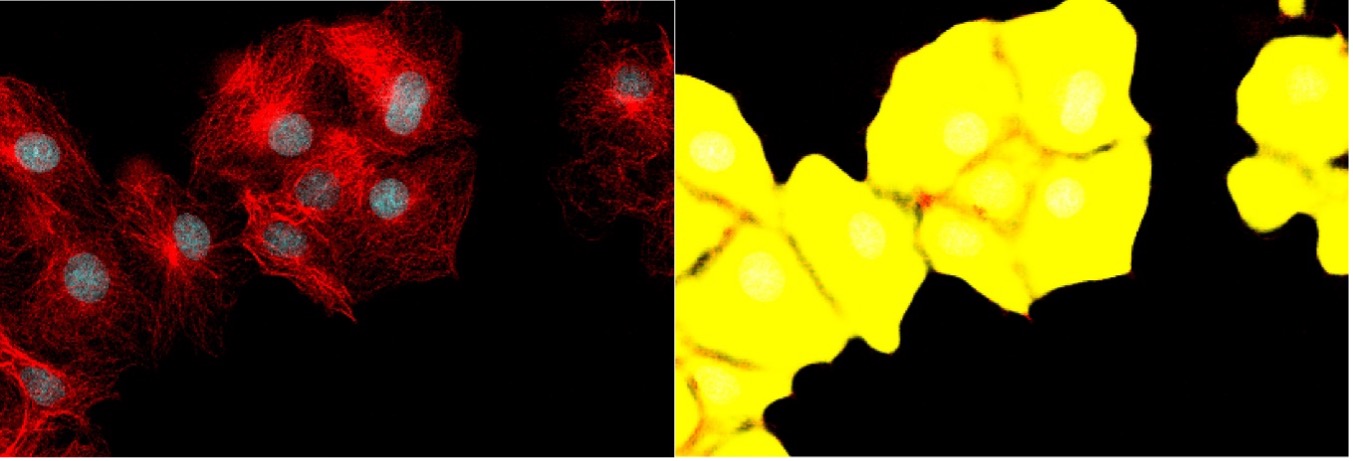
Abbildung 6: Detailansicht von Abbildung 4 (links) und Abbildung 5 (rechts). Die Grenzen zwischen benachbarten Zellen zeigen einen Abfall der Wahrscheinlichkeit für die Vordergrundklasse, aber eine Trennung der einzelnen Zellen würde eine komplexe Nachbearbeitung durch einen erfahrenen Benutzer erfordern.
Um diesen Arbeitsablauf für Bilder mit unterschiedlichster Komplexität zu vereinfachen, haben wir unsere TruAI Technologie für die scanR Screening-Station* um eine einfache Instanzsegmentierungsfunktion erweitert.
Die Instanzsegmentierung ist KI-basiert und erfordert keine manuellen Nachbearbeitungsschritte oder Anpassungen der Parameter. Sobald ein neuronales Netzmodell trainiert ist, kann es mit einem einzigen Klick auf neue Bilder angewendet werden, um sofortige Analyseergebnisse zu erhalten (siehe Abbildung 7).
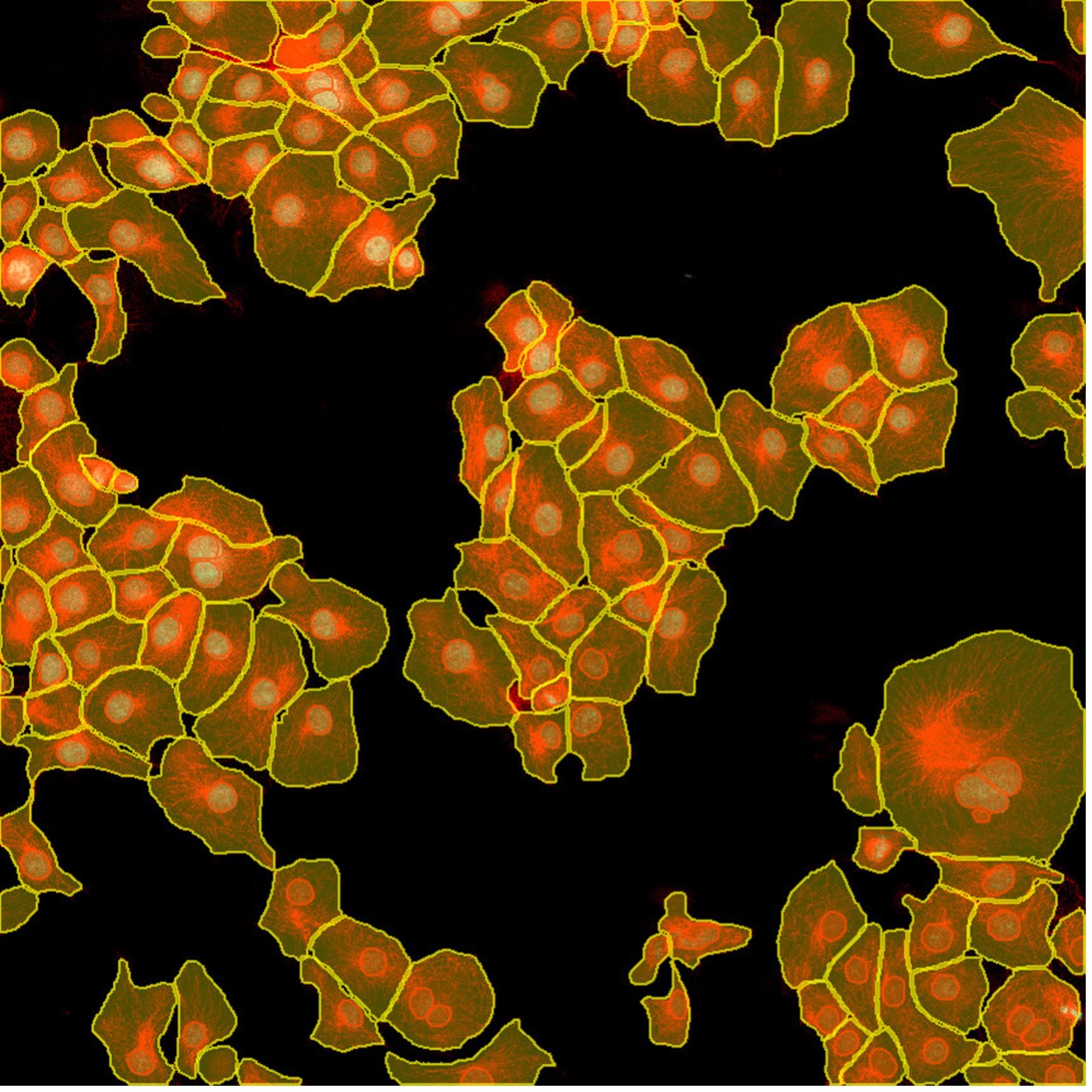
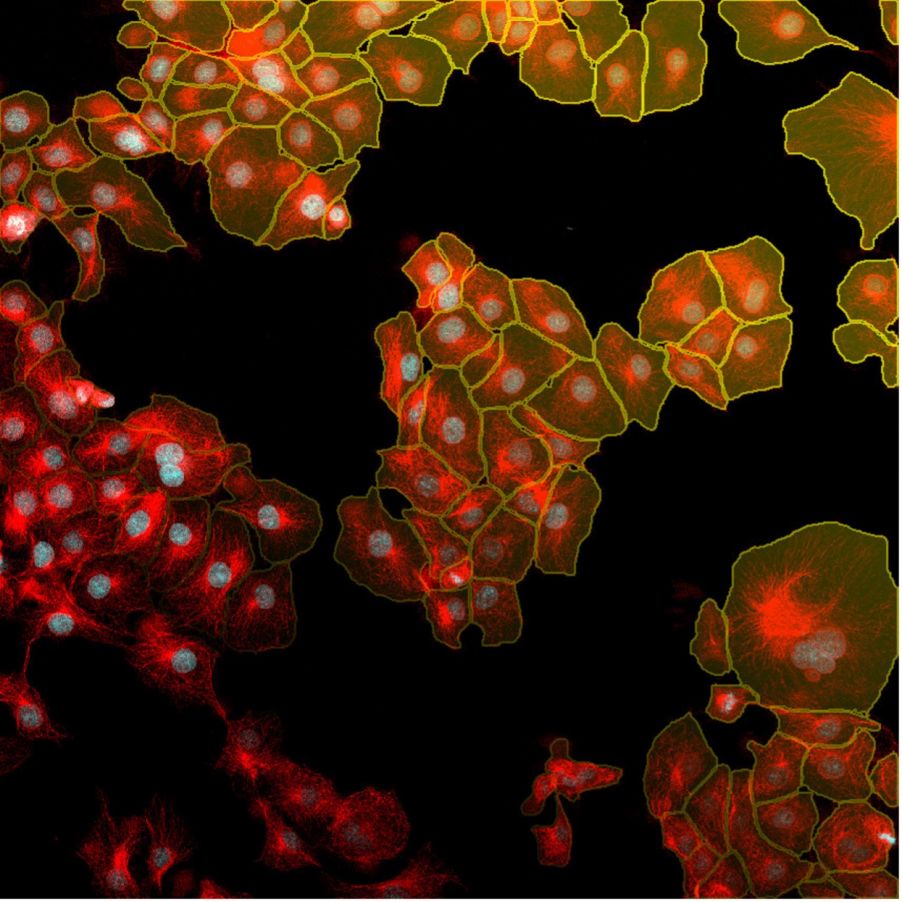
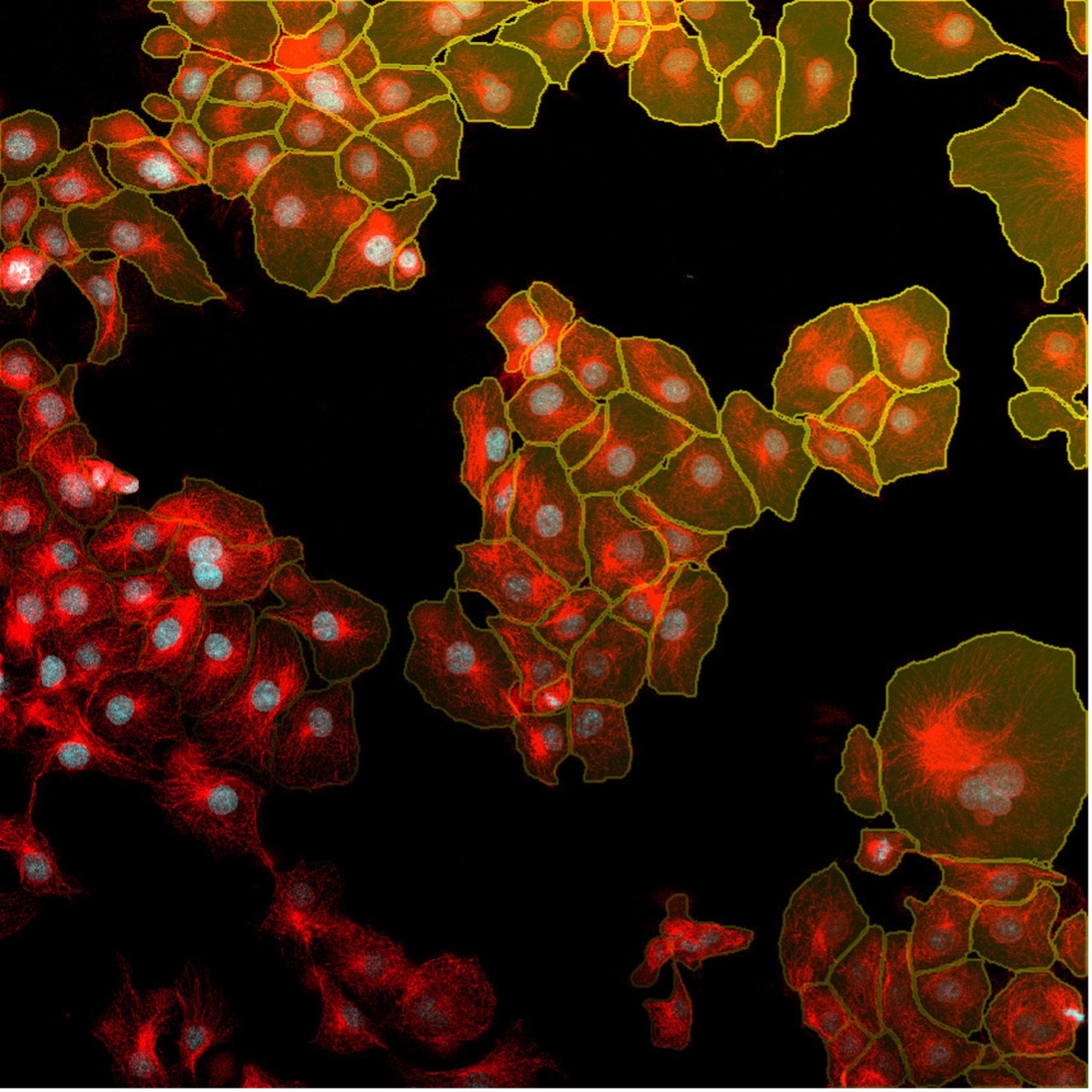
Abbildung 7: Mit der TruAI Instanzsegmentierung erhalten Sie die Ergebnisse für die Erkennung von Zellkernen (rot) und Zellen (gelb) mit nur einem Klick. Diese einfache Instanzsegmentierungsmethode erfordert keine Nachbearbeitungsschritte.
Abbildung 8 unten zeigt, wie die TruAI Instanzsegmentierung die Instanzen von Zellen und Zellkernen ohne weitere Nachbearbeitungsschritte direkt unterscheidet.
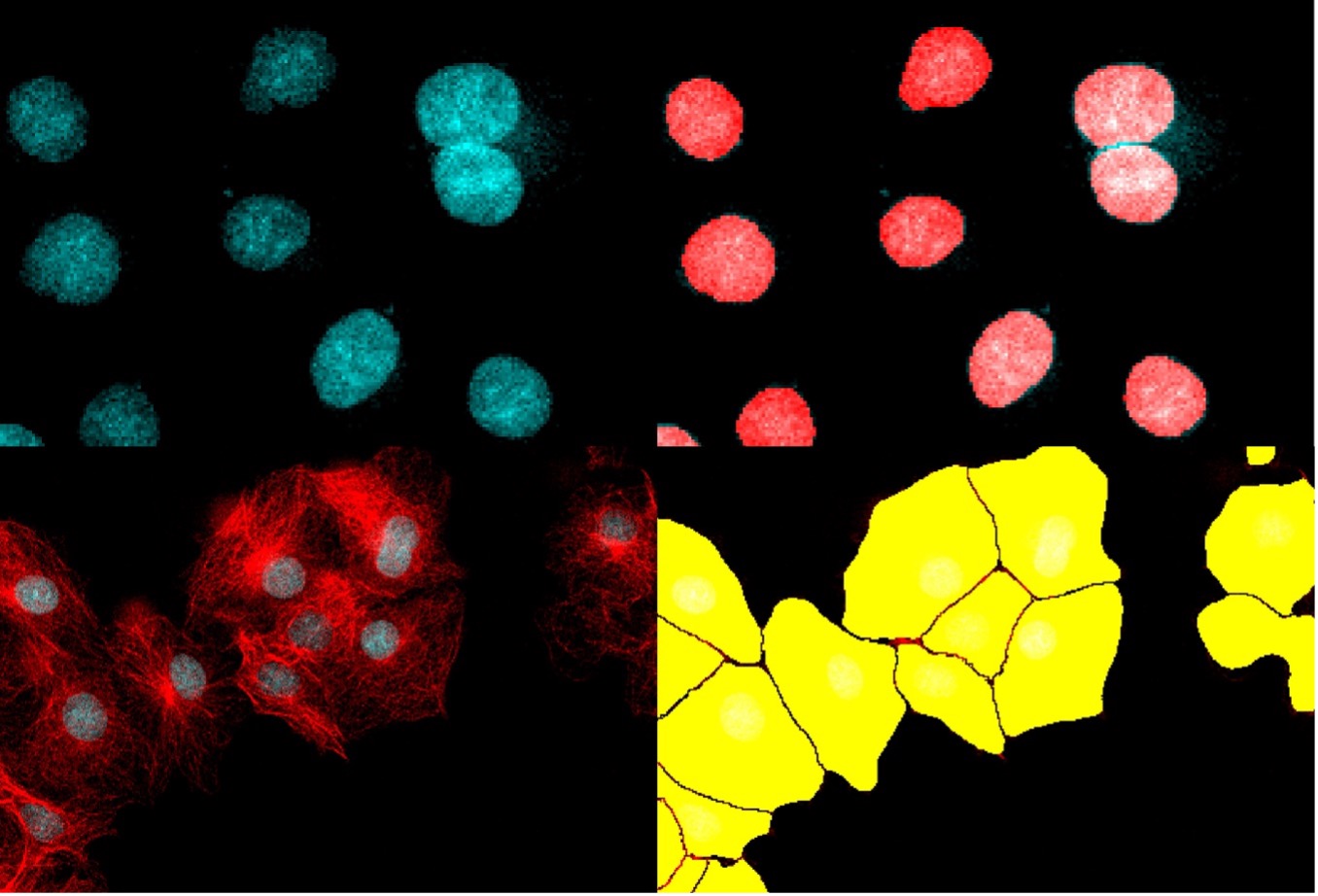
Abbildung 8: Mithilfe der TruAI Instanzsegmentierung können die neuronalen Netze Zellkerne (oben) und Zellen (unten) direkt unterscheiden.
In Abbildung 9 unten werden die Ergebnisse der Zelltrennung mit der Standardsegmentierung und der TruAI Instanzsegmentierung miteinander verglichen.
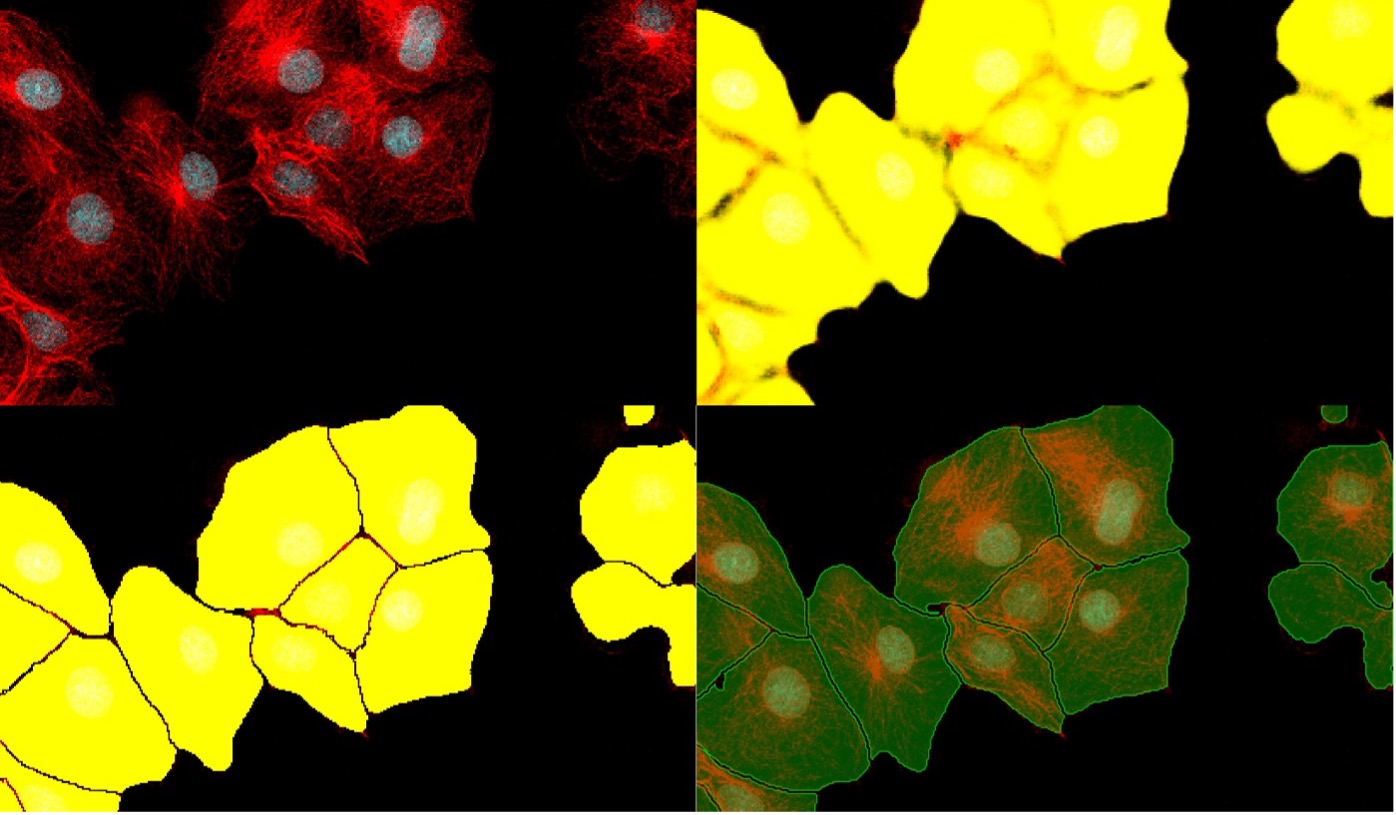
Abbildung 9: Entwicklung von der Standardsegmentierung (oben rechts) zur TruAI Instanzsegmentierung (unten links) bei der Analyse komplexer Bilder (oben links). Das Ergebnis (unten rechts) ist eine nahezu perfekte Trennung der Zellen.
KI-gestützte Segmentierung: Ein leistungsfähiges Tool für die Analyse von Mikroskopiebildern
Unsere aktualisierte TruAI Technologie bietet ein leistungsstarkes Tool zum Trainieren individueller neuronaler Netzmodelle für unterschiedlichste Bildanalyseanwendungen. Für das Training eines optimalen neuronalen Netzes sind zwar sorgfältig beschriftete Beispiele erforderlich (was durch unsere benutzerfreundliche Software unterstützt wird), die Anwendung des trainierten neuronalen Netzes auf neue Bilder ist jedoch einfach und zuverlässig.
Dank der TruAI Instanzsegmentierung sind auch zuverlässige vortrainierte Deep-Learning-Modelle für Standardanwendungen wie die Erkennung von Zellkernen oder Zellen möglich. In einem der nächsten Beiträge werden Sie mehr über die vortrainierten Modelle erfahren und Beispiele sehen.
Informieren Sie sich bis dahin über andere Anwendungen zur Instanzsegmentierung:
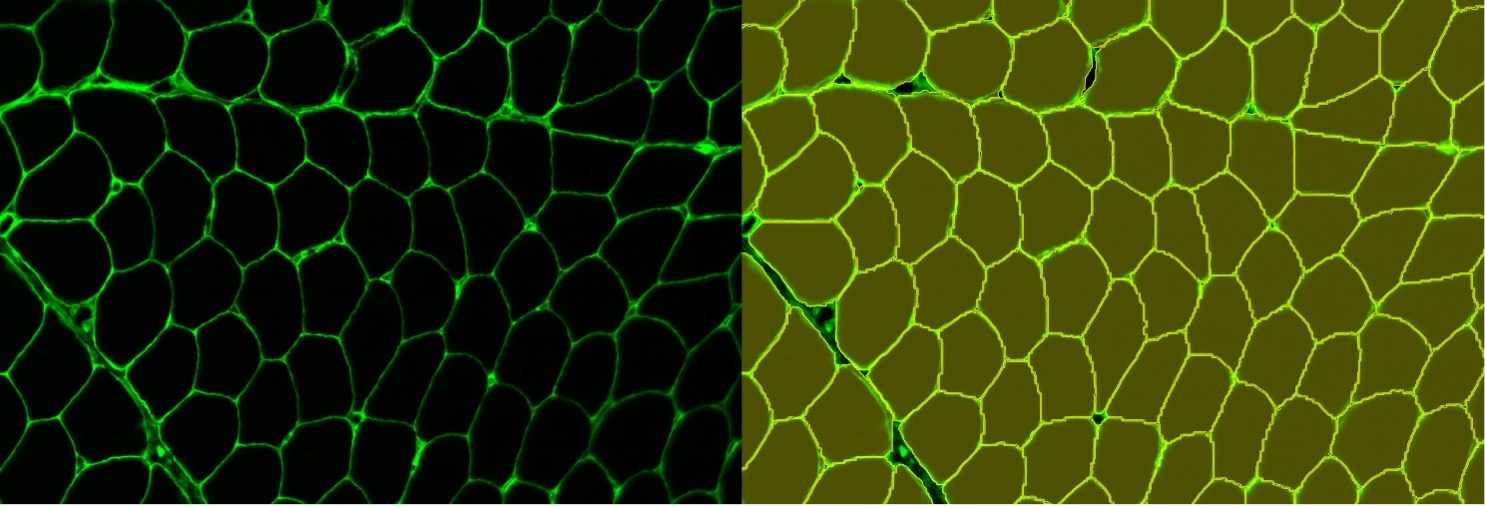
Abbildung 10: Instanzsegmentierung von gefärbten Muskelfasern
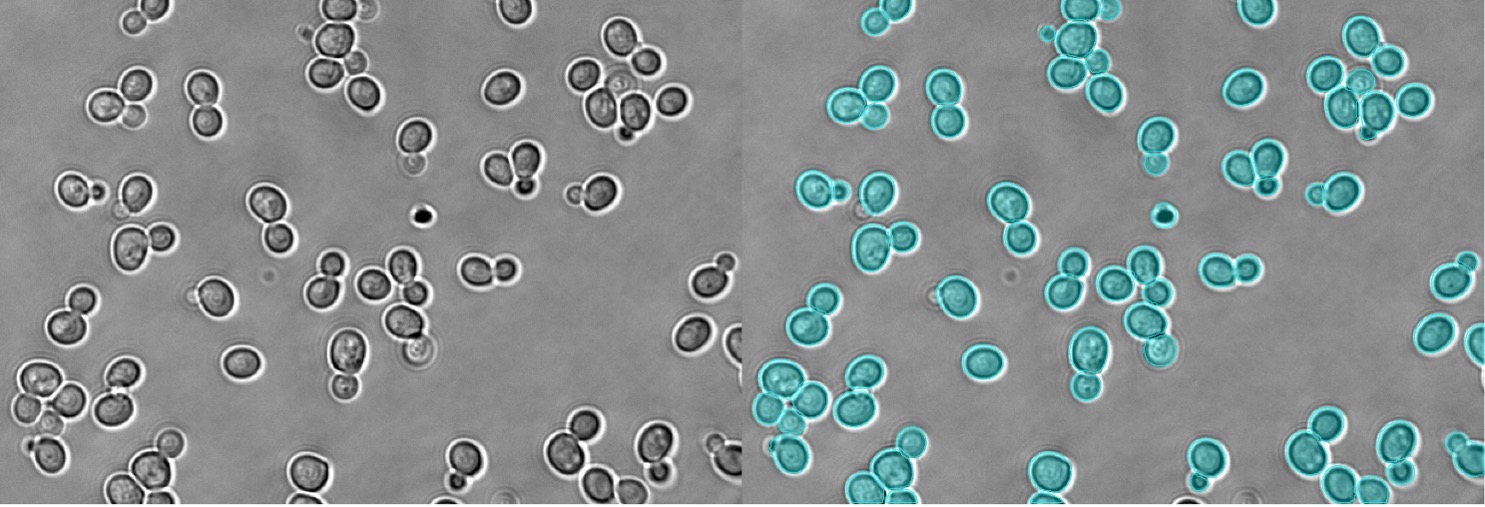
Abbildung 11: Instanzsegmentierung von Hefen im Hellfeld
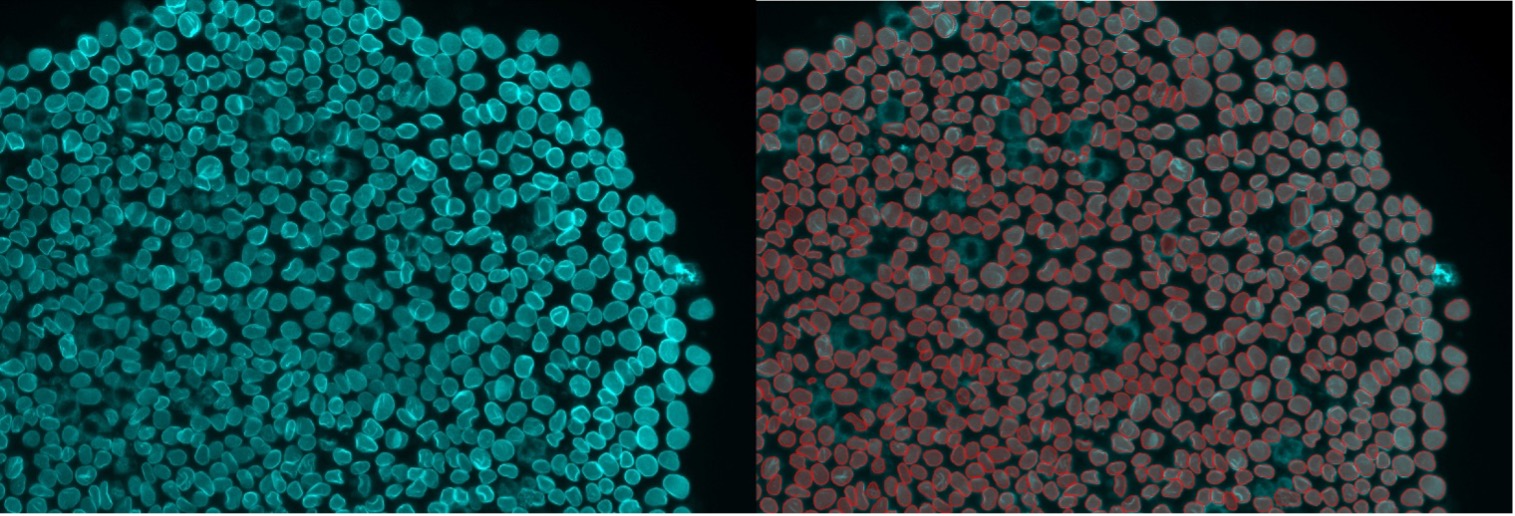
Abbildung 12: Instanzsegmentierung von gefärbten Zellkernhüllen
*Das scanR System ist in Japan nicht verfügbar.
Ähnliche Artikel
Video: Die Leistungsfähigkeit von Deep Learning für die selbstlernende Mikroskopie
Fortschrittliche Lebendzellanalyse mit KI-gesteuerten High-Content-Screening-Systemen
Markierungsfreie Quantifizierung, auf die man sich verlassen kann: Ein Experiment mit Deep Learning