Procédure d’analyse 3D des tests de viabilité cellulaire
Résumé
Le logiciel d’analyse cellulaire 3D NoviSight fournit des données statistiques sur les sphéroïdes et tout autre objet 3D en mesurant des paramètres tels que le volume, la sphéricité et le nombre de cellules. Cette note d’application a pour objectif de présenter la procédure d’analyse cellulaire 3D NoviSight des tests de viabilité cellulaire sur des sphéroïdes.
Introduction
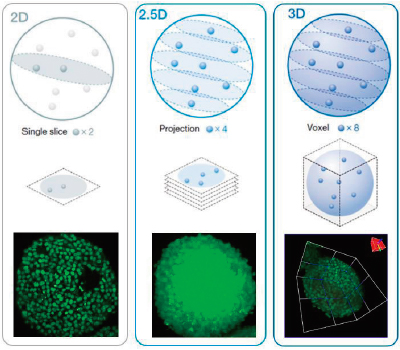
L’analyse 3D à haut débit sur des sphéroïdes ou des organoïdes peut s’avérer compliquée. Dans l’idéal, nous pouvons analyser des échantillons 3D afin d’acquérir des informations sur leur morphologie et leur état interne. L’analyse 3D du logiciel NoviSight permet de simplifier ce processus en mesurant le volume, le nombre de cellules et l’état des cellules d’échantillons 3D à partir d’un empilement d’images en fluorescence de plans Z. L’analyse conventionnelle quant à elle fait appel à une seule image (figure 1, gauche, analyse 2D) ou à une projection d’images (figure 1, centre, analyse 2,5 D). Ces deux techniques entraînent une perte importante de données de l’échantillon 3D, notamment les informations spatiales, morphologiques et volumétriques (figure 1). Dans cette note d’application, nous vous expliquerons comment analyser de manière quantitative des échantillons 3D avec le logiciel NoviSight en utilisant test de viabilité des cellules de sphéroïdes. |
Figure 1 (de gauche à droite) : représentations schématiques des analyses 2D, 2,5D et 3D. |
Méthodes
Préparation des échantillons
Nous avons mis en culture 500 cellules HT-29 par puits d’une microplaque à 96 puits PrimeSurface® (Sumitomo Bakelite) et les avons cultivées pendant huit jours afin de former des sphéroïdes, puis nous avons appliqué différentes concentrations de staurosporine (STS) ainsi qu’une sonde NucView® 550 (Biotium) le jour suivant. Le substrat NucView 550 de la caspase-3 est une sonde fluorescente rouge utilisée pour marquer le noyau des cellules mortes. Après l’application de la STS et du NucView 550, les sphéroïdes de cellules HT-29 ont été fixés avec paraformaldéhyde à 4 % et perméabilisés avec 0,5 % Triton X-100 dans du tampon phosphate salin(-). Les noyaux des sphéroïdes ont été marqués au Hoechst 33342 à 4 °C (39,2 °F) pendant une nuit. Une fois le marquage terminé, les sphéroïdes ont été clarifiés avec du SCALEVIEW-S4.
Imagerie et analyse
Nous avons pris des images des échantillons en trois dimensions à l’aide du microscope confocal FV3000 et d’un objectif semi-apochromatique (LUCPLFLN20X). Vous pouvez consulter les conditions d’acquisition ci-dessous. Le pas de déplacement sur l’axe Z était de 2 µm. Un laser de 405 nm a été utilisé pour fluophore Hoechst 33342 (fluorescence bleue) tandis qu’un laser de 561 nm a été utilisé pour la sonde NucView
550 (fluorescence magenta). Les images volumétriques des sphéroïdes traités à la STS et non traités acquises avec le microscope sont présentées dans la figure 2. Le centre du sphéroïde non traité cultivé pendant 8 jours était mort probablement à cause d’une carence nutritionnelle ou d’une hypoxie. Dans l’ensemble, la STS a induit la mort des cellules des sphéroïdes HT-29.
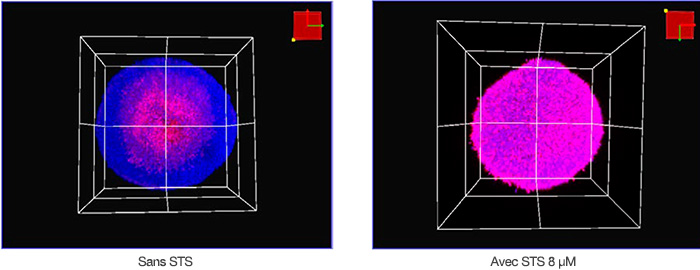
Figure 2 : représentation volumétrique des sphéroïdes de cellules HT-29 traités à la STS (à droite) et non traités (à gauche).
Avantages
- Réaliser facilement un test de viabilité des cellules 3D
- L’interface utilisateur intuitive vous aide à transcrire l’empilement d’images de plans Z en données quantitatives reproductibles
Stratégie d’analyse 3D de la viabilité des cellules
Les étapes décrites dans le tableau 1 sont requises pour analyser de manière quantitative la viabilité des cellules de sphéroïdes avec le logiciel NoviSight™. Pour permettre au logiciel NoviSight d’identifier en trois dimensions les noyaux à partir d’images de plans Z, sélectionnez un pas de déplacement du plan Z de 3 µm, soit le pas la plus large pour détecter un seul noyau.
| Étape | Détails |
| Préparation des échantillons |
Culture cellulaire et marquage par fluorescence
Option : fixation et clarification Si vous souhaitez évaluer le nombre de cellules, vous devez marquer les noyaux ↓ |
| Imagerie |
Acquisition de plusieurs images selon l’axe Z
ex. microscopie confocale, microscopie multiphotonique ↓ |
| Analyse |
1. Identifier les objets qui vous intéressent, par ex., les noyaux, le volume, les organites
2. Définir les paramètres d’analyse 3. Analyse graphique par fenêtrage 4. Analyse statistique |
Tableau 1 : Procédure d’analyse 3D depuis la préparation des échantillons jusqu’à l’analyse.
Étapes de l’analyse du logiciel NoviSight
Identification des objets principaux
La viabilité des cellules a été évaluée en fonction du rapport entre le nombre de cellules mortes et le nombre de cellules vivantes. Le logiciel NoviSight détecte le nombre de cellules grâce au marquage des noyaux. Pour identifier les noyaux cellulaires, nous avons utilisé le module spécial de détection des noyaux NoviSight : NuclearL. Lorsque vous saisissez la taille approximative des cellules, l’intensité du bruit de fond ainsi que le rapport Z/XY (allongement sur l’axe Z du noyau), le logiciel NoviSight est capable de détecter automatiquement le noyau des cellules des sphéroïdes en 3D (figure 3A).
Définition des paramètres d’analyse
Parmi tous les noyaux marqués au Hoechst 33342, ceux présentant un signal NucView 550 de forte intensité étaient des noyaux de cellules mortes. Le logiciel NoviSight est donc capable de classer les cellules mortes et vivantes en fonction de l’intensité des signaux NucView 550 détectés par le canal 2 des noyaux identifiés (figure 3B). Cela signifie que le logiciel NoviSight peut classer les cellules du sphéroïde en cellules mortes ou vivantes en utilisant simplement le paramètre d’identification du noyau.
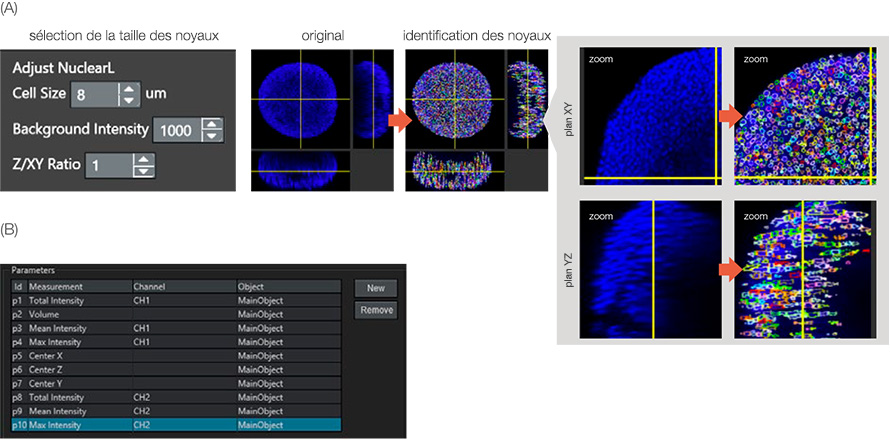
Figure 3 : Identification des objets parents. (A) Les noyaux ont été identifiés par le module NuclearL. Chaque noyau identifié a été entouré d’un cercle de couleur. (B) Les paramètres d’analyse ont été définis. Par exemple, le paramètre 10 correspond à l’intensité maximale (signal des cellules mortes) sur le canal 2 (CH2) de l’objet principal (noyau).
Une fois l’identification des noyaux terminée, les noyaux identifiés peuvent être représentés en tant que données d’objet sur un diagramme de dispersion ou un histogramme. L’axe X ou Y du diagramme de dispersion peut être ajusté à partir des paramètres d’analyse. Lorsque vous les définissez respectivement sur le centre X/Y, les centres X/Y des objets principaux sont représentés sur le graphique (figure 4A).
Lorsque vous cliquez sur un point sur le graphique, le logiciel NoviSight peut indiquer la position correspondante sur l’image originale (figure 4B).
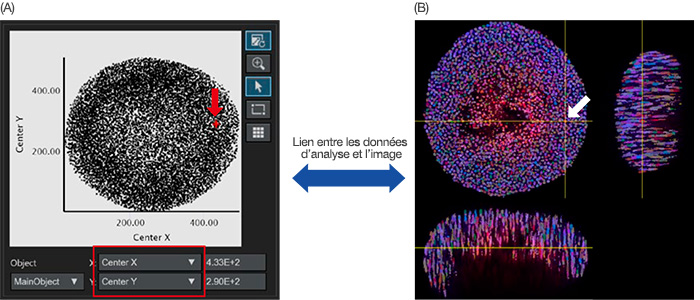
Fugure 4 : image de l’analyse quantitative d’un sphéroïde. (A) Lorsque les paramètres d’analyse du centre X et du centre Y (définis à la figure 3B) ont été définis, respectivement, sur l’axe X et l’axe Y, chaque noyau identifié a été représenté sur le graphique. Les paramètres d’analyse sur les axes X/Y peuvent être modifiés si nécessaire (carré rouge). (A/B) Le
logiciel NoviSight peut relier visuellement les données d’analyse aux images originale. Lorsque vous cliquez sur le tracé d’un noyau identifié sur le graphique (A, flèche rouge) la position correspondante de ce noyau identifié s’affiche automatiquement sur l’image originale (flèche blanche).
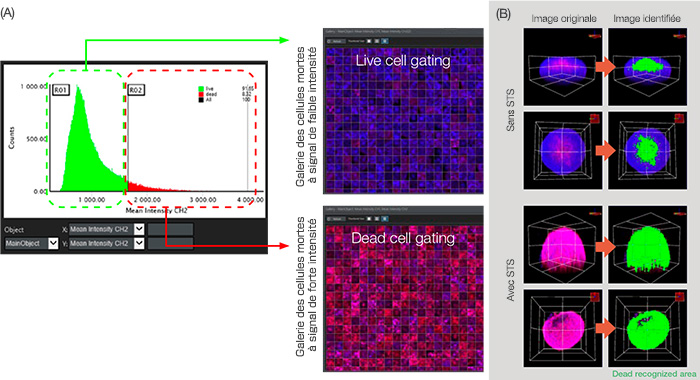
Figure 5 : Analyse graphique de la classification des cellules.
(A) Lorsque le paramètre d’analyse de l’intensité moyenne sur le CH2 (défini à la figure 3B) a été défini, respectivement, sur l’axe X et l’axe Y, le graphique le nombre d’objets principaux (noyaux) en fonction de l’intensité moyenne sur le CH2 (signal des cellules mortes). Grâce à l’analyse par fenêtrage des intensités des signaux détectés sur le CH2, nous avons pu séparer les
cellules mortes des cellules vivantes. (B) Représentation volumétrique de la zone morte identifiée en vert.
Analyse statistique
Le logiciel NoviSight est capable d’analyser simultanément de nombreux échantillons. Les données quantitatives issues de l’analyse peuvent être affichées sous la forme d’une carte de densité (figure 6A) ou exportées au format CSV pour une analyse plus poussée. Les données peuvent également être lues pour chaque fenêtrage. Dans le cas présent, les données montrent que la STS a induit l’apoptose des cellules des sphéroïdes HT-29 de manière proportionnelle à la dose(figure 6B).
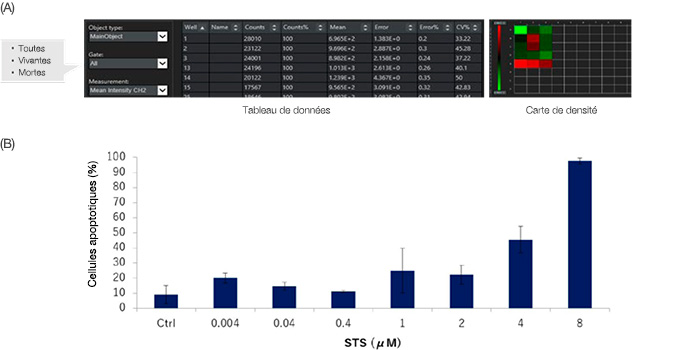
Figure 6 : Résultats de la quantification de l’analyse de la viabilité des cellules de sphéroïdes HT-29 traités ou non à la STS. (A) Le logiciel NoviSight permet de lire les données de quantification pour chaque fenêtrage.
Les données peuvent également être affichées sur une carte de densité. (B) La STS a induit la mort cellulaire des cellules HT-29, et ce, proportionnellement à la dose.
Lier les données aux images
Un des avantages clés de l’analyse à haut contenu est le lien entre les données et les images. Contrairement à un lecteur de microplaques, l’analyse à haut contenu permet de référencer l’image originale afin de vérifier visuellement la morphologie, la position et l’intensité des cellules. Le logiciel NoviSight™ tire pleinement profit de cette capacité. Par exemple, le logiciel est capable d’afficher l’image correspondante lorsque vous cliquez sur un point de données spécifique ou un objet (figure 7) tel que :
- Un point sur un diagramme de dispersion
- Une image de l’objet dans la galerie
- Un puits sur une carte de densité
- Un nombre dans un tableau
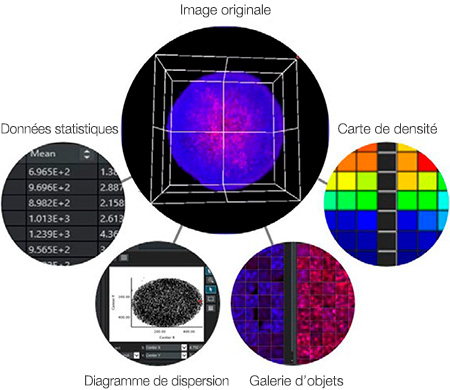
Figure 7 : Le logiciel NoviSight permet d’afficher l’image correspondant à un point de données ou un objet lorsque vous cliquez sur ce point de données ou cet objet.
Conclusion
L’analyse quantitative 3D d’échantillons 3D peut s’avérer compliquée. Heureusement, le logiciel NoviSight permet de simplifier ce processus de bout en bout. Par exemple, l’identification des noyaux constitue le seul paramètre nécessaire pour l’analyse de la viabilité des cellules de sphéroïdes. Cette procédure simplifiée s’applique également à tout autre modèle 3D pertinent.
Auteur
Mayu Ogawa (Olympus)
Produits utilisés pour cette application
a bien été ajouté à vos favoris
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
Not Available in Your Country
Sorry, this page is not
available in your country.