Visualisation des protéines de réparation de l’ADN avec le microscope confocal FV3000
Imagerie de cellules U2OS vivantes après des lésions de l’ADN provoquées par un laser
Les ruptures bicaténaires de l’ADN constituent l’une des formes de dégâts causés à l’ADN les plus nocives. En réaction aux dégâts, les voies de réponse aux dommages de l’ADN (DDR, pour DNA Damage Response) dans la cellule se déclenchent et entraînent la mobilisation de facteurs DDR sur le site de rupture, ainsi que la signalisation des points de contrôle du cycle cellulaire et la régulation des activités de réparation de l’ADN. Une signalisation et une réparation immédiates et précises du site de rupture sont essentielles à la viabilité de la cellule et pour empêcher les mutations pouvant aboutir au développement d’un cancer. Par conséquent, la compréhension des mécanismes intervenant dans le processus de réparation de l’ADN est d’une importance vitale. Dans cette application, nous avons utilisé des cellules épithéliales cancéreuses d’ostéosarcome humain (U2OS) pour étudier le recrutement des protéines de réparation de l’ADN après des dégâts provoqués par un laser, notamment des ruptures bicaténaires de l’ADN, à l’aide du microscope confocal FV3000. Les images ainsi obtenues nous ont permis (1) de déterminer les propriétés cinétiques et les niveaux d’accumulation des protéines de réparation sur les sites de rupture, et (2) de caractériser la colocalisation des régulateurs de transcription endogènes et les facteurs des voies DDR sur les sites de rupture d’ADN.
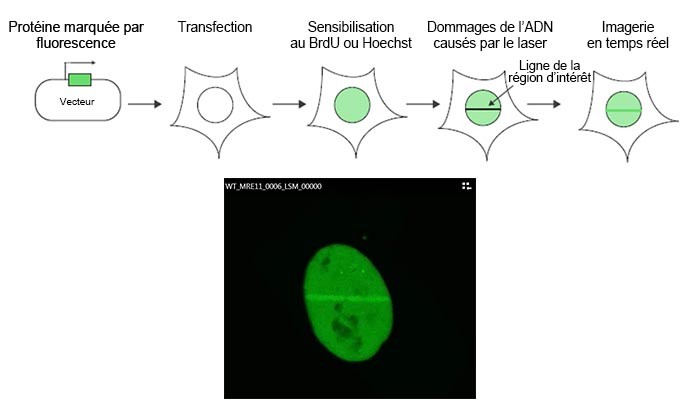
Figure 1 : Schéma du protocole expérimental
L’ADNc de MRE11 a été cloné dans un vecteur d’expression marqué à la GFP, qui a ensuite transféré dans des cellules U2OS. Après une sensibilisation par bromodéoxyuridine (BrdU) ou Hoechst, on induit des lésions de l’ADN par un balayage linéaire de la région d’intérêt dans le noyau à l’aide du laser de 405 nm du microscope FV3000. On réalise alors une imagerie des cellules vivantes à l’aide du laser de 488 nm afin de suivre la cinétique du recrutement de la protéine MRE11 pour répondre aux dégâts causés par le laser.
Conditions d’imagerie
Objectif : objectif à immersion dans l’huile super-corrigé X60 (PLAPON60XOSC)
Microscope : Microscope confocal à balayage laser FLUOVIEW FV3000
Lasers : 405 nm (stimulation de la région d’intérêt), 488 nm (GFP, vert)
Imagerie de cellules vivantes sûre pour les échantillons en vue de réaliser des mesures
L’exposition répétée à la lumière d’excitation laser au cours d’une imagerie par intervalles de temps réguliers peut provoquer un photoblanchiment et la phototoxicité, ce qui a des conséquences sur la capacité à obtenir des mesures de données quantitatives lors de l’expérience. Dans notre protocole, nous avions besoin de trouver un compromis entre une stimulation laser puissante et une imagerie de cellules vivantes sûre pour les échantillons, afin de capturer de manière quantifiable la dynamique des protéines de réparation immédiatement après des dégâts causés à l’ADN par le laser. Pour cela, nous avons utilisé le microscope confocal FV3000 qui, grâce à la technologie de détection TruSpectral d’Olympus et aux détecteurs GaAsp à sensibilité élevée, minimise la puissance laser requise pour l’imagerie continue des cellules vivantes. De plus, nous avons utilisé TruFocus pour maintenir la mise au point tout au long de l’expérience d’imagerie. Ensemble, ces technologies nous ont permis d’obtenir des données par intervalles de temps réguliers précises, que nous avons pu quantifier pour déterminer l’accumulation du facteur DDR MRE11 sur les sites de rupture d’ADN à la suite de lésions.
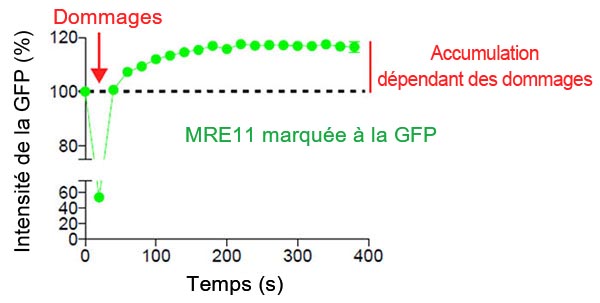 |
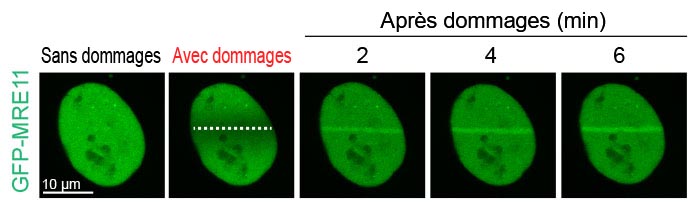
Figure 2 : Accumulation de MRE11 sur le site de rupture de l’ADN à la suite de lésions
Des cellules U2OS exprimant la protéine MRE11 marquée à la GFP ont été soumises à des lésions de l’ADN causées par un laser de 405 nm. Une imagerie de la GFP par intervalles de temps réguliers a ensuite été réalisée avec le laser de 488 nm. Les images des cellules ont été enregistrées pendant 6 minutes à intervalles de 20 secondes afin de visualiser et de quantifier la localisation de MRE11 avant et après les lésions de l’ADN.
L’objectif super-corrigé permet une analyse précise de la colocalisation
En plus d’étudier les paramètres cinétiques du recrutement de MRE11 sur les sites de rupture des brins d’ADN, nous avons également examiné les réponses de la protéine γH2AX, une histone qui est phosphorylée au niveau des ruptures bicaténaires de l’ADN et qui active les voies DDR, ainsi que de CHD4, une protéine qui joue un rôle important dans la régulation transcriptionnelle épigénétique. Pour réaliser des études de colocalisation précises, il est important de minimiser les aberrations chromatiques qui peuvent provoquer un glissement latéral significatif des différents canaux d’acquisition. Pour minimiser les effets des aberrations chromatiques dans nos études de colocalisation, nous avons utilisé l’objectif Olympus PLAPON60XOSC2 avec super-correction des aberrations chromatiques afin d’obtenir des images de colocalisation fiables présentant des aberrations chromatiques latérales et axiales extrêmement faibles. Cela nous a permis de déterminer que CHD4 et γH2AX colocalisent sur les sites de lésions de l’ADN dans le noyau.
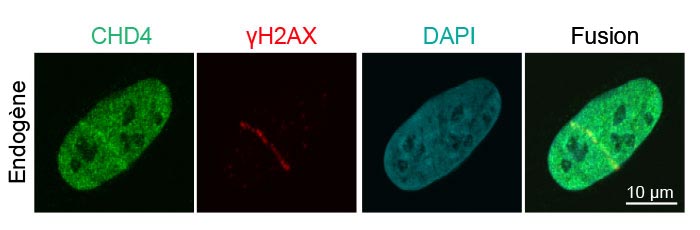
Figure 3 : Recrutement de protéines endogènes de réparation des lésions de l’ADN lors des ruptures des brins d’ADN
Détection des protéines endogènes CHD4 (vert, Alexa Fluor 488) et γH2AX (rouge, Alexa Fluor 594) dans des cellules épithéliales cancéreuses U2OS d’ostéosarcome humain après des lésions de l’ADN causées par un laser. L’image fusionnée représente la colocalisation des protéines CHD4 et γH2AX.
L’apport du microscope confocal FV3000 pour notre expérience
Le système entièrement spectral avec détecteurs GaAsP haute efficacité offre la grande sensibilité nécessaire à l’imagerie des cellules vivantes
Le microscope FV3000 repose sur la technologie de détection TruSpectral d’Olympus, qui diffracte la lumière en la transmettant à travers une unité holographique de phase volumique. Cette technologie permet d’obtenir une luminosité largement supérieure par rapport aux unités de détection spectrale conventionnelles équipées de réseaux de type à réflexion. Le détecteur haute sensibilité (HSD) à deux canaux du microscope FV3000 utilise la technologie TruSpectral avec des photomultiplicateurs GaAsP refroidis par effet Peltier pour une efficacité quantique élevée (45 %) et un rapport signal/bruit élevé. L’association de ces technologies de détection permet une détection haute sensibilité et minimise la puissance laser requise pour l’observation de tissus vivants.
Objectif super-corrigé PLAPON60XOSC2 pour une analyse de colocalisation fiable avec peu d’aberrations chromatiques
Cet objectif à immersion dans l’huile minimise les aberrations chromatiques latérales et axiales sur le spectre de 405 à 650 nm. Les images de colocalisation sont acquises de manière fiable, et les images sont mesurées avec une précision de position supérieure. L’objectif compense également les aberrations chromatiques sur le proche-infrarouge jusqu’à 850 nm, ce qui le rend avantageux pour l’imagerie quantitative.

Objectif à faibles aberrations chromatiques
Grossissement : 60X
ON : 1,4 (immersion dans l’huile)
D.T. : 0,12 mm
Plage de compensation de l’aberration chromatique : 405–650 nm
Maintien de la mise au point avec le système TruFocus d’Olympus
Le module TruFocus utilise une lumière infrarouge à phototoxicité minimale (laser de classe 1) afin d’identifier la position du plan de l’échantillon. Le mode de mise au point automatique (AF) one-shot permet à l’utilisateur de définir plusieurs positions de mise au point selon les besoins pour les échantillons plus profonds, pour des acquisitions en focus stacking efficaces dans les expériences à positions multiples.
Commentaire du Dr Kyle Miller
Dr Kyle Miller |
Dr JaeJin Kim | Fluorescence imaging is a widely used technique in DNA damage signaling and repair studies to analyze the localization and kinetics of DNA damage response factors to DNA damage sites. Obtaining this information has been critical in identifying how these factors detect and repair DNA lesions at single cell resolution. The FV3000 microscope has allowed Dr. JaeJin Kim and others in the Miller lab to generate DNA damage using laser-microirradiation and study DNA damage response factor behavior both in fixed and live cells. The availability of sensitive detectors, automatic focus capabilities, and super-corrected objectives has made the FV3000 microscope an effective instrument in conducting studies on DNA damage response pathways in human cancer cells. |
Remerciements
Cette note d’application a été rédigée avec l’aide des chercheurs suivants :
Dr Kyle Miller et Dr JaeJin Kim, NMS, The University of Texas à Austin
Produits utilisés pour cette application
a bien été ajouté à vos favoris
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
Not Available in Your Country
Sorry, this page is not
available in your country.

