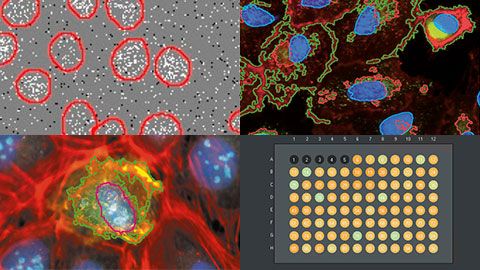
Neste webinar, Manoel e Shohei, nossos especialistas em deep learning e triagem de alto conteúdo, apresentarão o criador de ensaios do sistema scanR. O sistema scanR é a plataforma dedicada de triagem de alto conteúdo da Olympus com uma abordagem única de navegação de amostras e análise inspirada na citometria de fluxo.
Perguntas frequentesPerguntas frequentes sobre o webinar | Triagem de alto conteúdo: análise personalizada simplificadaAlgumas células apresentam duas cores diferentes (por exemplo, verde e azul), sugerindo duas fases diferentes do ciclo celular. Isso é uma indicação de que o modelo não está funcionando corretamente?O sistema pode diferenciar entre múltiplas cores/canais, de modo que quando há duas cores diferentes, o software identifica o fundo do primeiro plano (onde o primeiro plano é a camada de células). Contudo, como algumas fases do ciclo celular podem ser identificadas com características morfológicas, sugerimos fortemente o uso de uma etiqueta de fase celular para marcar as diferentes fases celulares. Isso será fundamental para garantir que o treinamento (deep learning) seja realizado com todas as fases do ciclo celular que você deseja diferenciar. Entretanto, como as células são avaliadas por pixel e não "por célula", há cenários em que o sistema será capaz de discernir automaticamente as fases do ciclo celular automaticamente, mesmo que você não tenha treinado especificamente com essa fase. Como o sistema funciona ao analisar culturas de esferoides ou outras culturas?O sistema scanR é projetado para funcionar eficazmente com culturas de esferoides ou com outros tipos de cultura. Existem alguns detalhes do fluxo de trabalho que nós observamos que funcionam bem com esta aplicação. Primeiro, recomendamos que você aplique a deconvolução 3D às imagens coletadas e, em seguida, faça a anotação manual. Um exemplo de fluxo de trabalho consistiria em usar 10–20 imagens e segmentar manualmente 10 células. É possível obter imagens das bordas dos poços nas placas de 384 poços?Sim, mas existem limitações. A capacidade de obter imagens das bordas dos poços depende da combinação do tipo de placa de poços e da objetiva usada. As placas de poços vêm em uma variedade de formas, incluindo com saia, semissaia e sem saia. Portanto, a informação importante ao obter imagens das bordas dos poços é a diferença de altura entre o fundo da placa e o fundo da saia. Dessa forma, chega-se a um ponto em que a distância vertical do fundo da placa até o fundo da saia impede que uma objetiva de alta ampliação obtenha imagens, pois a objetiva pode colidir com a placa. Nesse caso, sugerimos fortemente uma objetiva de longa distância de trabalho. Particularmente, a Olympus oferece uma grande variedade de objetivas de longa distância de trabalho. Como é feito o foco no sistema scanR?O sistema scanR usa uma combinação de foco automático de hardware e software. O foco automático de hardware detecta a superfície inferior da célula e o foco automático de software encontra a parte da amostra. Esse é um processo muito robusto, de modo que basicamente você pode abandonar o sistema depois de iniciar o experimento. É possível usar o sistema de disco giratório de super-resolução para a triagem de alto conteúdo?Sim, é possível usar o software scanR para controlar o sistema de microscópio Olympus IXplore SpinSR. É possível atingir uma resolução de 120 nm com esse sistema e, em seguida, importar as imagens adquiridas para o software cellSens para processamento posterior e resultados ainda melhores. Qual é o desempenho da segmentação e do deep learning em cortes de tecido?De acordo com a nossa experiência, a segmentação funciona bem em cortes de tecido, especialmente ao usar o sistema de disco giratório. Contudo, é possível adquirir boas imagens sem o sistema de disco giratório apenas aplicando a deconvolução 3D. Se os tecidos são particularmente difíceis de trabalhar, o deep learning com anotação manual tem mostrado funcionar bem. A anotação manual fornece ao algoritmo de deep learning um conjunto de treinamento robusto a partir do qual aprender e prever melhor os segmentos, com base na identificação do usuário. |