Quantifizierung von C-Fos-positiven Neuronen in Mausgehirnschnitten mit TruAI Deep-Learning-Technologie
Fortschritte in der künstlichen Intelligenz (KI) —vom maschinellen Lernen zum Deep Learning
Der Begriff „künstliche Intelligenz“ ist seit langem mit der Vorstellung einer futuristischen Technologie verbunden, die die Möglichkeiten für Forschung und technische Entwicklung erheblich erweitern kann. Künstliche Intelligenz gibt es seit den 1950er Jahren, und seither wurde sie in vielfältiger Weise eingesetzt. Im weitesten Sinne bezeichnet künstliche Intelligenz (KI) jede Technik, die es Maschinen ermöglicht, menschliche Intelligenz zu imitieren. Zur KI gehört auch eine weiter entwickelte Technik, das sogenannte „maschinelle Lernen“. Dabei werden Maschinen mit statistischen Methoden in die Lage versetzt, ohne direkte Programmierung Aufgaben aus Daten zu lernen. Die modernste Form der KI wird als „Deep Learning“ bezeichnet und nutzt neuronale Netze mit vielen Schichten, um Darstellungen und Aufgaben direkt aus komplexen Datensätzen zu lernen. Deep Learning ist inzwischen so leistungsfähig, dass es den Menschen hinsichtlich der Genauigkeit der Bildklassifizierung häufig übertrifft.
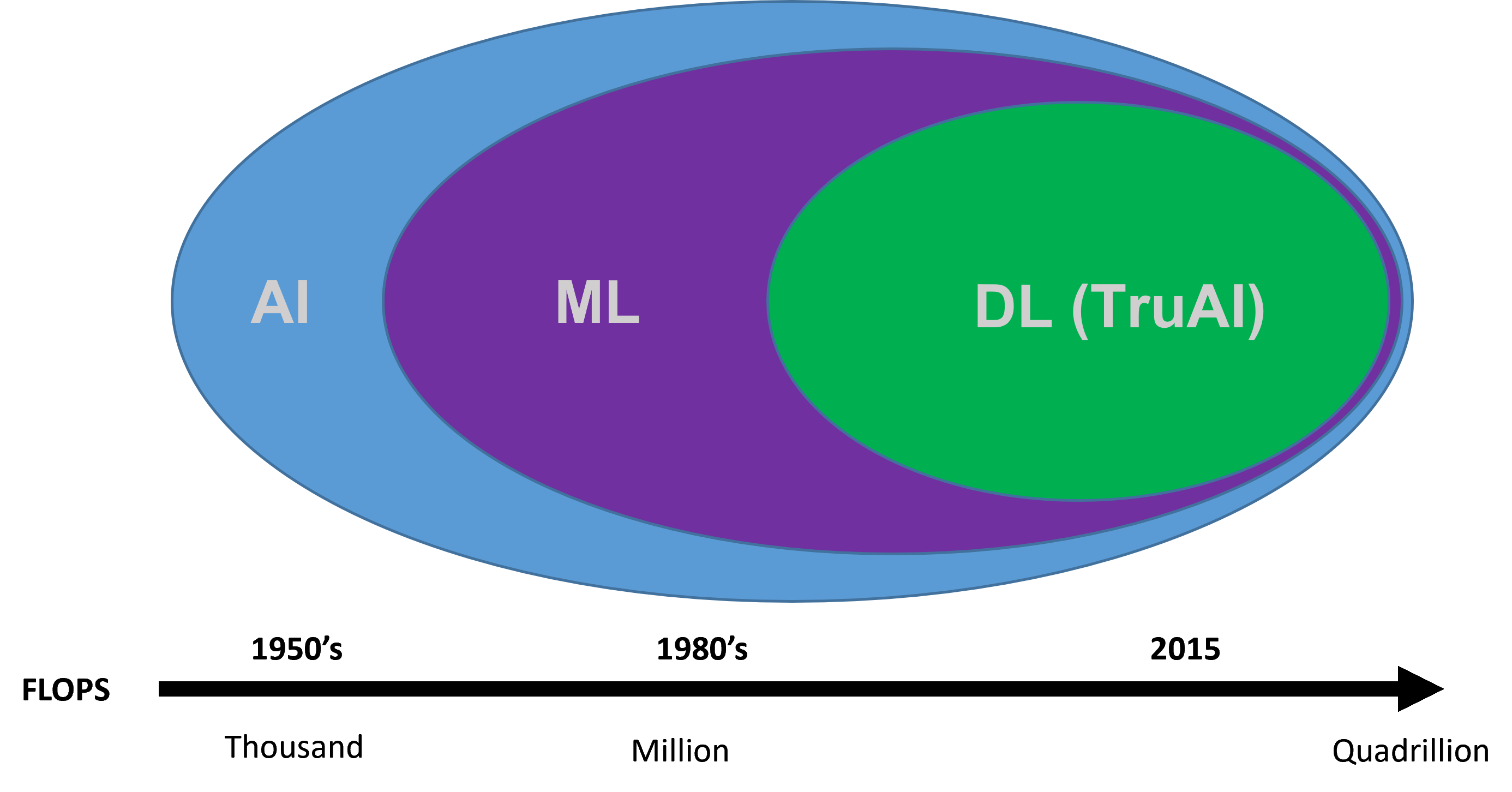
Abbildung 1. Zeitleiste der Entwicklung künstlicher Intelligenz
Anwendung von Deep-Learning KI in der Mikroskopie
In der Mikroskopie können es KI und insbesondere Deep Learning ermöglichen, eine robustere Analyse von Daten mit höherer Genauigkeit und in kürzerer Zeit durchzuführen. Bei den neuronalen Netzen der TruAI Technologie von Olympus handelt es sich um neuronale Konvolutionsnetze, deren Technologie zur Segmentierung von Objekten sich sehr gut an die Analyse komplexer oder schwieriger Datensätze anpassen lässt. Diese Netze können die verschiedensten Eingabedaten oder Belege auswerten und Entscheidungen zu diesen Daten treffen—eine Aufgabe, die bei manueller Ausführung normalerweise der menschlichen Subjektivität unterliegt. Diese Anwendungsmitteilung zeigt an einem praktischen Beispiel, wie die TruAI Technologie die automatische Segmentierungsanalyse von c-Fos-positiven Neuronen im Mäusegehirn unterstützt, insbesondere im Vergleich zu weniger fortschrittlichen KI-Tools.
Experimentübersicht—Beurteilung der systemischen Folgen einer gestörten Neurogenese
In diesem Experiment sollte die Expression von c-Fos-positiven Neuronen in der Hippocampus-Region des Gehirns (dem Bereich des Gehirns, der für Gedächtnis und Lernen verantwortlich ist) quantifiziert werden, um die systemischen Folgen einer gestörten Neurogenese zu bewerten. c-Fos ist ein Proto-Onkogen, dessen Expression als Marker für neuronale Aktivität verwendet werden kann. Herkömmliche Intensitäts-/Morphometrie-Segmentierungsmethoden erzeugen robuste Datensätze weder effektiv noch effizient, da die Datensätze unterschiedliche Hintergrund- und Zielintensitäten aufweisen, die manuelle Analyseversuche erschweren (Abbildung 2) und eine zeitaufwändige Überprüfung der Ergebnisse erfordern, um falsch-positive Ergebnisse zu entfernen.
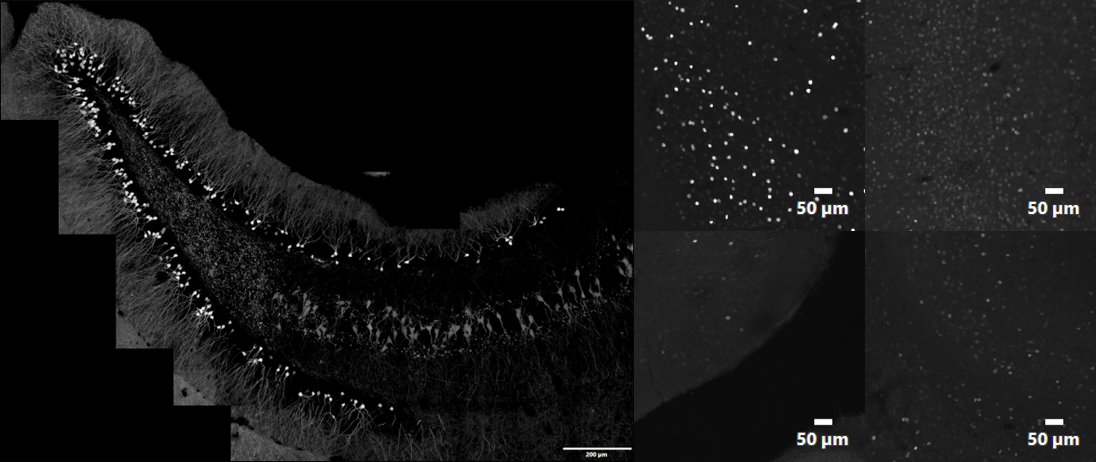
Abbildung 2: Beispiel für die Art von Datensätzen, die eine Segmentierungsanalyse erfordern: Panoramabild der gesamten Hippocampus-Region einer transgenen Maus, die tdTomato unter einem c-Fos-Promotor exprimiert (links), sowie Einzelbilder zur Veranschaulichung der Variabilität des c-Fos-Signals sowie der Bildhintergrund-Level innerhalb des zusammengefügten Bildes (rechts). 1k × 1k Z-Stapel-Bilder, aufgenommen mit 10X SAPO und 2x Zoom bei 1 AU.
Training des neuronalen TruAI Netzes zur Identifizierung der C-Fos-positiven Expression
Das Trainingsprotokoll des neuronalen Netzes der TruAI Technologie erfordert zunächst einen „Trainings“-Datensatz als Grundlage für die weitere Identifizierung und Analyse. Diese Vergleichswerte können entweder auf den Anmerkungen des Benutzers zu den Rohdaten basieren oder durch die Intensitäts-/Morphometrie-Schwellenwert- und Segmentierungsfunktion der cellSens Software automatisch erzeugt werden. Ausgehend von diesem ersten Dateninput lernt das neuronale TruAI Netz und nutzt experimentelle Datensätze während der iterativen Trainingsperiode, statt zusätzliche Eingaben vom Benutzer zu verlangen. Durch diese Festlegung der Vergleichswerte kann das neuronale TruAI Netz immer besser erkennen, was real ist (Target) und was nicht (kein Target). In diesem Experiment wurde das neuronale TruAI Netz mit acht Datensätzen und unterschiedlichen c-Fos-Expressionsniveaus trainiert (Abbildung 3). Ausgehend von diesen Datensätzen generierte das neuronale TruAI Netz durch robustes Training mit 40.000 Iterationen das neuronale Netz, das zur Analyse aller weiteren Bilder verwendet werden sollte. Während des iterativen Trainingsprozesses wurde ein Validierungsbild aus dem ersten Trainingssatz ausgewählt, kontrolliert und als Vergleich herangezogen.
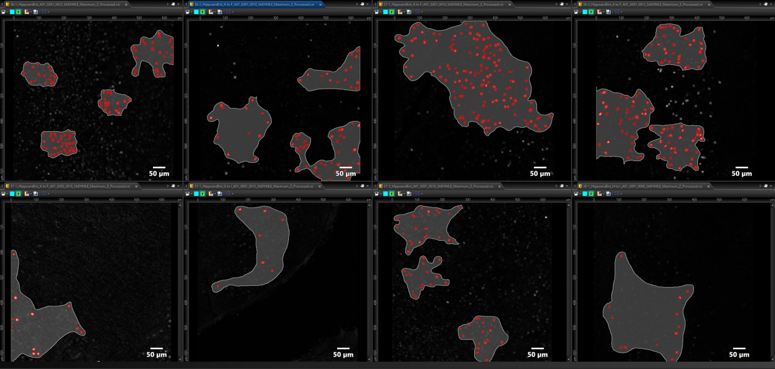
Abbildung 3: Trainingsdatensätze, die zur Erstellung des neuronalen TruAI Netzes verwendet werden: Es wurden acht Datensätze mit unterschiedlichen Expressionsniveaus verwendet, um Objekt (rot) und Hintergrund (grau) voneinander abzugrenzen.
Verwendung des neuronalen TruAI Netzes zur Analyse von Stapeldatensätzen
Sobald das trainierte neuronale Netz mit TruAI generiert war, wurde es zur Stapelverarbeitung und Analyse der übrigen Datensätze verwendet. Abbildung 4 zeigt die Wahrscheinlichkeitskarte und die anschließende Segmentierungsanalyse, die mit dem resultierenden neuronalen TruAI Netz durchgeführt wurde. Mit dem neuronalen TruAI Netz wurde der Datensatz anhand der Wahrscheinlichkeitsschicht segmentiert und automatisch durch die Auto-Split- und Klassifizierungsalgorithmen von cellSens verarbeitet, um quantitative Metriken der mittleren Intensität, Gesamtintensität und Fläche der c-Fos-positiven Zellen zu erhalten. Die Makro-Manager-Funktion der cellSens Software ermöglichte die Stapelverarbeitung aller übrigen Datensätze.
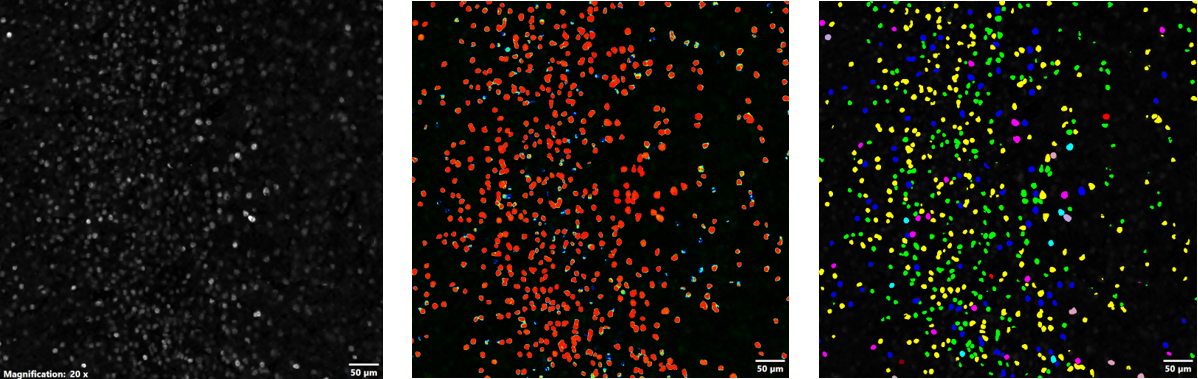
Abbildung 4: Verwendung des neuronalen Netzes zur Schwellenwertbildung und Segmentierung: Mit dem FV3000-Mikroskop aufgenommenes Bild c-Fos-positiver Zellen (links), vom neuronalen Netz erstellte Wahrscheinlichkeitskarte c-Fos-positiver Zellen (Mitte) und Segmentierung der c-Fos-positiven Zellen anhand der Wahrscheinlichkeitsschicht (rechts); die Daten wurden automatisch aufgeteilt und anhand der mittleren Intensität, Gesamtintensität und Fläche klassifiziert.
Wie bereits erwähnt, besteht der Vorteil des Einsatzes von Deep-Learning-Technologien bei der Bildanalyse darin, dass sie eine robustere Identifizierung von Objekten ermöglichen als andere KI-Programme. In diesem Experiment verwendete die TruAI Deep-Learning-Technologie acht Datensätze mit 40.000 Iterationen, um ein neuronales Netz für die weitere Analyse der übrigen Datensätze zu erzeugen. Der Vorteil dieser robusten Trainingsprotokolle besteht darin, dass das neuronale Netz Objekte auch dann genauer erkennen kann, wenn das Bild nicht ideal ist, z. B. wenn das Hintergrundsignal stark und die Zielexpression niedrig ist. Dieser Vorteil wird in Abbildung 5 deutlich, in der die Leistung des neuronalen TruAI Netzes mit der eines weniger strikt trainierten Netzes mit nur einem Trainingsdatensatz verglichen wird. Bei höheren Expressions-Levels identifizierten beide Algorithmen die gleiche Anzahl von Zellen. Bei sehr niedrigen Expressions-Levels überschätzte das weniger strikt trainierte Netz jedoch die Anzahl der c-Fos-positiven Zellen um fast das Doppelte, was die Schlussfolgerungen aus dem Experiment erheblich verändert hätte. Die TruAI Deep-Learning-Technologie erkannte erfolgreich unterschiedliche Hintergrund- und c-Fos-Intensitäten und lieferte genauere Ergebnisse, ohne dass eine manuelle Prüfung der Daten zur Beseitigung falsch positiver Ergebnisse erforderlich war.
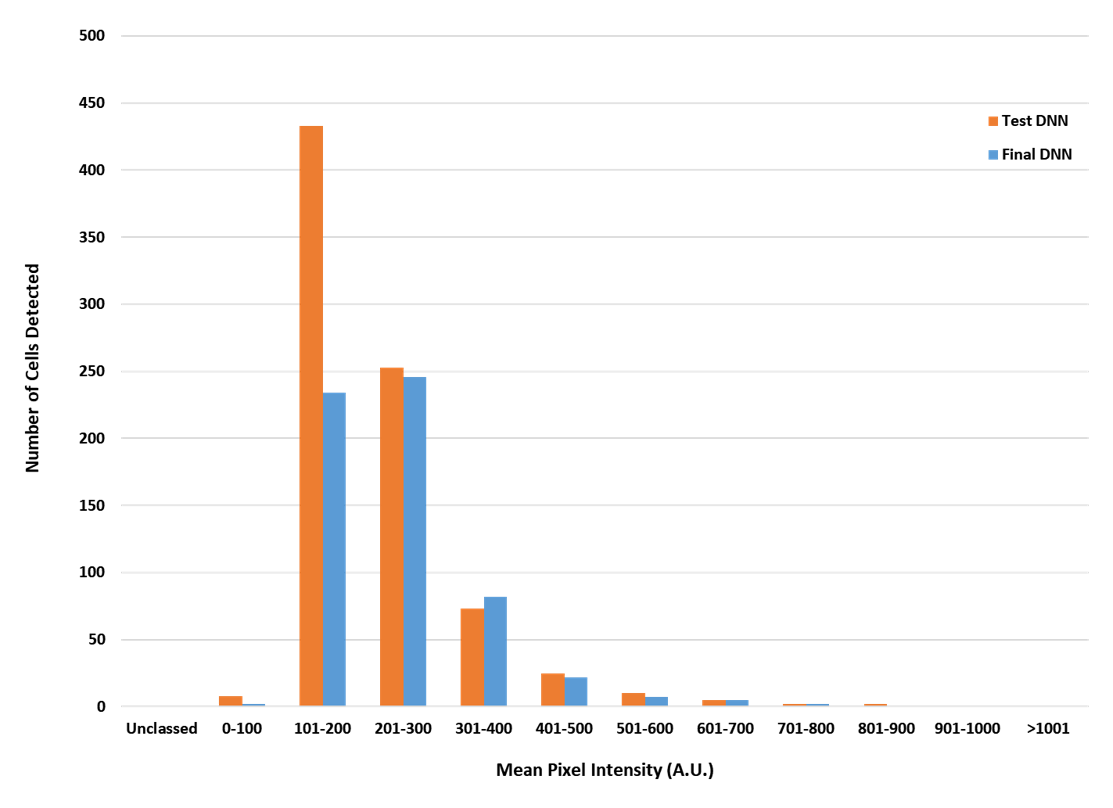
Abbildung 5: Vergleich der c-Fos-positiven Erkennungsraten eines weniger robust trainierten Netzes mit denen des neuronalen TruAI Netzes; die orangefarbenen Balken stellen das weniger strikt trainierte Netz dar, das aus einem einzigen Datensatz mit 2000 Iterationen abgeleitet wurde; die blauen Balken zeigen die Ergebnisse des neuronalen TruAI Netzes, erstellt aus acht Datensätzen mit 40.000 Iterationen.
Kommentar des Forschers Dr. Jonathan Epp, Universität von Calgary
 | Der Nutzen von KI-Konzepten für die wissenschaftliche Forschung hängt meiner Meinung nach von mehreren Faktoren ab. Ein wichtiger Punkt, den ich bei der Diskussion über solche Systeme immer wieder anspreche, ist die Beseitigung der Subjektivität bei der Quantifizierung von Bildern. Auch wenn wir aus irgendeinem Grund die Versuchsbedingungen berücksichtigen, entscheiden wir nicht anhand des tatsächlichen Versuchsgewebes, was eine markierte Zelle ist und was nicht. Abgesehen von der Subjektivität verringert sich dadurch auch die Variabilität zwischen verschiedenen Experimentatoren und Experimenten im Labor, was letztlich die Reproduzierbarkeit unserer Daten erhöht. Meiner Meinung nach ist dieser Ansatz ein wichtiger Weg hin zur Verbesserung der Reproduzierbarkeit, eines akuten Problems in der heutigen Wissenschaft. |
Danksagungen
Dieser Anwendungshinweis wurde mit Hilfe des folgenden Wissenschaftlers erstellt:
Dr. Jonathan Epp, Universität von Calgary
Verwendete Produkte
wurde erfolgreich zu Ihren Lesezeichen hinzugefügt
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
Not Available in Your Country
Sorry, this page is not
available in your country.