ディープラーニングを用いた薬品検査のための マルチクラス核表現型の予測
はじめに
薬品に反応した核内受容体などのタンパク質の動きを定量化するには、核染色を使用した安定した核セグメンテーション手法が必要です。しかし、このプロセスには細胞染色に時間や費用がかかることをはじめ、光毒性、光退色、および感光性化合物の不活性化などにつながる可能性があるなどの課題があります (e.g., R1881※1)。
これらの課題を克服するために、TruAIディープラーニング機能を備えたイメージングソフトウェアcellSensを用いて、核染色をせずに細胞核を検出するためのニューラルネットワーク(NN)を開発、非染色の細胞核の検出をしました。さらに、薬品に応答した異なる細胞表現型を区別するようにNNを学習させた後、アンドロゲン受容体(AR)標的薬で処理された生きた前立腺癌細胞の細胞内ARダイナミクスを予測しました。
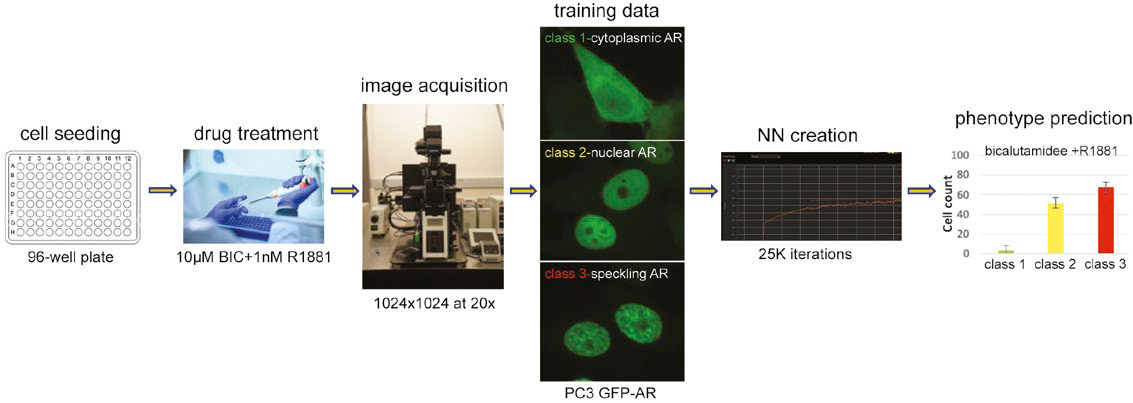
図1: 学習させてNNを用いてマルチクラス核表現型を予測するワークフロー
細胞質内のARは、活性化されることにより核に移行し、転写活性部位に局在します。この様子は共焦点顕微鏡で観察すると、明確な斑点パターンとして確認することができます。 このARダイナミクスを予測するために、まず核染色をせずに細胞核を検出するNNを作成し、次にGFP-AR核転座とトランス活性化(スペックル)状態に基づいて細胞を分類しました。その結果、NNワークフローが核を染色することなくこれらの重要なAR表現型を正常に予測できることを確認しました。
利点
- 核染色を使用せずにターゲットシグナルのみを用いて異なる細胞の表現型の特定が可能
- 染色や画像解析に費やしていた時間やコストの軽減
- 光毒性と光退色の低減
- 光感受性化合物の活性を維持
実験方法
薬物反応時に細胞核が示す主な3パターンのAR表現型から学習データを作成しました。
最初に画像の半分を使用してNNを構築した後、残りの半分を使用してそのパフォーマンスを評価しました。最後にマルチクラスNNを核染色していない独立したデータに適用しました。
3つのAR表現型からの学習データの準備
NNを構築する最初のステップとして、3つの主要なAR表現型で構成される学習データを作成しました。
- 不活性な細胞質内AR(未処理、NTC)
- リガンド結合しているが不活性な核内AR(10 µMビカルタミド)
- アクティブな核内ARスペックル(1 nM R1881)
教師画像を作成するために、SiR-DNAを使用して核を染色しました。オリンパスの共焦点レーザー走査型顕微鏡FV3000のガルバノスキャナーを使用して、UPLSAPO20X対物レンズで1024×1024ピクセル比の画像を取得しました。その後、取得した画像データ(60枚)を学習用と検証用(各30枚の画像)に分けました。NNを学習させるために、核染色とGFP-AR染色を使用した3つのAR表現型(データの50%、合計30枚の画像)からそれぞれ10枚のMIP画像を使用しました。
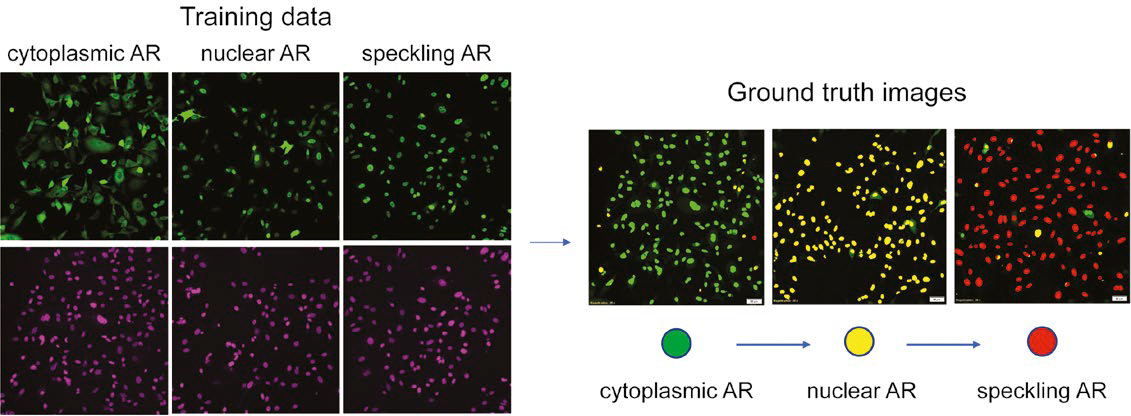
図 2: 核染色に基づくマルチクラスAR表現型学習データの準備。 (左)緑:AR-GFP、紫:SiR-DNA。 (右)教師画像の評価
教師画像の調整
図2はNNによって評価された教師画像を示しています。細胞質内ARは緑、核内ARは黄色、斑点ARは赤色で示されています。この色分けされたセグメンテーションマップを用いて、核の検出とセグメンテーションのエラーを手動で修正し、誤って分類された核を再分類することにより、教師画像を調整しました。
TruAIを使用したニュートラルネットワークの作成
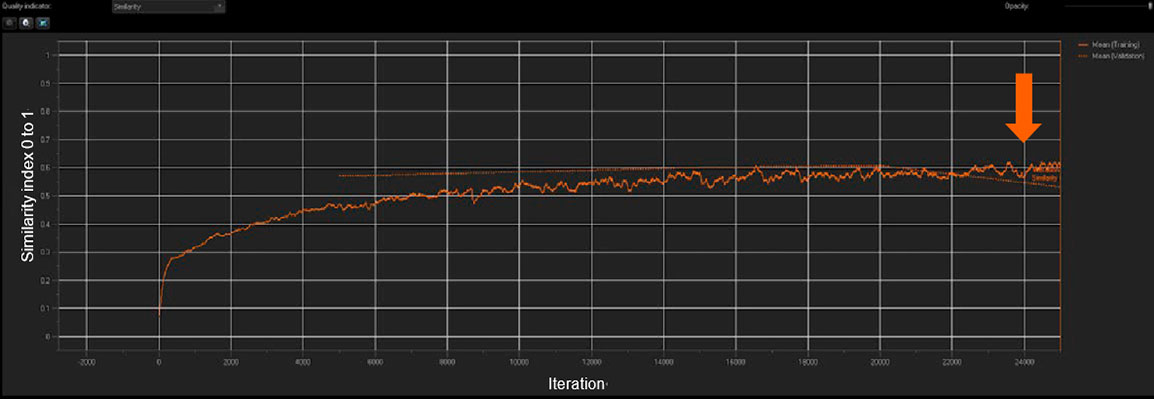
図 3: NNの学習は、類似性指数と検証平均値をリアルタイムで測定
TruAIを用いて、各AR表現型からの10枚の学習画像を組み込んだマルチクラス構成の標準NNを作成しました。 cellSensのTruAIディープラーニング機能では、NNの品質と精度がソフトウェア上にリアルタイムで表示されます。図3に示すように、学習プロセス中、類似性指数はプラトーに達するまでは反復ごとに増加し、最終的に学習が飽和状態であることが示されます(矢印箇所)。チェックポイントは5,000回の反復(20%の進行)ごとに作成され、最適なチェックポイントを類似性指数とともに学習済みNNとして保存されます。
結果
ブラインドデータでのマルチクラスニューラルネットワークの検証
教師画像またはNNの学習(ブラインドデータ)に使用されていない半分の画像を使用して、マルチクラスNNモデルを検証しました。これらの検証結果を使用して、エリアフィルター(最小49.83 µm² –最大555.44 µm²)を適用しモデルパラメーターの微調整を行い、細胞の破片や誤ってセグメント化された細胞を排除しました。学習したNNモデルは、核染色なしでGFP-ARシグナルからマルチクラスAR表現型を予測しました。図4は、10枚の画像のデータを使用した表現型クラスの分布を示しています。
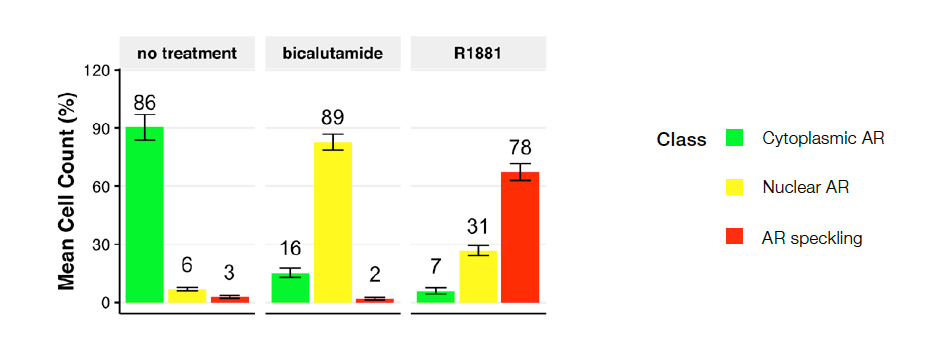
図 4:未処理(左)、ビカルタミド処理(中央)、R1881処理(右)を行ったもの。プロットは、クラス1(細胞質内AR)、クラス2(核内AR)、およびクラス3(スペックル状AR)に分類された細胞のパーセンテージを示す。ビカルタミド処理またはR1881処理を行ったデータではほとんどクラス1細胞を得られなかった。ビカルタミド(ARアンタゴニスト)はクラス2へのシフトを誘発し、R1881(ARアゴニスト)はクラス3につながり、転写活性部位に局在する活性ARを得られた。
独立したデータでのマルチクラスAR表現型の予測
次に、独立した画像でマルチクラスNNモデルを評価するために、抗アンドロゲン薬であるビカルタミドとエンザルタミドを使用して新しいデータセットを作成しました (図5)。
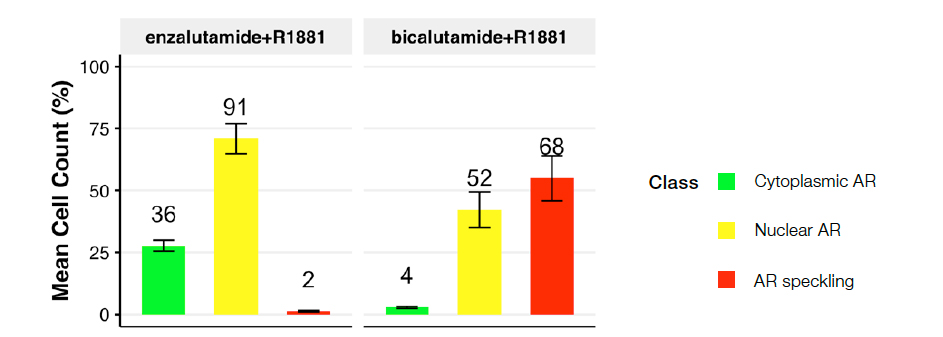
図 5: R1881とエンザルタミド処理(左)またはビカルタミド処理(右)を行ったもの。プロットは、処理条件全体で予測される核クラスの分布を示す。
第二世代の抗アンドロゲン薬であるエンザルタミドによる処理は、斑点状のAR(クラス3)がほとんどない顕著な細胞質内AR(クラス2)が多く、リガンド誘導性AR活性化を遮断していることを示唆しています。一方、古く効果の低い抗アンドロゲン薬であるビカルタミドは、アクティブAR(クラス3)への顕著にシフトしており、1nMR1881によって誘発されるAR活性化を効果的にブロックできないことを示唆しています。これらのデータは、これまでの研究結果を裏付けています※2。
結論
オリンパスの共焦点レーザー走査型顕微鏡FV3000とTruAIを備えたイメージングソフトウェアcellSensを用いて、核染色を使用せずに核位置を推定・セグメント化し、さらに薬品に応じたAR表現型の変化に基づいて細胞をワンステップで分類するNNを構築しました。このアプローチは実験に費やす時間とコストを削減することにより、薬品検査の効率化に貢献します。
さらに、NNは光毒性、光退色、感光性化合物の不活性化などのアーティファクトを低減することでデータ品質を向上させ、研究者や従来の画像分析パイプラインでは見落とされる可能性のある僅かな変化の検出が可能です。cellSensのTruAI機能は、タンパク質の局在化から形態計測の変化に至るまで、核または細胞ベースのパラメーターに基づいた細胞集団の細分類に幅広く活用することができます。
参考文献
- Risek B, Bilski P, Rice AB, Schrader WT. Androgen receptor-mediated apoptosis is regulated by photoactivatable androgen receptor ligands. Mol Endocrinol. 2008 Sep;22(9):2099-115. doi: 10.1210/me.2007-0426. Epub 2008 Jun 18. PMID: 18562628; PMCID: PMC2631375.
- Sugawara T, Lejeune P, Köhr S, Neuhaus R, Faus H, Gelato KA, Busemann M, Cleve A, Lücking U, von Nussbaum F, Brands M, Mumberg D, Jung K, Stephan C, Haendler B. BAY 1024767 blocks androgen receptor mutants found in castration-resistant prostate cancer patients. Oncotarget. 2016 Feb 2;7(5):6015-28. doi: 10.18632/oncotarget.6864. PMID: 26760770; PMCID: PMC4868737.
著者
- Harish Sura, Katherin Patsch, and Seungil Kim of the Lawrence J. Ellison Institute for Transformative Medicine, University of Southern California, Los Angeles, CA
- Shohei Imamura, Olympus Tokyo
このアプリケーションノートに関連する製品
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
このページはお住まいの地域ではご覧いただくことはできません。