Überwachung der Zellzyklusdynamik während der Stammzelldifferenzierung mit der TruAI Deep-Learning-Technologie des scanR Systems
Einführung
Während der frühen Entwicklung von Säugetieren durchlaufen embryonale Stammzellen (ES-Zellen) eine zelluläre Spezifikation und bringen alle embryonalen Keimblätter hervor, aus denen schließlich alle unterschiedlichen Zelltypen im erwachsenen Organismus hervorgehen. ES-Zellen zeichnen sich durch ihre unglaubliche Fähigkeit zur Selbsterneuerung und Proliferation aus und haben aufgrund verkürzter GAP-Phasen (G1 und G2) kurze Teilungszyklen. Während der zellulären Differenzierung finden markante zelluläre Ereignisse statt: Die Zellen verändern ihre Morphologie: Sie vergrößern sich um etwa das Zehnfache, verändern das Verhältnis von Zellkern zu Zytoplasma und werden flach und länglich; es werden Genexpressionsprogramme aktiviert, welche die weitere Entwicklung der Zelle beeinflussen, und globale Chromatinveränderungen treten auf. Vor allem aber verlangsamt sich der Zellzyklus. Differenzierte somatische Zellen haben lange G1- und G2-Phasen, eine strenge Checkpoint-Kontrolle sowie gut regulierte Teilungszyklen (Padgett und Santos 2020) (Abbildung 1).
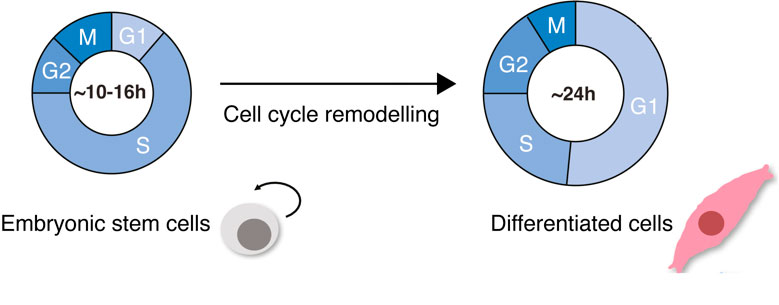
Die Remodellierung des Zellzyklus ist ein Phänomen, das nicht nur bei der Differenzierung von ES-Zellen auftritt. Veränderungen in der Zellzyklusdynamik kommen in der Biologie häufig vor, zum Beispiel bei der Regeneration von Zellen nach Virusinfektionen oder bei malignen Tumoren. Die Klärung der Veränderungen der Zellzyklusdynamik im Zusammenhang mit der zellulären Differenzierung ist daher wichtig für das Verständnis potenziell konservierter Mechanismen der Zellzykluskontrolle und wird sich auf unser Verständnis des Gleichgewichts zwischen gesund und krank auswirken.
Die Veränderungen während der Differenzierung können in einzelnen Zellen durch Messung der Aktivität von genetisch codierten Proteinen, die in verschiedenen Stadien des Zellzyklus beteiligt sind, nachverfolgt werden (Araujo et al. 2016; Sakaue-Sawano et al. 2008). Ein solcher Sensor ist der fluoreszierende, auf Ubiquitinierung basierende Zellzyklusindikator FUCCI(CA), der von Miyawaki und Kollegen entwickelt wurde (Sakaue-Sawano et al. 2017) und der den Abbau von Cdt1 und Geminin, zwei wichtigen Zellzyklusproteinen, überwacht und eine scharfe Trennung der G1-, der S- und der G2-Phase in drei Farben (rot, grün und gelb) erzeugt, die im YFP- und im mCherry-Kanal eines Mikroskops beobachtet werden können (Abbildung 2).
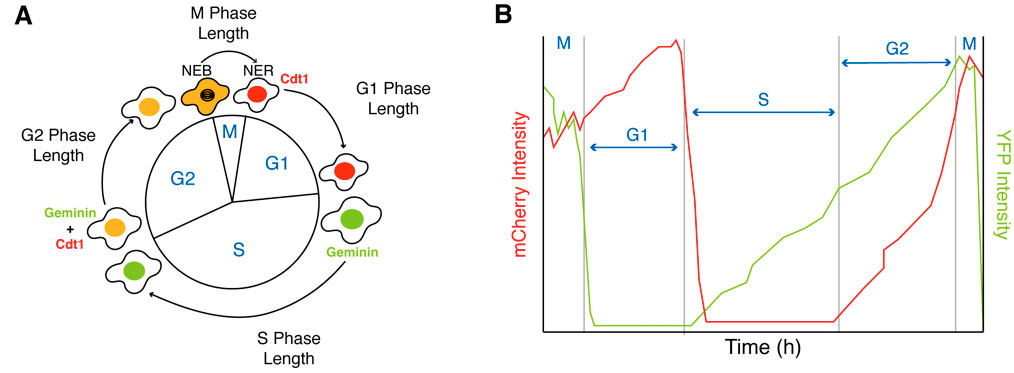
Abbildung 2: Messung der Phasen G1, S, G2 und M des Zellzyklus mit dem FUCCI(CA)-Sensor. (A) Schematische Darstellung des FUCCI(CA)-Sensors. (B) Beispielspuren der Dauer der Phasen G1, S, G2 und M auf der Grundlage der Expressionsniveaus von Cdt1 (mCherry-Kanal) und Geminin (YFP-Kanal).
Die Überwachung der Zellzyklusdynamik während der Zelldifferenzierung stellt jedoch eine Herausforderung sowohl für die Bildgebung als auch für die Datenanalyse dar. Die Langzeitdarstellung lebender Zellen über Tage hinweg erfordert eine stabile Temperatur- und CO2-Kontrolle, eine schonende Beleuchtung zur Minimierung der Phototoxizität und robuste Autofokus-Einstellungen, um während des gesamten Zeitrafferprozesses eine gute Bildqualität zu gewährleisten.
Die größte Herausforderung bei der Analyse ist die Segmentierung und anschließende Verfolgung einzelner Zellen bei niedrigen Fluoreszenzintensitäten und hoher Konfluenz, während sich die ES-Zellen während der Differenzierung bewegen, teilen und ihre Form drastisch verändern.
All diese Herausforderungen lassen sich mit dem Olympus ScanR Mikroskop zur Aufnahme von High-Content-Epifluoreszenz meistern, das mit einem cellVivo-Inkubator, einer Hardware für Z-Drift-Kompensation (ZDC) zur Autofokussierung, der TruAI™ Deep-Learning-Technologie für die Segmentierung und einem kinetischen Modul für das Tracking ausgestattet ist.
Ziele
In diesem Anwendungshinweis zeigen wir, dass die Anwendung der TruAI-Technologie die Erkennung und Verfolgung einzelner ES-Zellen durch Langzeitbildgebung auf der Grundlage des FUCCI(CA)-Sensors erheblich verbessern kann. Dies ist besonders bei solchen Zellen von Vorteil, die keinen Kernmarker aufweisen, der ein Tracking ermöglicht, und vermeidet unnötige Beleuchtung und Phototoxizität.
Mit der TruAI-Technologie wird das Modell eines tiefen neuronalen Netzes (DNN) generiert, das die Position von pluripotenten ES-Zellen und differenzierten Zellen zu allen Zeitpunkten robust identifizieren kann, selbst wenn die Fluoreszenzintensität sehr schwach ist. Dies ermöglicht eine robuste Überwachung der Phasen G1, S und G2 von Zellpopulationen im Zeitverlauf. Anschließend können wir mit dem scanR Kinetikmodul Tausende von Zellen verfolgen, ihre Spuren über lange Zeiträume auswerten und quantitative dynamische Informationen über Zellzyklusveränderungen auf der Ebene einzelner Zellen gewinnen.
Um die Vielseitigkeit des scanR Systems als Forschungsinstrument zu demonstrieren, wurden alle gezeigten Daten, einschließlich Grafiken, Streudiagramme, Objektgalerien, Tracks und kinetische Tracks, ausschließlich mit der scanR Software erzeugt.
Versuchsaufbau
H1 humane embryonale Stammzellen (hES-Zellen), die den FUCCI(CA)-Sensor exprimieren, werden mit 50 ng/ml BMP4 (Bone Morphogenic Protein 4) behandelt, um eine Differenzierung zum Mesendoderm anzuregen. Diese H1-Zellen werden dann mit dem scanR Mikroskop mit einem 10X UPLANSAPO Objektiv im mCherry- und im YFP-Kanal über 6 Tage alle 15 Minuten abgebildet.
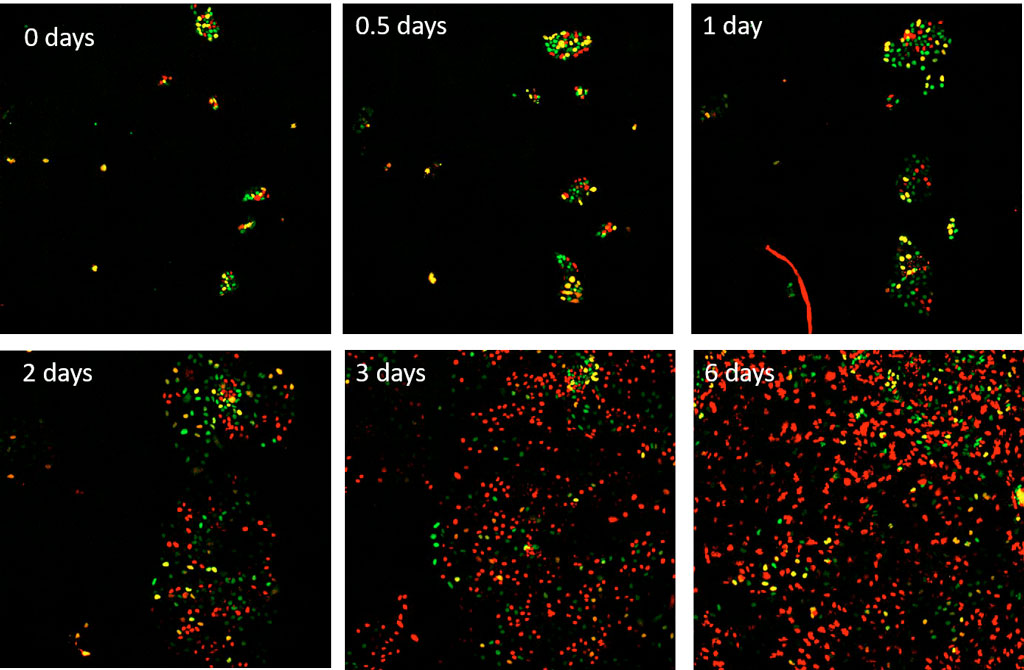
Abbildung 3: Zeitreihenaufnahmen von hESC H1-Zellen, die den FUCCI(CA)-Sensor exprimieren und zu Beginn der Aufnahme mit BMP4 stimuliert wurden, über 6 Tage. Rot, grün und gelb entsprechen der G1-, S- bzw. G2-Phase.
Entwicklung eines DNN zur Bildgebung lebender Zellen
Eine einzelne Zelle, die den FUCCI(CA)-Sensor exprimiert, kann im Verlauf mehrerer Tage starke Schwankungen in der Fluoreszenzintensität aufweisen. Insbesondere beim Übergang von der G1- zur S-Phase kann die Gesamtintensität sehr niedrig sein (Abbildung 2b). Dies kann dazu führen, dass Tracking-Algorithmen versagen, da die Tracks abbrechen, wenn die Zellen nur schwer zu erkennen sind.
Um eine Zellverfolgung über mehrere Tage für eine möglichst große Anzahl von Zellen zu erreichen, muss ein DNN-Modell entwickelt werden, das in der Lage ist, Zellen auch bei sehr schwachen Intensitäten zu erkennen. In einem früheren White Paper haben wir gezeigt, dass dies möglich ist, indem wir Bildpaare mit langer Belichtung und intensiver Beleuchtung von fluoreszierenden Zellen im Vergleich zu kurzer Belichtung und schwacher Beleuchtung verwenden (Woerdemann 2020). Das heißt, es werden Langzeitbelichtungsbilder für die Segmentierung der Zellen verwendet und es wird eine Maske erstellt. Diese Maske wird als „Grundwissen“ (Ground Truth) verwendet, um das DNN-Modell zur Erkennung von Zellen in den niedrigen Expositionsstufen zu trainieren.
Alternativ kann das DNN-Modell auch durch manuelle Anmerkungen in dem zu analysierenden Datensatz trainiert werden. Für diese Aufgabe kann die cellSens™ Software von Olympus verwendet werden. Die in der cellSens Software entwickelten DNN-Modelle können in die scanR Software importiert werden. In diesem Anwendungshinweis haben wir die letztgenannte manuelle Methode verwendet und Hunderte von Kennzeichnungen von Zellen mit unterschiedlichen Intensitäten zu verschiedenen Zeitpunkten unternommen, um das DNN-Modell zu trainieren.
Segmentierung: Vergleich der TruAI Deep-Learning-Technologie mit klassischen Methoden
Nach der Erstellung des DNN-Modells wird es individuell auf den mCherry- und den YFP-Fluoreszenzkanal angewendet. Auf diese Weise wird eine Pixel-KI-Wahrscheinlichkeitskarte für jeden Kanal erstellt. Je höher die KI-Wahrscheinlichkeit in einem Pixel ist, desto höher ist die Wahrscheinlichkeit, dass das Pixel zu einer Zelle gehört. Anschließend werden die KI-Wahrscheinlichkeitskarten beider Kanäle summiert, und die Segmentierung erfolgt durch Anwendung einer Intensitätsschwelle auf diese Summierung (Abbildung 4e und 4f).
Um die Ergebnisse mit klassischen Methoden zu vergleichen, werden der mCherry- und der YFP-Fluoreszenzkanal mit einem Rolling-Ball-Algorithmus hintergrundkorrigiert, summiert und die Summe wird mit Hilfe eines Intensitätsschwellenwerts (Abbildung 4c) oder einer Randerkennungsmethode (Abbildung 4d) segmentiert. Alle oben beschriebenen Bildverarbeitungsschritte werden mit der scanR Analysesoftware durchgeführt.
Die statistischen Werkzeuge des scanR Systems werden zur Erstellung von Tabelle 1 verwendet, in der sich die Ergebnisse auf ein einziges Sehfeld über einen Zeitraum von 6 Tagen beziehen.
Tabelle 1: Vergleich der Anzahl der segmentierten Zellen pro Methode
| Zeitpunkte | Methoden der Segmentierung | ||
|---|---|---|---|
| Intensitätsschwelle | Randerkennung | TruAI-Technologie | |
| 0 h | 78 | 103 | 93 |
| 24 h | 209 | 188 | 240 |
| 48 h | 421 | 281 | 475 |
| 72 h | 847 | 654 | 986 |
| 96 h | 1480 | 1330 | 1590 |
| 160 h | 1340 | 1460 | 1610 |
|
Summierung aller Frames
0–160 h | 504131 | 573179 | 659199 |
Ein einfacher Vergleich des Fluoreszenzbildes mit dem KI-Wahrscheinlichkeitsbild (Abbildung 4a und 4b) zeigt, dass der TruAI-Ansatz eine höhere Empfindlichkeit aufweist und alle Zelltypen erkennt. Dies wird durch die in Tabelle 1 zusammengefassten Ergebnisse sowie den gesamten Zeitablauf bestätigt. Mit der klassischen Intensitätsschwellenmethode können schwach fluoreszierende Zellen nicht erkannt und ihre korrekte Form nicht bestimmt werden (Abbildung 4c). Die Randerkennungsmethode verbessert die Kontur der erkannten Zellen, lässt aber immer noch viele unscharfe Zellen außer Acht (Abbildung 4d). Die TruAI Deep-Learning-Technologie erkennt zuverlässig auch unscharfe Zellen, und deren Ränder sind gut definiert (Abbildungen 4e und 4f).
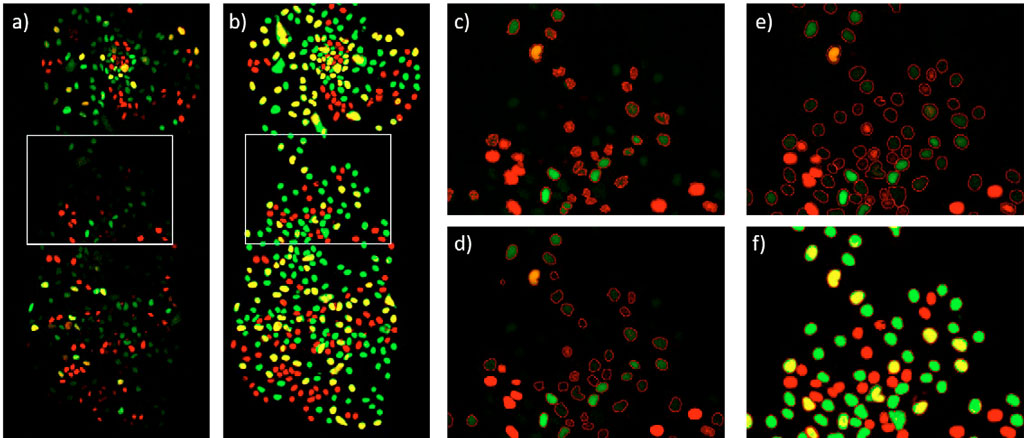
Abbildung 4: ES-Zellkolonie zum Zeitpunkt 48 h. a) Fluoreszenzkanäle mCherry (rot, G1-Phase) und YFP (grün, S-Phase). Die G2-Phase ist als Kombination von mCherry und YFP (gelb) dargestellt. b) TruAI-Wahrscheinlichkeit im Kanal mCherry (rot) und im Kanal YFP (grün). Zellen mit hoher KI-Wahrscheinlichkeit in beiden Kanälen sind gelb dargestellt. c) Segmentierung unter Verwendung einer Intensitätsschwelle in der Summierung des mCherry- und des YFP-Fluoreszenzkanals d) Segmentierung unter Verwendung eines Randdetektors in der Summierung des mCherry- und des YFP-Fluoreszenzkanals e) Segmentierung in der Summierung der TruAI-Wahrscheinlichkeitsintensitäten, überlagert mit dem Fluoreszenzbild f) wie e), jedoch überlagert mit der TruAI-Wahrscheinlichkeit.
Analyse der Zellpopulationen im Zeitverlauf
Die mCherry- und die YFP-Intensität segmentierter Zellen mit der TruAI-Technologie werden in einem Streudiagramm in verschiedenen Zeitintervallen dargestellt (Abbildung 5). Zellen, die nur eine mCherry-Intensität aufweisen, entsprechen der G1-Phase, Zellen, die nur eine YFP-Intensität aufweisen, entsprechen der S-Phase, und Zellen mit Intensitäten in beiden Kanälen entsprechen Zellen im G2-Stadium. Abbildung 5 zeigt, dass sich zu Beginn die meisten Zellen im S-Stadium befinden, während sich zu einem späteren Zeitpunkt die meisten Zellen im G1-Stadium befinden. Diese Populationsverschiebung stimmt mit den Zellzyklusprofilen überein, die für pluripotente bzw. differenzierte Zellen zu erwarten sind (Abbildung 1).
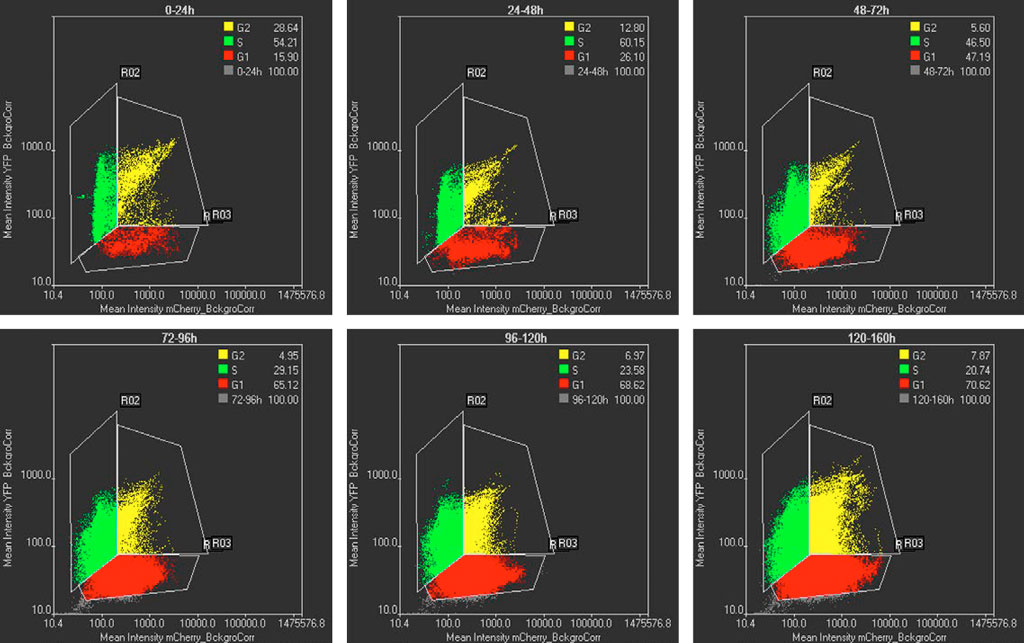
Abbildung 5: Streudiagramme der mCherry- und YFP-Intensitäten in verschiedenen Zeitintervallen. Jeder Punkt im Streudiagramm stellt eine mit der TruAI-Technologie segmentierte Zelle dar. G1-, S- und G2-Zellen sind rot, grün bzw. gelb dargestellt. Der prozentuale Anteil der G1-S-G2-Populationen ist in den Inserts angegeben. Über jedem Diagramm sind die Zeitintervalle angegeben.
Zell-Tracking: Vergleich der TruAI-Technologie mit klassischen Methoden
Die mit den drei Methoden (Intensitätsschwelle, Randerkennung und TruAI-Technologie) segmentierten Zellen wurden mit denselben Tracking-Einstellungen des kinetischen scanR Moduls verfolgt. Zur Erstellung von Tabelle 2 wurden die Statistikfunktionen des scanR Systems verwendet. Die in der Tabelle dargestellten Ergebnisse beziehen sich auf ein einziges Sehfeld über einen Zeitraum von 6 Tagen.
Tabelle 2: Vergleich der Anzahl der verfolgten Zellen pro Methode
| Zeitpunkte | Anzahl der verfolgten Zellen | ||
|---|---|---|---|
| Intensitätsschwelle | Randerkennung | TruAI-Technologie | |
| Mindestens 24 h | 302 | 1030 | 1965 |
| Mindestens 48 h | 16 | 126 | 426 |
| Mindestens 72 h | 0 | 27 | 72 |
| Mindestens 96 h | 0 | 0 | 5 |
Die Tabelle zeigt, dass die Verwendung der TruAI-Technologie für die Segmentierung die nachfolgenden Tracking-Ergebnisse deutlich verbessert. Bei diesem Datensatz können bei einer durchschnittlichen Anzahl von 200 Zellen pro Sehfeld zum Zeitpunkt von 24 h (Tabelle 1) mehr als 70 Zellen über drei Tage verfolgt werden, und es ist sogar möglich, Zellen über 4 Tage oder länger zu verfolgen. Die Möglichkeit, diese große Anzahl von Zellen über so lange Zeiträume zu verfolgen, ist wichtig, weil sie die Untersuchung der Zellzyklusdynamik über viele Zellteilungen hinweg auf statistisch aussagekräftige Weise ermöglicht.
Analyse von Zell-Tracks
Für jeden vom scanR System erzeugten Track können zahlreiche Parameter extrahiert werden.
- Zur Identifizierung der längsten Tracks kann der Parameter „Lifetime“ (Lebensdauer) in einem Histogramm aufgetragen werden, und es kann ein Gate für Tracks über 24 Stunden erstellt werden (Abbildung 6a).
- Um die Spuren weiter zu filtern, können die Parameter „First (time)“ und „Last (time)“ in einem Streudiagramm dargestellt werden (Abbildung 6b). Auf diese Weise können wir die Zellen identifizieren, die nur in den ersten Tagen verfolgt wurden (ES-Zellen mit kurzen Zykluszeiten), sowie die Zellen, die nur in den letzten Tagen verfolgt wurden (differenzierte Zellen mit langen Zellzykluszeiten), und die Zellen, die von Anfang bis Ende des Zeitablaufs verfolgt wurden (Zellen, bei denen der Differenzierungsprozess überwacht wird). Sobald diese Tracks identifiziert sind, können die Intensitäten in mCherry und YFP über die Zeit aufgetragen werden, um quantitative Informationen über die Dynamik des Zellzyklus zu erhalten (Abbildung 6 c, d, e).
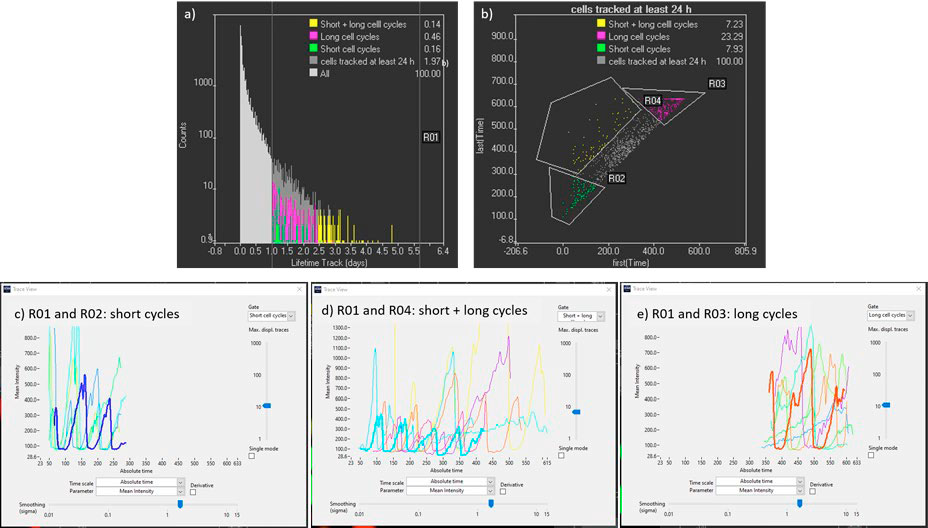
Abbildung 6: a) „Lifetime“-Histogramm zum Filtern langer Tracks (R01). b) Streudiagramm mit Anfangs- und Endzeitpunkt der Tracks, um Zellen zu identifizieren, die vom Anfang bis zum Ende des Zeitraums verfolgt wurden (R01 und R04). c), d) und e) Kinetische Tracks von mCherry-Zellen, die mehr als 24 Stunden verfolgt wurden, mit frühen Anfangs- und Endpunkten, mit frühen Anfangs- und späten Endpunkten bzw. mit späten Anfangs- und Endpunkten. Die Zeitpunkte liegen im Abstand von 15 Minuten.
Aus Abbildung 6c bis 6e geht hervor, dass die Dauer des mCherry-Signals (G1-Stadium) mit der Zeit zunimmt, von etwa 2 Stunden bei ES-Zellen bis zu 24 Stunden, wenn sich die Zellen differenziert haben. Für die eingehende Analyse der Zellzyklusdynamik können Tracks der Regionen R01 und R04 ausgewählt werden, in denen die Transformation von ES-Zellen in differenzierte Zellen über vier oder mehr Zellteilungen hinweg verfolgt wird (Abbildung 6d).
Abbildung 7 zeigt das Monitoring einer sich differenzierenden ES-Zelle über fünf Zellzyklen. In Abbildung 7a ist die Position der ES-Zelle in ihrer Kolonie zum Zeitpunkt 52 (13 h) dargestellt, zusammen mit ihrem Track bis zum Zeitpunkt 422 (105 h). In Abbildung 7b ist ein Schnappschuss zum Zeitpunkt 280 (70 h) dargestellt. Es ist zu beachten, dass die Intensität dieser Zelle extrem schwach ist und mit den klassischen Methoden nicht entdeckt werden konnte. In Abbildung 7c ist derselbe Schnappschuss zu sehen, allerdings mit dem TruAI-Wahrscheinlichkeitsbild. Die Wahrscheinlichkeitsintensität der verfolgten Zelle ist genauso hoch wie die der übrigen Zellen. Die Abbildungen 7d und 7e zeigen eine Galerie der verfolgten Zelle und die zugehörigen Fluoreszenz- bzw. TruAI-Wahrscheinlichkeiten alle 15 Minuten. Es ist zu beachten, dass die Gesamtintensität bei Beendigung der G1-Phase (rote Farbe) immer sehr schwach ist. Abbildung 7f zeigt die Fluoreszenzintensitätsschwankungen in mCherry (oben) und YFP (unten), aus denen die Zellzyklusperioden der Phasen G1, S, G2 und M extrahiert werden können.
Aus diesen Daten geht hervor, dass diese spezielle Zelle im Laufe der Zeit immer mehr Zeit im G1-Stadium und weniger Zeit in den Phasen S und G2 verbringt. Das M-Stadium ist immer sehr kurz; seine genaue Position zeigt sich als Verringerung des mCherry-Signals und als drastischer Abfall des YFP-Signals.
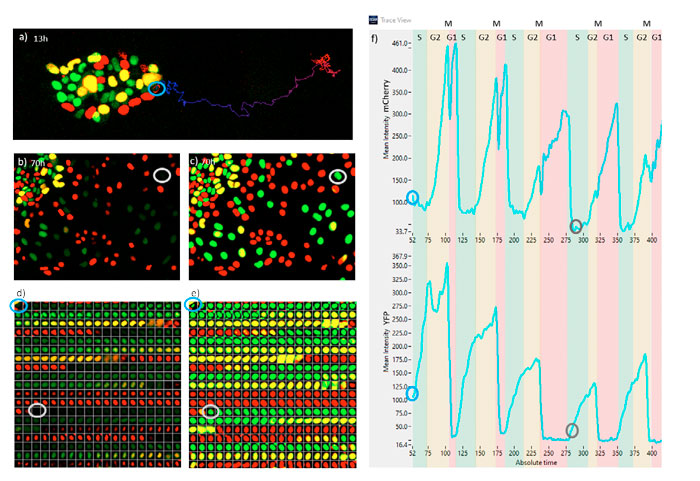
Abbildung 7: a) Fluoreszenz-Schnappschuss nach 13 h. b) Fluoreszenz-Schnappschuss nach 70 h. c) TruAI-Schnappschuss nach 70 h. d) Fluoreszenz-Zeitraffer-Sequenz der verfolgten Zelle in 15-Minuten-Intervallen. e) TruAI-Zeitraffer-Sequenz der verfolgten Zelle in 15-Minuten-Intervallen. f) Fluoreszenz-Tracks der verfolgten Zelle im mCherry- und im YFP-Kanal, die die Zeiträume der verschiedenen Zellzyklusphasen anzeigen. Die Zeitabstände entsprechen 15 Minuten. Die blauen und grauen Markierungen in a–f zeigen dieselbe Zelle nach 13 h bzw. 70 h.
Zusammenfassung
Dieser neuartige Deep-Learning-Ansatz zur Analyse der Zellzyklusdynamik während der Differenzierung von ES-Zellen in Kombination mit dem kinetischen Modul des scanR Systems für High-Content-Screening ermöglicht die Erfassung hochgradig reproduzierbarer, quantitativer und statistisch aussagekräftiger Daten darüber, wie sich die Zellzyklusdynamik in einzelnen Zellen während der Differenzierung verändert. Es können tausende von Zellen, die sich in Form und Morphologie verändern, erkannt und über Tage der Differenzierung durch Fluoreszenzmessungen verfolgt werden. Dies ist ein leistungsfähiges Tool zur Untersuchung der Zellzyklusdynamik in der frühen Entwicklung und Reprogrammierung sowie bei Zellen, die eine Zelltransformation durchlaufen (Malignität), bei der die Zellzyklusregulierung eine zentrale Rolle spielt.
Literaturnachweis
Padgett, J. und Santos, S.D.M. 2020. „From clocks to dominoes: lessons on cell cycle remodeling from embryonic stem cells.“ FEBS Letters. 10.1002/1873-3468.13862.
Araujo, A.R., Gelens, L., Sheriff, R.S.M. und Santos, S.D.M. 2016. „Positive feedback keeps duration of mitosis temporally insulated from upstream cell cycle events.“ Molecular Cell 64, 362–375.
Sakaue-Sawano et al. 2008. „Visualizing Spatiotemporal Dynamics of Multicellular Cell-Cycle Progression.“ Cell 132, 487–498.
Sakaue-Sawano et al. 2017. „Genetically Encoded Tools for Optical Dissection of the Mammalian Cell Cycle.“ Molecular Cell 68, 626–640.
Woerdemann, M. und Genenger, M. 2020. „TruAI™ Technology with Deep Learning for Quantitative Analysis of Fluorescent Cells with Ultra-Low Light Exposure.“ Olympus Application Note. www.olympus-lifescience.com/en/resources/white-papers/ultra-low_light_exposure_analysis/
Autoren
Joe Pagdett und Silvia Santos
Quantitative Stem Cell Biology Lab, The Francis Crick Institute, 1 Midland Road, NW1 1AT, London, UK
Manoel Veiga Gutierrez
Olympus Soft Imaging Solutions GmbH, Johann-Krane-Weg 39, 48149 Münster, Deutschland
Verwendete Produkte
wurde erfolgreich zu Ihren Lesezeichen hinzugefügt
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
Not Available in Your Country
Sorry, this page is not
available in your country.