Monitorización de la dinámica del ciclo celular durante la diferenciación de células madre usando la tecnología de deep learning TruAI™ del sistema scanR
Introducción
Durante el desarrollo temprano de los mamíferos, las células madre embriónicas (ESC) se someten a una especificación celular y dan lugar a todas las capas germinales embrionarias, que en último lugar generan diversos tipos de células en el organismo adulto. Las ESC se caracterizan por su increíble habilidad para auto-renovarse y proliferar, y cuentan con ciclos de división cortos debido a las fases GAP truncadas (G1 y G2). Durante la diferenciación celular, ocurren eventos celulares distintivos: cambios celulares en su morfología, crecimiento de tamaño multiplicado por diez, cambio del índice núcleo-citoplasma y cambio de forma por una forma plana y alargada; se activan programas específicos de expresión genética específicos para la grasa y se producen modificaciones en la cromatina global. Además, y muy importante, el ciclo celular se ralentiza. las células somáticas diferenciadas tienen fases G1 y G2 largas, verificación estricta de punto de control y ciclos de divisiones bien regulados (Padgett and Santos 2020) (Figura 1).
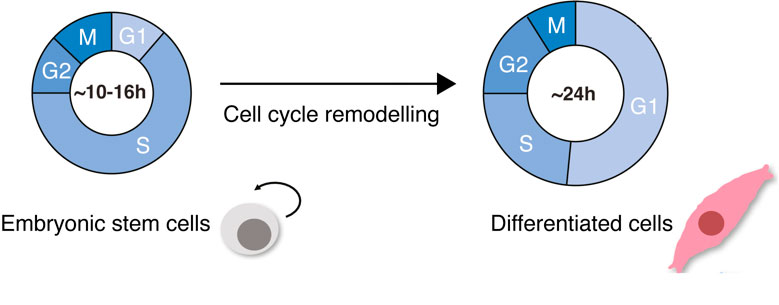
La remodelación del ciclo celular no es un fenómeno que solo ocurra en la diferenciación de ESC. Los cambios en la dinámica del ciclo celular se producen frecuentemente en biología; por ejemplo, cuando las células se someten a una regeneración después de infecciones virales o durante una malignidad. Por lo tanto, el estudio de los cambios de la dinámica celular en el contexto de la diferenciación celular es importante para entender los mecanismos potencialmente conservados del control de ciclo celular. Además, tendrá un impacto en nuestra comprensión del equilibrio entre los estados de salud y enfermedad.
Los cambios durante la diferenciación pueden monitorizarse en células únicas midiendo la actividad de las proteínas codificadas genéticamente que participan en las distintas fases del ciclo celular (Araujo et al. 2016; Sakaue-Sawano et al. 2008). Uno de estos sensores es el indicador de ciclo celular basado en ubiquitinación fluorescente FUCCI (CA), desarrollado por Miyawaki y sus compañeros (Sakaue-Sawano et al. 2017), que monitoriza la degradación de Cdt1 y Geminin, dos proteínas importantes del ciclo celular, produciendo una separación nítida de tres colores diferentes de las fases G1, S y G2 (rojo, verde y amarillo) que pueden observarse en los canales YFP y mCherry de un microscopio (Figura 2).
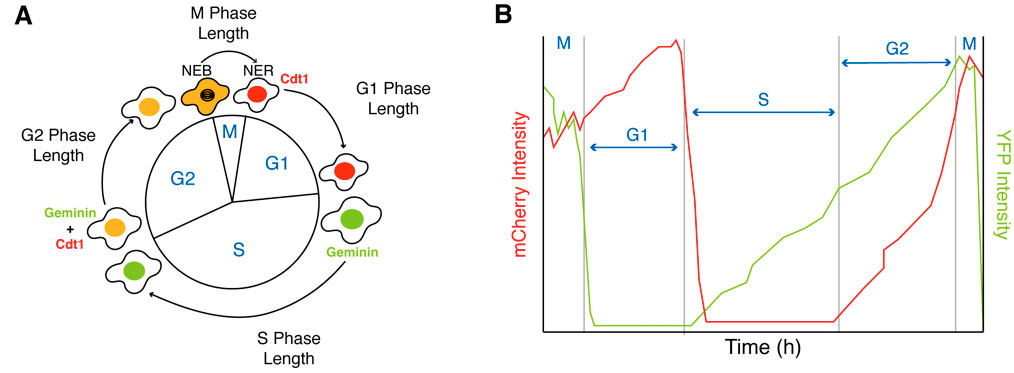
Figura 2. Medición de las fases G1, S, G2 y M del ciclo celular usando el sensor FUCCI(CA). (A) Esquema del sensor FUCCI(CA). (B) Ejemplo de trazas de la duración de las fases G1, S, G2 y M basadas en niveles de expresión de Cdt1 (canal mCherry) y Geminin (canal YFP).
Sin embargo, la monitorización de la dinámica del ciclo celular durante la diferenciación celular presenta algunos desafíos de procesamiento de imágenes y análisis de datos. El procesamiento de imágenes de células vivas a largo plazo requiere una temperatura estable, un control de CO2, iluminación suave para minimizar la fototoxicidad y rutinas de enfoque automático robustas para garantizar imágenes de buena calidad durante todo el proceso de lapso de tiempo.
Para el análisis, el mayor desafío es la segmentación y el posterior trazado de las células individuales a bajas intensidades de fluorescencia y altas condiciones de confluencia durante el movimiento, la división y el cambio de forma de las ESC durante la diferenciación.
Todos estos desafíos pueden superarse con el microscopio scanR de epifluorescencia de alto contenido de Olympus, equipado con una incubadora cellVivo, hardware de compensación de deriva Z (ZDC) para el enfoque automático, tecnología de deep learning TruAI™ para la segmentación y un módulo cinético para trazado.
Objetivos
En esta nota de aplicación, demostramos que la tecnología TruAI puede mejorar considerablemente la detección y el trazado de las ESC individuales a lo largo del procesamiento de imágenes a plazo largo con el sensor FUCCI(CA). Esto resulta especialmente valioso con células que no tienen marcador nuclear para trazado, ya que evita la iluminación y la fototoxicidad innecesarias.
Para ello, se emplea la tecnología TruAI para generar un modelo de red neuronal profunda (DNN) que puede identificar con fiabilidad la posición de las ESC pluripotentes y las células diferenciadas en todos los puntos temporales, incluso cuando la intensidad de fluorescencia es muy débil. Esto permite realizar una monitorización fiable de las fases G1, S y G2 de las poblaciones celulares en el tiempo. Posteriormente, se aplica el módulo cinético scanR para hacer el seguimiento de miles de células, evaluar sus trazas para periodos de tiempo largos y obtener información dinámica cuantitativa sobre los cambios en el ciclo celular a nivel de célula individual.
Para demostrar la versatilidad del sistema scanR como herramienta de investigación, todos los datos mostrados, con gráficos, diagramas de dispersión, galerías de objetos, pistas y trazas cinéticas son generados usando solo el software scanR.
Configuración experimental
Las células madres embriónicas humanas H1 (hESC) con expresión del sensor FUCCI(CA) son tratadas con 50 ng/ml de proteína morfológica ósea 3 (BMP4) para guiar la diferenciación en la ascendencia mesoderma. Posteriormente, estas células H1 se visualizan usando el microscopio scanR con una lente UPLANSAPO 10X en los canales mCherry e YFP cada 15 minutos durante 6 días.
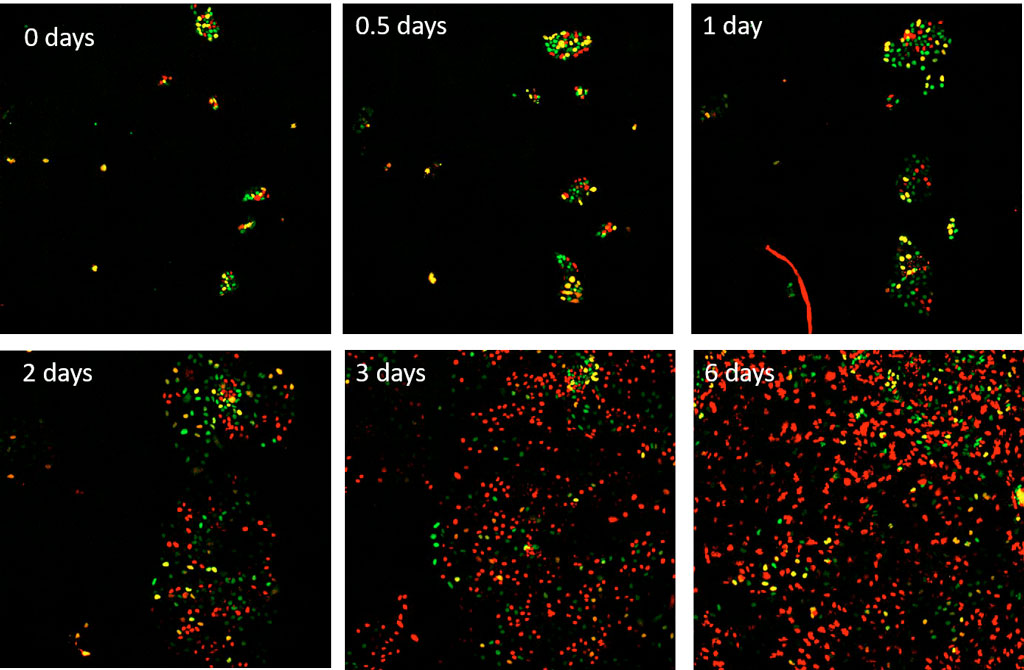
Figura 3. Serie temporal en 6 días de células hESC H1 con expresión del sensor FUCCI(CA), que fueron estimuladas con BMP4 al inicio del procesamiento de imágenes. Los colores rojo, verde y amarillo se corresponden con las fases G1, S y G2, respectivamente.
Desarrollo de un DNN para detectar células para condiciones de procesamiento de imágenes de células vivas
Una única célula con expresión del sensor FUCCI(CA) puede mostrar grandes variaciones en la intensidad de fluorescencia al ser monitorizada durante varios días. En particular, la intensidad total puede ser muy baja en la transición de la fase G1 a S (Figura 2b). Esto puede hacer que los algoritmos de trazado fallen, ya que las pistas se rompen en puntos temporales en los cuales las células son difíciles de detectar.
Para conseguir el trazado celular en varios días para el mayor número de células posible, es necesario desarrollar un modelo DNN capaz de detectar células a intensidades muy débiles. En un documento técnico anterior, demostramos que esto es posible usando pares de imágenes de exposición larga e iluminación intensa de células fluorescentes vs. exposición corta e iluminación débil (Woerdemann 2020). En resumen, podríamos decir que las imágenes de exposición larga se utilizan para la segmentación de las células y se genera una máscara. Esta máscara se utiliza como verdad terreno para formar el modelo DNN para detectar las células en los niveles de exposición baja.
Alternativamente, el modelo DNN puede formarse realizando anotaciones manuales en el conjunto de datos que va a analizarse. Para esta tarea, podemos usar el software cellSens™ de Olympus. Los modelos DNN desarrollados en el software cellSens pueden importarse al software scanR. En esta nota de aplicación, hemos usado el último método manual y hemos proporcionado cientos de anotaciones de células de intensidades variables en distintos puntos temporales para formar el modelo DNN.
Segmentación: Comparación de la tecnología de deep learning TruAI vs. métodos clásicos
Una vez desarrollado, el modelo DNN se aplica a los canales individuales de fluorescencia mCherry e YFP. Esto crea un mapa de probabilidad de IA en píxeles para cada canal. Cuanto más alta sea la probabilidad de IA en un píxel, más alta será la certeza de que el píxel pertenece a una célula. Posteriormente, los mapas de probabilidad de IA de los dos canales se suman y se realiza la segmentación aplicando un umbral de intensidad a esta suma (Figura 4e y 4f).
Para comparar los resultados con los métodos clásicos, los canales de fluorescencia Cherry e YFP se corrigen en segundo plano usando un algoritmo de esfera rodante, se suman y la suma se segmenta usando un umbral de intensidad (Figura 4c) o un método de detección de bordes (Figura 4d). Todos los pasos de procesamiento de imágenes descritos anteriormente se realizan en el software de análisis scanR.
Las herramientas estadísticas del sistema scanR se utilizan para crear la Tabla 1, en la cual los resultados hacen referencia a un campo único de visión para un periodo de 6 días.
Tabla 1. Comparación de número de células segmentadas por método
| Puntos temporales | Métodos de segmentación | ||
|---|---|---|---|
| Umbral de intensidad | Detector de bordes | Tecnología TruAI | |
| 0 h | 78 | 103 | 93 |
| 24 h | 209 | 188 | 240 |
| 48 h | 421 | 281 | 475 |
| 72 h | 847 | 654 | 986 |
| 96 h | 1480 | 1330 | 1590 |
| 160 h | 1340 | 1460 | 1610 |
|
Suma de todos los fotogramas
0-160 h | 504131 | 573179 | 659199 |
Una comparación única de la imagen de fluorescencia con la imagen de probabilidad de IA (Figuras 4a y 4b) revela que el enfoque TruAI tiene una sensibilidad más alta para detectar cualquier tipo de célula. Esta afirmación se ve respaldada por los resultados resumidos en la Tabla 1 junto con todo el lapso de tiempo. El método de umbral de intensidad clásico no detecta las células fluorescentes débiles y no puede determinar su forma correcta (Figura 4c). El método de detección de bordes mejora el contorno de las células detectadas, pero omite muchas células tenues (Figura 4d). La tecnología de deep learning TruAI detecta las células tenues con fiabilidad y sus límites están bien definidos (Figuras 4e y 4f).
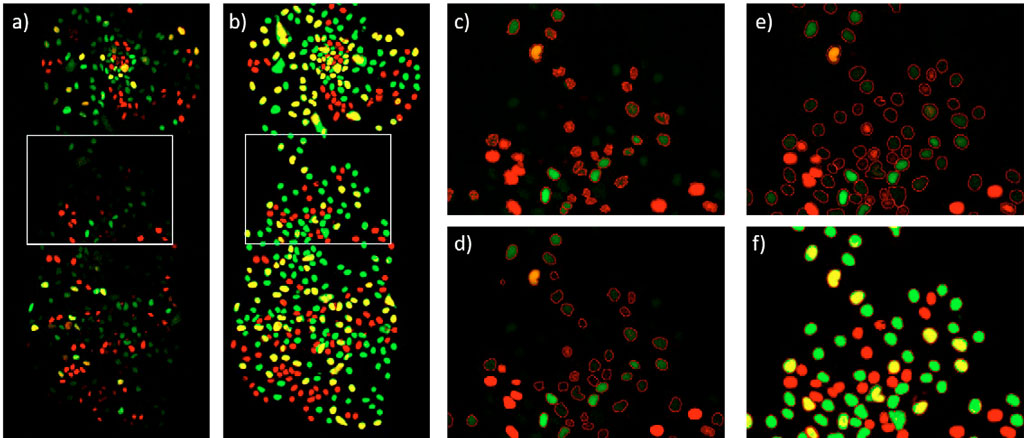
Figura 4: Colonia de células ESC en el punto temporal 48 h. a) Canales de fluorescencia mCherry (rojo, fase G1) e YFP (verde, fase S). La fase G2 se visualiza como una combinación de mCherry e YFP (amarillo). b) Probabilidad TruAI en mCherry (rojo) e YFP (verde). Las células con una probabilidad alta de IA en los dos canales aparecen de color amarillo. c) Segmentación usando un umbral de intensidad en la suma de los canales de fluorescencia mCherry e YFP d) Segmentación usando un detector de bordes en la suma de los canales de fluorescencia mCherry e YFP e) Segmentación en la suma de las intensidades de probabilidad TruAI, superpuesta con la imagen de fluorescencia f) igual que e) pero superpuesta por la probabilidad TruAI.
Análisis de poblaciones celulares en el tiempo
Las intensidades mCherry e YFP de las células segmentadas usando la tecnología TruAI se muestran en un diagrama de dispersión a diversos intervalos de tiempo (Figura 5). Las células que solo tienen intensidad mCherry se corresponden con la fase G1, las células que muestran solo la intensidad YFP se corresponden con la fase S y las células que tienen intensidades en los canales se corresponden con las células en el estado G2. La Figura 5 muestra que en una fase temprana la mayoría de células están en el estado S, mientras que en las fases más tardías las células están en el estado G1. Este cambio poblacional coincide con los perfiles de ciclo celular previstos para células pluripotentes y diferenciadas, respectivamente (Figura 1).
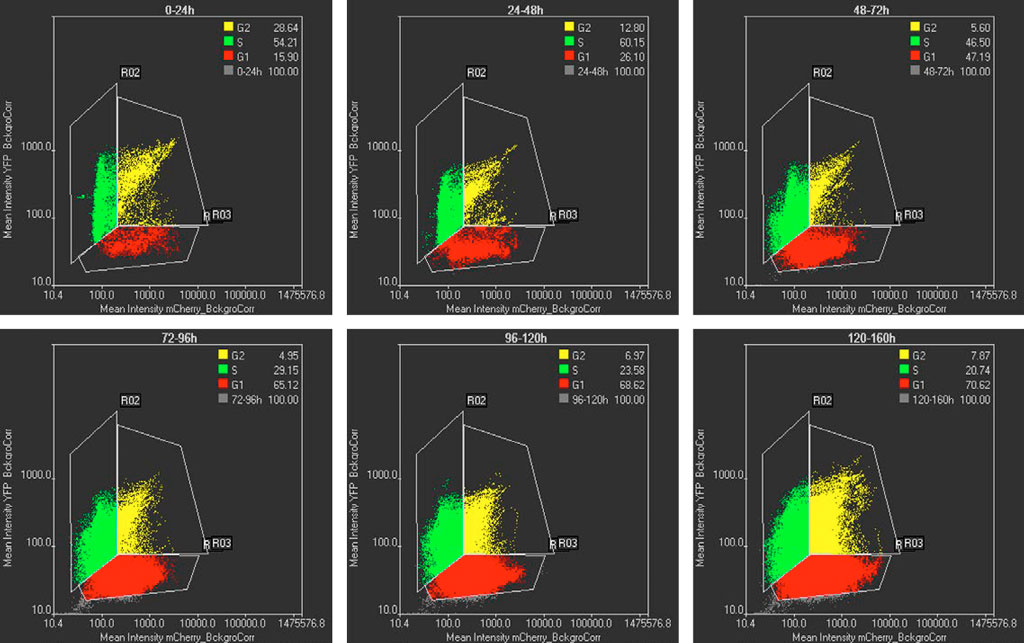
Figura 5: Diagramas de dispersión de intensidades mCherry vs. YFP en distintos intervalos de tiempo. Cada punto en el diagrama de dispersión representa una célula segmentada por la tecnología TruAI. Las células G1, S y G2 están representadas por los colores rojo, verde y amarillo, respectivamente. El % de las poblaciones G1-S-G2 se muestra en las inserciones. Los intervalos de tiempo se muestran en la parte superior de cada gráfico.
Trazado celular: Comparación de la tecnología TruAI vs. los métodos clásicos
Las células segmentadas con los tres métodos (umbral de intensidad, detector de bordes y tecnología TruAI) se trazaron usando los mismos ajustes de trazado que en el módulo cinético scanR. Las herramientas estadísticas del sistema scanR se utilizan para crear la Tabla 2, donde los resultados se refieren a un campo de visión único para un periodo de 6 días.
Tabla 2. Comparación de número de células trazadas por método
| Puntos temporales | Número de células trazadas | ||
|---|---|---|---|
| Umbral de intensidad | Detector de bordes | Tecnología TruAI | |
| 24 h o más | 302 | 1030 | 1965 |
| 48 h o más | 16 | 126 | 426 |
| 72 h o más | 0 | 27 | 72 |
| 96 h o más | 0 | 0 | 5 |
La tabla muestra que la utilización de tecnología TruAI para la segmentación mejora claramente los resultados de trazado posteriores. Para este conjunto de datos, considerando un promedio de 200 células por campo de visión en el momento de 24 h (Tabla 1), pueden trazarse más de 70 células durante 3 días e incluso pueden trazarse células durante 4 días o más. La capacidad de trazar este número de células tan alto durante periodos de tiempo tan prolongados es importante, ya que permite realizar un estudio de la dinámica del ciclo celular en muchas divisiones celulares de forma estadística para obtener información valiosa.
Análisis de trazados celulares
Para cada trazado generado por el sistema scanR, puede extraerse una amplia gama de parámetros.
- Para identificar los trazados más largos, el parámetro "ciclo de vida" puede desglosarse en un histograma y puede crearse una compuerta para los trazados durante 24 h (Figura 6a).
- Para filtrar los trazados, los parámetros "Primer (momento) y "Último (momento)" pueden representarse en un diagrama de dispersión (Figura 6b). Esto nos puede ayudar a identificar las células trazadas solo durante los primeros días (células ESC con tiempos de ciclo cortos), las células trazas solo en los últimos días (células diferenciadas con tiempos de ciclo celular largos) y las células trazadas desde el principio hasta el final del lapso de tiempo (células en las que se monitoriza el proceso de diferenciación). Después de identificar los trazados, las intensidades de mCherry e YFP pueden desglosarse por tiempo para obtener información cuantitativa sobre la dinámica del ciclo celular (Figuras 6c, d y e).
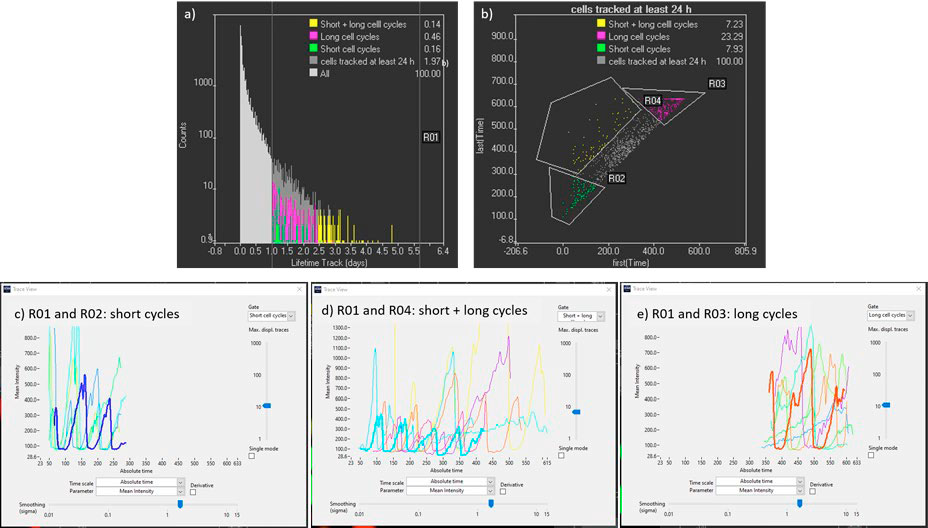
Figura 6. a) Histograma de ciclo de vida del trazado usado para filtrar trazados largos (R01). b) Diagrama de dispersión con punto temporal de inicio y fin de los trazados para identificar las células trazadas desde el principio hasta el final del lapso de tiempo (R01 y R04). c), d), y e) Trazas cinéticas de células mCherry trazadas durante más de 24 h con puntos de inicio y fin tempranos, con puntos de inicio tempranos y puntos de fin tardíos, y con puntos de inicio y fin tardíos respectivamente. Los puntos temporales tienen una separación de 15 minutos entre ellos.
De la Figura 6c a 6e, se observa que la duración de la señal mCherry (estado G1) aumenta con el paso del tiempo, de aproximadamente 2 horas para las células ESC a 24 h después de diferenciar las células. Para el análisis profundo de la dinámica del ciclo celular, se pueden seleccionar trazados de la región R01 y R04, en las cuales se monitoriza la transformación de ESC a células diferenciadas durante cuatro o más divisiones celulares (Figura 6d).
La Figura 7 representa la monitorización de una célula ESC sometida a diferenciación durante cinco ciclos celulares. En la Figura 7a, la posición de ESC en su colonia se muestra en el punto temporal 52 (13 h), junto con su trazado hasta el punto temporal 422 (105 h). En la Figura 7b, se muestra una instantánea en el momento 280 (70 h). Podemos observar que la intensidad de esta célula es muy débil y que no se ha detectado usando los métodos clásicos. En la Figura 7c, se muestra la misma instantánea pero con la imagen de probabilidad TruAI. La intensidad de probabilidad de la célula trazada es tan alta como para el resto de células. Las Figuras 7d y 7e representan una galería de la célula trazada en los puntos temporales cada 15 minutos para fluorescencia y probabilidad TruAI, respectivamente. Podemos observar que al finalizar la fase G1 (color rojo), la intensidad total siempre es muy débil. La Figura 7f representa las oscilaciones de intensidad de fluorescencia mCherry (arriba) e YFP (abajo), de las cuales pueden extraerse los periodos del ciclo celular de los estados G1, S, G2 y M.
A partir de estos datos, podemos observar que esta célula especifica pasa más tiempo en el estado G1 y menos tiempo en las fases S y G2 a medida que avanza el tiempo. El estado M siempre es muy corto; su posición exacta aparece como una bajada en la señal mCherry y una caída acusada en la señal YFP.
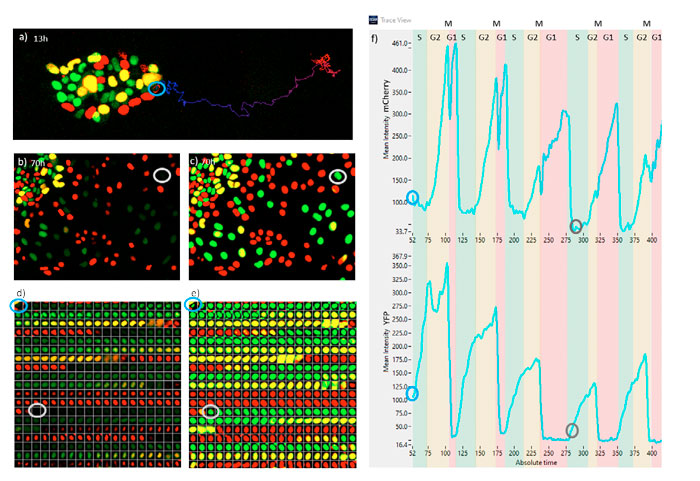
Figura 7. a) Instantánea de fluorescencia en el punto temporal 13 h. b) Instantánea de fluorescencia en el punto temporal 70 h. c) Instantánea TruAI a las 70 h. d) Secuencia de lapso de tiempo de fluorescencia de la célula trazada a intervalos de 15 minutos. e) Secuencia de lapso de tiempo TruAI de la célula trazada a intervalos de 15 minutos. f) Trazas de fluorescencia de mCherry e YFP de la célula trazada indicando los periodos de las diversas fases del ciclo celular. Los pasos temporales son de 15 min. Los puntos azul y gris en a–f indican la misma célula a las 13 h o 70 h, respectivamente.
Conclusión
Este innovador enfoque de deep-learning de analizar la dinámica del ciclo celular durante la diferenciación de ESC combinada con el módulo cinético del sistema de cribado de alto contenido scanR permite adquirir datos reproducibles, cuantitativos y estadísticamente valiosos sobre los cambios en la dinámica del ciclo celular en células individuales durante la diferenciación. Este enfoque permite reconocer y trazar miles de células que cambian de forma y morfología en varios días de diferenciación usando mediciones de fluorescencia. Se trata de una herramienta muy potente para estudiar la dinámica del ciclo celular en las fases tempranas de desarrollo y reprogramación, así como en las células sometidas a transformación celular (malignidad) en las que la regulación del ciclo celular cobra una gran importancia.
Referencias
Padgett, J., and Santos, S.D.M. 2020. “From clocks to dominoes: lessons on cell cycle remodeling from embryonic stem cells.” FEBS Letters. 10.1002/1873-3468.13862.
Araujo, A.R., Gelens, L., Sheriff, R.S.M., and Santos, S.D.M. 2016. “Positive feedback keeps duration of mitosis temporally insulated from upstream cell cycle events.” Molecular Cell 64, 362–375.
Sakaue-Sawano et al. 2008. “Visualizing Spatiotemporal Dynamics of Multicellular Cell-Cycle Progression.” Cell 132, 487–498.
Sakaue-Sawano et al. 2017. “Genetically Encoded Tools for Optical Dissection of the Mammalian Cell Cycle.” Molecular Cell 68, 626–640.
Woerdemann, M., and Genenger, M. 2020. “TruAI™ Technology with Deep Learning for Quantitative Analysis of Fluorescent Cells with Ultra-Low Light Exposure.” Nota de aplicación de Olympus. www.olympus-lifescience.com/en/resources/white-papers/ultra-low_light_exposure_analysis/
Autores
Joe Pagdett y Silvia Santos
Quantitative Stem Cell Biology Lab, The Francis Crick Institute, 1 Midland Road, NW1 1AT, London, UK
Manoel Veiga Gutierrez
Olympus Soft Imaging Solutions GmbH, Johann-Krane-Weg 39, 48149 Muenster, Alemania
Productos usados para esta aplicación
se ha añadido correctamente a sus marcadores
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
Not Available in Your Country
Sorry, this page is not
available in your country.