scanR 시스템의 TruAI™ 딥러닝 기술을 사용한 줄기세포 분화시의 세포주기 역학 모니터링
서문
배아줄기세포(ESC)는 초기 포유동물 발달 중에 세포 특성화 과정을 거치고 배아의 배엽을 모두 발생시켜 결과적으로 성체 생물에서 수많은 세포 유형을 생성합니다. 배아줄기세포는 생략된 GAP 단계(G1기, G2기)로 인해 분열 주기가 짧고 자가 재생 및 증식하는 놀라운 능력이 특징입니다. 세포가 분화될 때는 특징적인 세포적 사건이 발생합니다. 세포는 약 10배 크기로 성장하고 핵 대 세포질(N/C) 비율이 바뀌며 평평하고 길어지면서 변형됩니다. 운명 특이적 유전자 발현 프로그램이 활성화되고 전체 염색질 변형이 발생합니다. 세포주기가 느려진다는 것이 중요합니다. 분화된 체세포는 G1기와 G2기가 길고, 체크포인트 조절이 엄격하며, 분열 주기가 잘 조정됩니다(Padgett 및 Santos, 2020년)(그림 1).
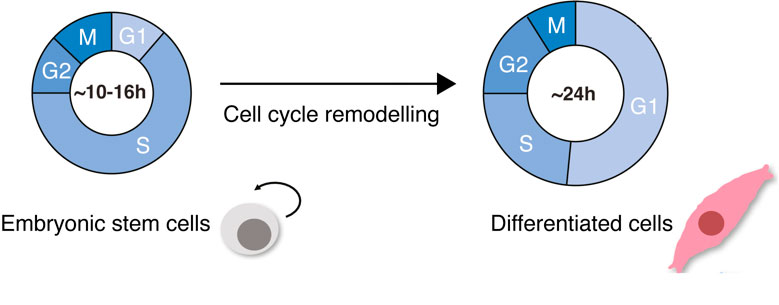
세포주기 재형성은 배아줄기세포 분화에서만 일어나는 현상이 아닙니다. 세포주기 역학의 변화는 생물학에서 자주 발생합니다. 그 예로, 세포가 바이러스 감염 이후나 악성 종양 발병 중에 재생하는 경우가 있습니다. 따라서 세포 분화의 맥락에서 세포주기 역학의 변화를 연구하는 것은 잠재적으로 보존된 세포주기 조절의 메커니즘을 이해하는 데 중요하며, 건강과 질병 상태 사이의 균형을 이해하는 데 영향을 미칠 것입니다.
분화 시의 변화는 세포주기의 여러 단계에 관여하는 유전자 암호화 단백질의 활성을 측정하여 단일 세포에서 모니터링할 수 있습니다(Araujo 외, 2016년, Sakaue-Sawano 외, 2008년). 이러한 센서 중 하나는 Miyawaki와 동료들(Sakaue-Sawano 외, 2017년)이 개발한 형광 유비퀴틴화 기반 세포주기 표시기 FUCCI(CA)입니다. 이는 두 가지 중요한 세포주기 단백질인 Cdt1 및 제미닌의 분해를 모니터링하여 현미경의 YFP 및 mCherry 채널에서 관찰할 수 있는 G1기, S기, G2기의 선명한 3가지 색상별 분해(적색, 녹색, 황색)를 일으킵니다(그림 2).
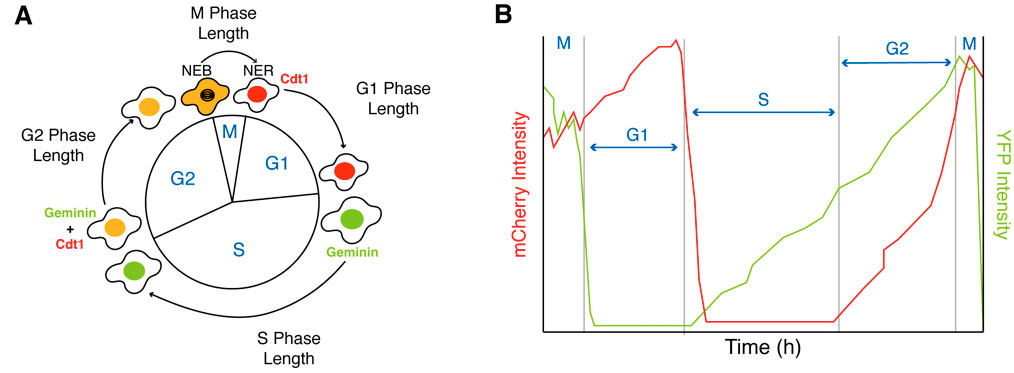
그림 2. FUCCI(CA) 센서를 사용한 세포주기의 G1기, S기, G2기, M기 측정. (A) FUCCI(CA) 센서의 구성도. (B) Cdt1(mCherry 채널) 및 제미닌(YFP 채널)의 발현 수준에 기반한 G1기, S기, G2기, M기 기간의 흔적 예시.
그러나 세포 분화 시의 세포주기 역학을 모니터링하면 이미징 및 데이터 분석 문제가 모두 나타납니다. 며칠에 걸친 장기간의 라이브셀 이미징에는 안정적인 온도와 이산화탄소 조절, 광독성을 최소화하기 위한 은은한 조명, 전체 타임랩스 과정에서 양질의 이미지를 보장하기 위한 강력한 자동 초점 루틴이 필요합니다.
분석 시 최대 문제는 배아줄기세포가 분화 시에 이동, 분열하고 급격하게 변형되는 동안 낮은 형광 강도와 높은 밀집 조건에서 단일 세포를 분할하고 차후 추적하는 것입니다.
이 모든 문제는 cellVivo 인큐베이터, 자동 초점을 위한 Z-드리프트 보정(ZDC) 하드웨어, 분할을 위한 TruAI™ 딥 러닝 기술, 추적을 위한 운동 모듈이 장착된 Olympus High-Content 형광 scanR 현미경으로 극복할 수 있습니다.
대물렌즈
이 애플리케이션 노트는 TruAI 기술을 적용할 때 FUCCI(CA) 센서 기반의 장기간 이미징을 통해 단일 배아줄기세포의 감지와 추적을 크게 향상할 수 있음을 보여줍니다. 이것은 추적을 위한 핵 마커가 없는 세포에서 특히 강력하며 불필요한 조명과 광독성을 방지합니다.
이를 달성하려면, TruAI 기술을 사용하여 모든 시점에, 심지어 형광 강도가 매우 약할 때도 만능 배아줄기세포와 분화된 세포의 위치를 충실하게 식별할 수 있는 심층 신경망(DNN) 모델을 생성합니다. 이로써 시간에 따른 세포 집단의 G1기, S기, G2기를 확실하게 모니터링할 수 있습니다. 그 뒤에 scanR 키네틱(Kinetic) 모듈을 적용하면 수천 개의 세포를 추적하고, 장기간의 세포 흔적을 평가하고, 단일 세포 수준에서 세포주기 변화에 대한 정량적 동적 정보를 얻을 수 있습니다.
연구 도구로서 scanR 시스템의 다용도성을 입증하기 위해 그래프, 산점도, 객체 갤러리, 추적, 운동 흔적을 포함하여 여기에 나온 모든 데이터를 scanR 소프트웨어만으로 생성했습니다.
실험 설정
FUCCI(CA) 센서를 발현하는 H1 인간배아 줄기세포(hESC)는 50ng/ml의 골형성 단백질 4(BMP4)로 처리되어 중내배엽 계통으로의 분화를 유도합니다. 그런 다음 6일 동안 15분마다 mCherry 및 YFP 채널에서 10X UPLANSAPO 렌즈가 장착된 scanR 현미경으로 이 H1 세포를 이미징합니다.
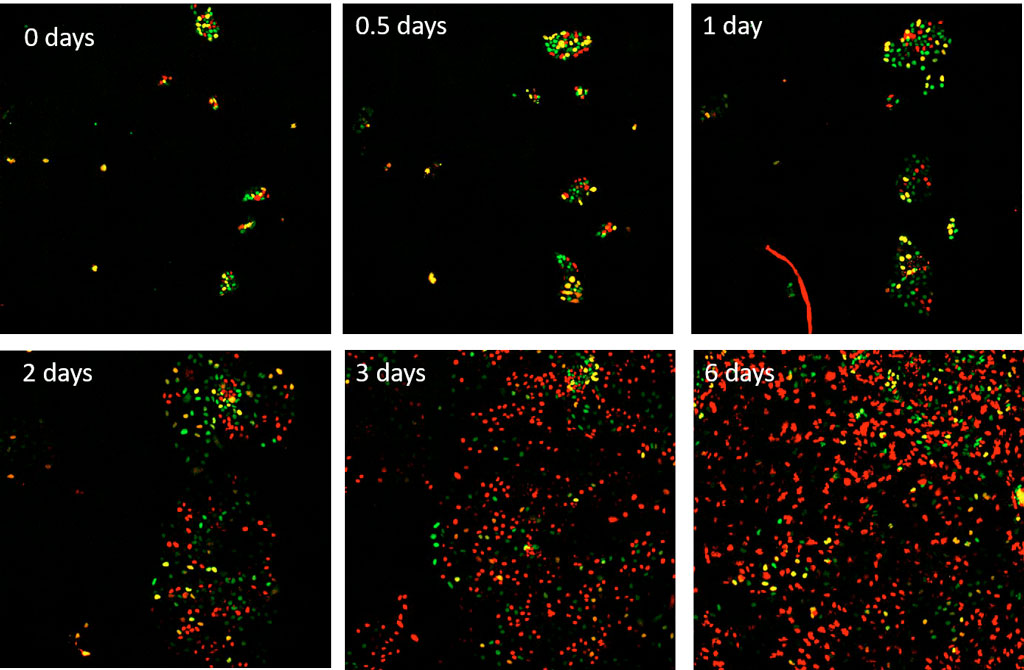
그림 3. 이미징 초반에 BMP4로 활성화된 FUCCI(CA) 센서를 발현하는 hESC H1 세포의 6일에 걸친 타임 시리즈. 적색, 녹색, 황색은 각각 G1기, S기, G2기에 해당합니다.
라이브셀 이미징 조건에서 세포를 감지하는 DNN 개발
FUCCI(CA) 센서를 발현하는 단일 세포를 며칠 동안 모니터링하는 경우 형광 강도의 강한 변화를 보일 수 있습니다. 특히 총 강도는 G1기에서 S기로의 전환 시 매우 낮을 수 있습니다(그림 2b). 따라서 세포를 감지하기 어려운 시점에는 추적이 중단되어 추적 알고리즘이 실패할 수 있습니다.
세포를 며칠 동안 최대한 많이 추적하려면 강도가 매우 약할 때도 세포를 감지할 수 있는 DNN 모델을 개발해야 합니다. 이전 백서에서는 형광 세포의 노출이 긴 강렬한 조명과 노출이 짧은 약한 조명의 이미지 쌍을 사용하여 이것이 가능함을 보여주었습니다(Woerdemann, 2020년). 간단히 말해서, 장노출 이미지는 세포 분할에 사용되며 마스크가 생성됩니다. 이 마스크는 낮은 노출 수준에서 세포를 감지하도록 DNN 모델을 훈련하기 위한 실측 자료로 사용됩니다.
한편, 분석할 데이터 세트에서 수동 주석을 수행하여 DNN 모델을 훈련할 수 있습니다. 이 작업에 Olympus의 cellSens™ 소프트웨어를 사용할 수 있습니다. cellSens 소프트웨어에서 개발한 DNN 모델을 scanR 소프트웨어로 가져올 수 있습니다. 이 애플리케이션 노트에서는 후자의 수동 방식을 사용하고 DNN 모델을 훈련하기 위해 다양한 강도의 세포에 대한 주석을 서로 다른 시점에 수백 개 제공했습니다.
분할: TruAI 딥러닝 기술과 기존 방식의 비교
DNN 모델은 개발 직후 개별 mCherry 및 YFP 형광 채널에 적용됩니다. 그러면 채널별로 픽셀 AI 확률 맵이 생성됩니다. 픽셀의 AI 확률이 높을수록 픽셀이 세포에 속할 신뢰도가 높아집니다. 그 후 두 채널의 AI 확률 맵이 합산되고 이 합계에 강도 역치를 적용하여 분할이 이루어집니다(그림 4e 및 4f).
이 결과를 기존 방식과 비교하기 위해 mCherry 및 YFP 형광 채널을 롤링 볼 알고리즘으로 배경 보정 및 합산하고 강도 역치(그림 4c) 또는 에지 검출법(그림 4d)을 사용하여 분할했습니다. 위에서 설명한 모든 이미지 처리 단계는 scanR 분석 소프트웨어에서 이루어집니다.
scanR 시스템의 통계 도구를 사용하여 6일 동안 하나의 단일 시야에서 결과를 참조하는 표 1을 작성했습니다.
표 1. 방식별로 분할한 세포 수 비교
| 시점 | 분할 방식 | ||
|---|---|---|---|
| 강도 역치 | 에지 검출기 | TruAI 기술 | |
| 0시간 | 78 | 103 | 93 |
| 24시간 | 209 | 188 | 240 |
| 48시간 | 421 | 281 | 475 |
| 72시간 | 847 | 654 | 986 |
| 96시간 | 1480 | 1330 | 1590 |
| 160시간 | 1340 | 1460 | 1610 |
|
모든 프레임의 요약
0~160시간 | 504131 | 573179 | 659199 |
형광 이미지와 AI 확률 이미지(그림 4a 및 4b)의 간단한 비교는 TruAI 접근 방식이 모든 유형의 세포를 감지할 때 감도가 더 높다는 것을 보여줍니다. 이는 전체 시간 경과와 함께 표 1에 요약된 결과로 뒷받침됩니다. 기존 강도 역치 방법은 약한 형광 세포를 감지하거나 올바른 모양을 분석하지 못합니다(그림 4c). 에지 검출 방식은 검출된 세포의 윤곽을 개선하지만, 여전히 어두운 세포를 놓치는 경우가 많습니다(그림 4d). TruAI 딥러닝 기술은 어두운 세포를 안정적으로 감지하며, 그 경계가 명확합니다(그림 4e 및 4f).
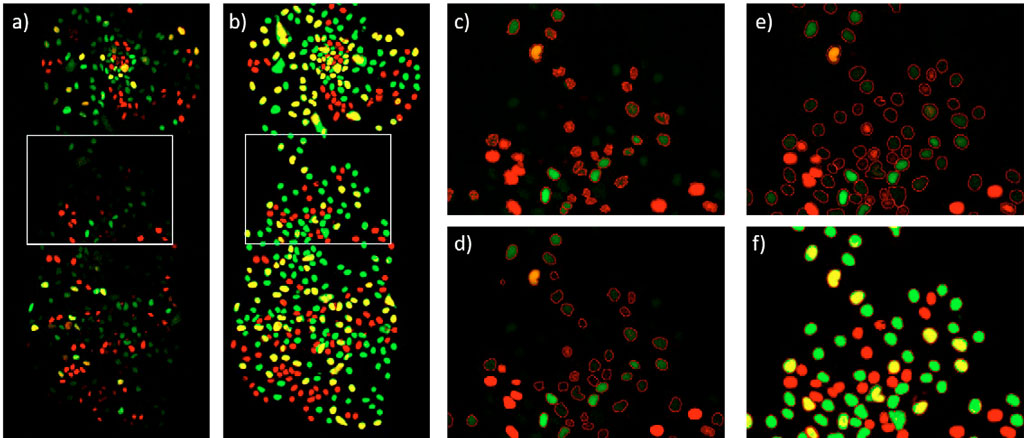
그림 4: 48시간 시점의 배아줄기세포 군체. a) mCherry(적색, G1기) 및 YFP(녹색, S기) 형광 채널. G2기는 mCherry와 YFP(황색)의 조합으로 표시됩니다. b) mCherry(적색) 및 YFP(녹색)의 TruAI 확률. 두 채널에서 모두 AI 확률이 높은 세포는 황색으로 표시됩니다. c) mCherry 및 YFP 형광 채널의 합계에서 강도 역치를 사용한 분할 d) mCherry 및 YFP 형광 채널의 합계에서 에지 검출기를 사용한 분할 e) 형광 이미지와 중첩된 TruAI 확률 강도 합계에서의 분할 f) e)와 동일하지만 TruAI 확률과 중첩됩니다.
시간에 따른 세포 개체군 분석
TruAI 기술을 사용하는 분할된 세포의 mCherry 및 YFP 강도는 시간 간격을 달리하여 산점도에 표시됩니다(그림 5). mCherry 강도만 있는 세포는 G1기에 해당하고, YFP 강도만 나타내는 세포는 S기에 해당하며, 두 채널에 모두 강도가 있는 세포는 G2 상태의 세포에 해당합니다. 그림 5는 대부분의 세포가 초기에는 S 상태인 반면, 후기에는 대부분의 세포가 G1 상태임을 보여줍니다. 이 개체군 이동은 만능 세포와 분화 세포 각각에 예상되는 세포주기 양상과 일치합니다(그림 1).
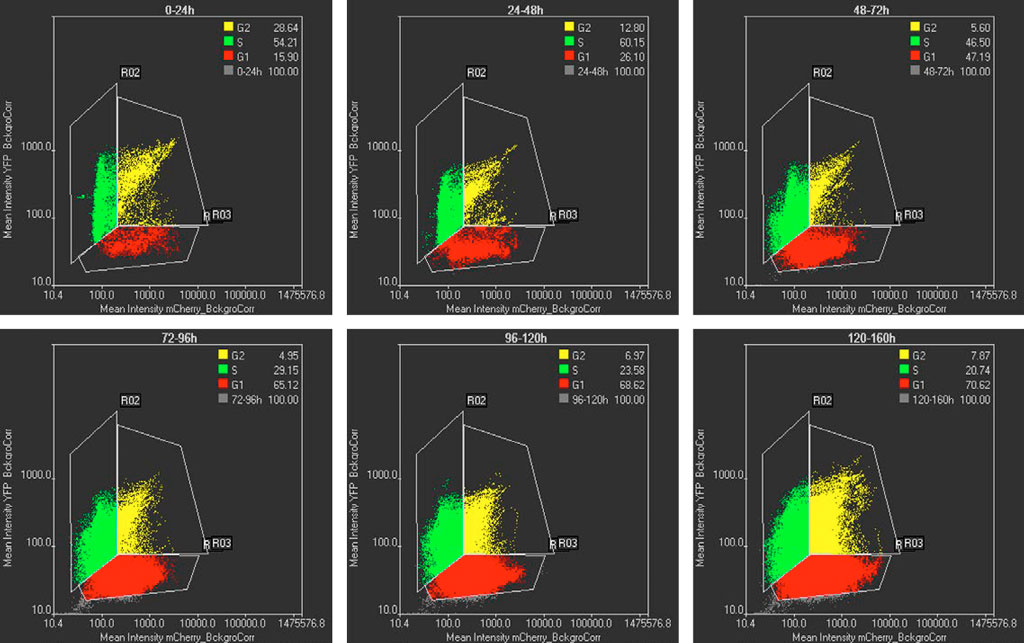
그림 5: 시간 간격을 달리할 때 mCherry 대 YFP 강도의 산점도. 산점도의 각 점은 TruAI 기술로 분할된 세포를 나타냅니다. G1, S 및 G2 세포는 각각 적색, 녹색 및 황색으로 표시됩니다. G1-S-G2 개체군의 백분율(%)은 인서트에 표시됩니다. 시간 간격은 각 그래프의 상단에 표시됩니다.
세포 추적: TruAI 기술과 기존 방식의 비교
세 가지 방식(강도 역치, 에지 검출기, TruAI 기술)으로 분할된 세포를 scanR 키네틱 모듈의 동일한 추적 설정으로 추적했습니다. scanR 시스템의 통계 도구를 사용하여 6일 동안 하나의 단일 시야에서 결과를 참조하는 표 2를 작성했습니다.
표 2. 방식별로 추적한 세포 수의 비교
| 시점 | 추적한 세포 수 | ||
|---|---|---|---|
| 강도 역치 | 에지 검출기 | TruAI 기술 | |
| 24시간 이상 | 302 | 1030 | 1965 |
| 48시간 이상 | 16 | 126 | 426 |
| 72시간 이상 | 0 | 27 | 72 |
| 96시간 이상 | 0 | 0 | 5 |
이 표는 분할에 TruAI 기술을 사용하면 후속 추적 결과가 확실하게 향상됨을 보여줍니다. 이 데이터 세트의 경우, 24시간 시점에서 시야당 평균 200개의 세포 수(표 1)를 고려하면 3일 동안 70개 이상의 세포를 추적할 수 있으며 4일 이상 세포를 추적하는 것도 가능합니다. 이렇게 많은 수의 세포를 오랫동안 추적하는 능력이 중요한 이유는 많은 세포 분열에 대한 세포주기 역학 연구를 통계적으로 유의미한 방식으로 가능케 하기 때문입니다.
세포 추적의 분석
scanR 시스템으로 생성된 각 추적에서 광범위한 매개변수를 추출할 수 있습니다.
- 최장 추적을 식별하기 위해 매개변수 "수명"을 히스토그램에 표시하고 24시간 이상의 추적에 대한 게이트를 생성할 수 있습니다(그림 6a).
- 추적을 추가로 필터링하기 위해 "초기(시점)" 및 "최종(시점)" 매개변수를 산점도에 표시할 수 있습니다(그림 6b). 이것은 처음 며칠간만 추적한 세포(주기 시간이 짧은 배아줄기세포), 마지막 며칠간만 추적한 세포(세포주기 시간이 긴 분화 세포), 처음부터 시간 경과의 끝까지 추적한 세포(분화 과정을 모니터링한 세포)를 식별하는 데 도움이 될 수 있습니다. 이러한 추적이 식별되면 mCherry 및 YFP의 강도를 시간에 따라 플로팅하여 세포주기 역학에 대한 정량적 정보를 얻을 수 있습니다(그림 6c, d, e).
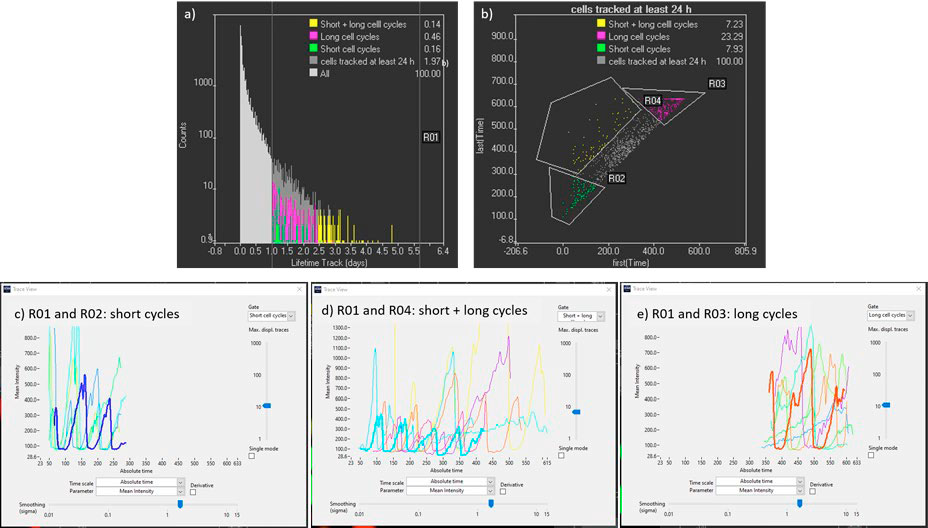
그림 6. a) 긴 추적(R01)을 필터링하는 데 사용한 추적 수명 히스토그램. b) 시간 경과의 시작부터 끝까지 추적한 세포를 식별하기 위한 추적 초기 및 최종 시점이 있는 산점도(R01 및 R04). c) , d) 및 e) mCherry 세포의 운동 흔적은 초기 시작점과 끝점, 이른 시작점과 늦은 끝점, 늦은 시작점과 끝점으로 각각 24시간 이상 추적했습니다. 시점은 15분 간격으로 표시됩니다.
그림 6c부터 6e까지에서, mCherry(G1 상태) 신호 지속시간은 배아줄기세포의 경우 약 2시간에서 세포가 분화되면 24시간으로 시간에 따라 증가하는 것으로 관찰됩니다. 세포주기 역학에 대한 심층 분석을 위해 배아줄기세포에서 분화 세포로의 변형을 4회 이상의 세포 분열을 통해 모니터링하는 R01 및 R04 영역의 추적을 선택할 수 있습니다(그림 6d).
그림 7은 5회의 세포주기에 걸쳐 분화를 겪고 있는 배아줄기세포 하나에 대한 모니터링을 나타냅니다. 그림 7a에서 배아줄기세포의 군체 내 위치는 시점 52(13시간)에 표시되고 시점 422(105시간)까지의 추적과 함께 표시됩니다. 그림 7b에서는 시점 280(70시간)의 스냅샷이 표시됩니다. 참고로, 이 세포의 강도는 극도로 약하며 기존 방식으로 감지되지 않았습니다. 그림 7c에는 동일한 스냅샷이 표시되지만 TruAI 확률 이미지가 있습니다. 추적한 세포의 확률 강도는 나머지 세포만큼 높습니다. 그림 7d와 7e는 각각 형광 및 TruAI 확률에 대해 15분마다의 시점에서 추적한 세포의 갤러리를 나타냅니다. 참고로, G1기(적색)를 마칠 때 전체 강도는 항상 매우 약합니다. 그림 7f는 G1, S, G2 및 M 상태의 세포주기 기간을 추출할 수 있는 mCherry(상단) 및 YFP(하단) 형광 강도 변동을 나타냅니다.
이 특정 세포가 시간 경과에 따라 G1 상태에서 더 많은 시간을 보내고 S기와 G2기에서 보내는 시간이 감소하는 것을 이 데이터에서 관찰할 수 있습니다. M 상태는 항상 매우 짧으며, 정확한 위치는 mCherry 신호의 하락과 YFP 신호의 급격한 감쇠처럼 나타납니다.
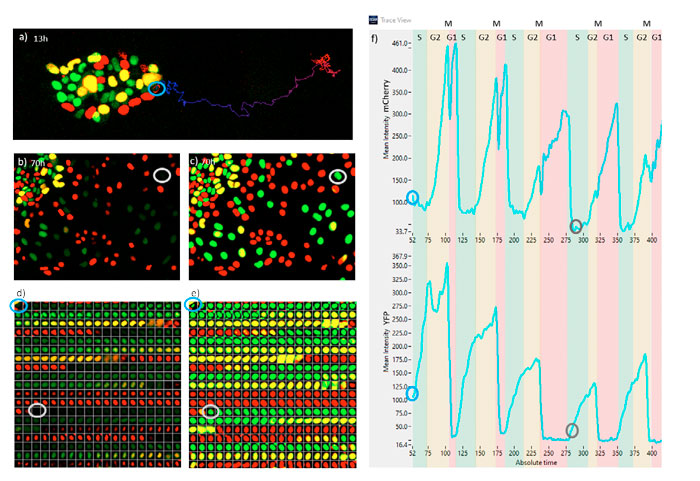
그림 7. a) 13시간 시점의 형광 스냅샷. b) 70시간 시점의 형광 스냅샷. c) 70시간 시점의 TruAI 스냅샷. d) 15분 간격으로 추적한 세포의 형광 시간 경과 시퀀스. e) 15분 간격으로 추적한 세포의 TruAI 시간 경과 시퀀스. f) 추적한 세포에서 다른 세포주기 단계의 기간을 나타내는 mCherry 및 YFP의 형광 흔적. 시간 단계는 15분에 해당합니다. a부터 f까지의 파란색 및 회색 풍선은 각각 13시간 또는 70시간 시점의 동일 세포를 나타냅니다.
결론
scanR High-Content 스크리닝 시스템의 키네틱 모듈과 결합한 배아줄기세포 분화 시의 세포주기 역학을 분석하는 이 새로운 딥러닝 접근 방식을 통해, 분화 시 단일 세포 내 세포주기 역학의 변화에 대한 재현 가능하고 정량적이고 통계적으로 유의미한 데이터를 얻을 수 있습니다. 모양과 형태가 변하는 수천 개의 세포를 분화 기간에 형광 측정을 사용하여 인지하고 추적할 수 있습니다. 이는 초기 발달 및 역분화뿐 아니라 세포주기 조절이 핵심이 되는 세포 변성(악성종양)을 겪고 있는 세포에서 세포주기 역학을 연구할 수 있는 강력한 도구입니다.
참고 문헌
Padgett, J., and Santos, S.D.M. 2020. “From clocks to dominoes: lessons on cell cycle remodeling from embryonic stem cells.” FEBS Letters. 10.1002/1873-3468.13862.
Araujo, A.R., Gelens, L., Sheriff, R.S.M., and Santos, S.D.M. 2016. “Positive feedback keeps duration of mitosis temporally insulated from upstream cell cycle events.” Molecular Cell 64, 362-375.
Sakaue-Sawano et al. 2008. “Visualizing Spatiotemporal Dynamics of Multicellular Cell-Cycle Progression.” Cell 132, 487–498.
Sakaue-Sawano et al. 2017. “Genetically Encoded Tools for Optical Dissection of the Mammalian Cell Cycle.” Molecular Cell 68, 626-640.
Woerdemann, M.,and Genenger, M. 2020. “TruAI™ Technology with Deep Learning for Quantitative Analysis of Fluorescent Cells with Ultra-Low Light Exposure.” Olympus Application Note. www.olympus-lifescience.com/en/resources/white-papers/ultra-low_light_exposure_analysis/
저자
Joe Pagdett 및 Silvia Santos
프랜시스 크릭 연구소의 정량적 줄기세포 생물학 실험실(1 Midland Road, NW1 1AT, London, UK)
Manoel Veiga Gutierrez
Olympus Soft Imaging Solutions GmbH(Johann-Krane-Weg 39, 48149 Muenster, Germany)
이 애플리케이션에 사용되는 제품
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
Not Available in Your Country
Sorry, this page is not
available in your country.