Système Super Resolution pour tous les types d’imagerie de cellules vivantes
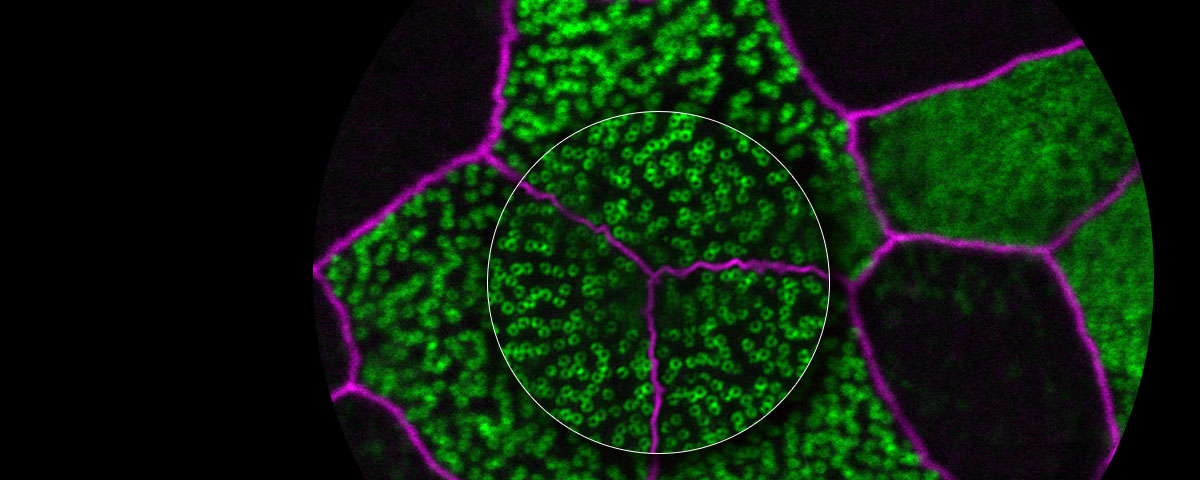
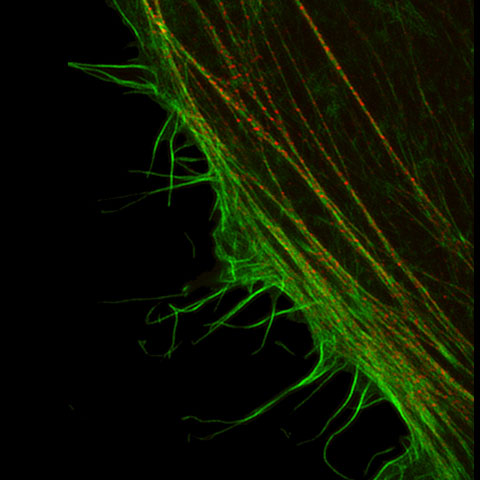
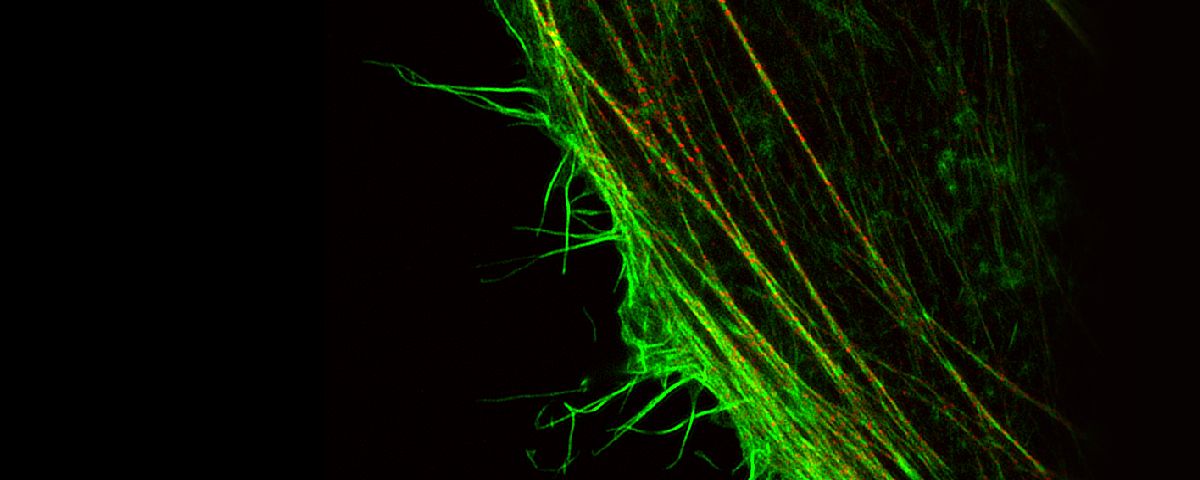
Les microscopes à fluorescence peuvent identifier des protéines spécifiques in vivo à l’aide de sondes fluorescentes. La résolution de plusieurs de ces microscopes est restreinte par une limite de diffraction d’environ 200 nm, qui empêche toute observation de structures fines. Mais grâce à la technologie Super Resolution d’Olympus, vous pouvez acquérir des images nettes d’une résolution jusqu’à 120 nm dans le sens horizontal.
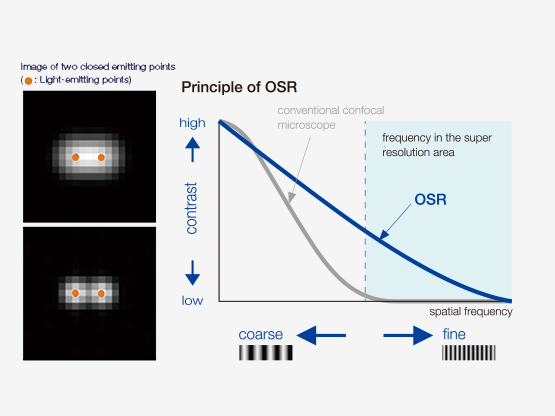
Comment l’OSR fonctionne-t-elle ?
| Les images à haute résolution sont produites grâce à la combinaison d’une détection améliorée, de paramètres matériels spécifiques, d’une optimisation du diamètre d’ouverture confocal et d’un traitement de pointe du signal. La technologie Super Resolution d’Olympus (OSR) permet d’obtenir une résolution latérale (XY) de 120 nm. Référence : |
Olympus Super Resolution (OSR)
IXplore SpinSR10
En savoir plus sur le SpinSR10 FLUOVIEW FV3000 avec FV-OSR
|
Pour en savoir davantage
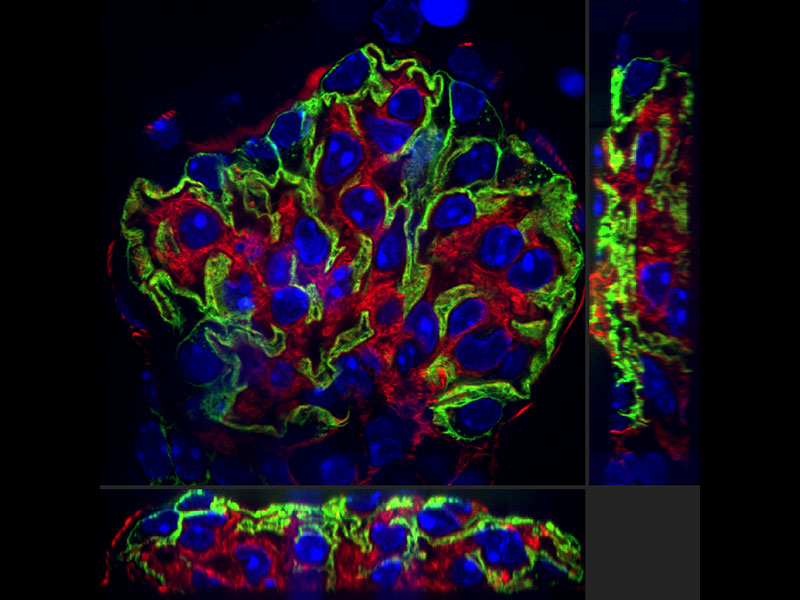
Notre algorithme de déconvolution produit des images de très grande résolution, plus claires et plus nettes. La déconvolution itérative restreinte en 3D supprime le flou le long de l’axe Z pour obtenir une image 3D plus nette.
|
Témoignages
Dr Yasushi Okada, Ph.D. Riken Quantitative Biology Center | La plupart des organelles intracellulaires et des complexes supramoléculaires ont une taille d’environ 100 nm, et la microscopie optique régulière ne permettait pas d’observer la structure de ces complexes. Plusieurs méthodes de microscopie Super Resolution sont en cours de développement. Toutefois, elles ne sont pas faciles à appliquer en biologie, car elles nécessitent des colorations spéciales ou des conditions d’observation ou des systèmes optiques précises. La mise à niveau d’un microscope confocal à disque rotatif vers ce microscope à très haute résolution d’Olympus est simple. L’association avec des objectifs à immersion dans du silicone aide à réduire l’aberration sphérique et permet d’obtenir en temps réel des images à très haute résolution dans un plan profond des cellules. Je pense que cette technologie deviendra un outil utilisé pour la plupart des applications de biologie. |
Sachiko Tsukita, Graduate School of Frontier Biosciences and Graduate School of Medicine, Osaka University | Nous étudions le mécanisme à l’origine de l’alignement et de l’orientation des corps basaux, qui génèrent des cils dans des cellules multiciliées. La disposition régulière des corps basaux est cruciale pour les vagues métachronales des cils. Une résolution maximale de 200 nm est nécessaire pour chacun des axes X, Y et Z pour observer clairement les corps basaux marqués par la centrine GFP lors de la différenciation des cellules multiciliées dans un système de culture trachéal principal. Grâce à un système d’imagerie de cellules vivantes qui incluait le microscope SpinSR10, nous avons pu analyser le mouvement des corps basaux. Par cette méthode, nous avons découvert un nouveau mécanisme dans lequel les corps basaux sont alignés selon l’auto-organisation du cytosquelette apical, dans des cellules multiciliées de la trachée. |
Yuji Ikegaya, Ph.D., Laboratory of Chemical Pharmacology, Graduate School of Pharmaceutical Sciences, The University of Tokyo | Lorsque j’ai vu pour la première fois les images en fluorescence Super Resolution à l’aide du microscope SpinSR10, j’ai été surpris par leur beauté. J’étudie la plasticité des microstructures dans le cerveau, en particulier la synapse postérieure (colonne vertébrale), les fibres des nerfs et les processus des cellules gliales. Grâce aux images acquises avec le microscope SpinSr10, j’ai enfin réalisé que je pouvais « voir ». Grâce à une méthode d’acquisition de la microstructure détaillée autre qu’un microscope électronique, la fiabilité des données récupérées a été améliorée, et l’efficacité de l’expérience a été grandement augmentée grâce à la simplification de la procédure d’acquisition des données. En outre, la mise au point d’images à intervalles de temps réguliers plus longs à partir de la même organisation a conduit à la mise en place de nouvelles méthodes de recherche. |
Références
S. Hayashi and Y. Okada, “Ultrafast superresolution fluorescence imaging with spinning disk confocal microscope optics,” Mol. Biol. Cell 26(9), 1743–1751 (2015).
S. Hayashi, “Resolution doubling using confocal microscopy via analogy with structured illumination microscopy,” Jpn. J. Appl. Phys. 55(8), 082501 (2016).
A. Nagasawa-Masuda and K. Terai, “Yap/Taz transcriptional activity is essential for vascular regression via Ctgf expression and actin polymerization,” PLoS ONE 12(4), e0174633 (2017).
H. Nakajima, et al., “Flow-Dependent Endothelial YAP Regulation Contributes to Vessel Maintenance,” Dev. Cell 40(6), 523-536.e6 (2017).
K. Tateishi, et al., “Three-dimensional Organization of Layered Apical Cytoskeletal Networks Associated with Mouse Airway Tissue Development,” Sci. Rep. 7, 43783 (2017).
E. Herawati, et al., “Multiciliated cell basal bodies align in stereotypical patterns coordinated by the apical cytoskeleton,” J. Cell Biol. 214(5) 571-586 (2016).
M.-T. Ke, et al., “Super-Resolution Mapping of Neuronal Circuitry With an Index-Optimized Clearing Agent,” Cell Rep. 14(11) 2718–2732 (2016).
Sorry, this page is not
available in your country.
Sorry, this page is not
available in your country.
.jpg?rev=2087)